中国海洋湖沼学会主办。
文章信息
- 宋会银, 王一奇, 张梦佳, 陈楠生. 2023.
- SONG Hui-Yin, WANG Yi-Qi, ZHANG Meng-Jia, CHEN Nan-Sheng. 2023.
- 中国新记录定鞭藻物种冠状棕囊藻的遗传进化分析
- GENETIC EVOLUTION OF A NEW RECORD OF PHAEOCYSTIS REX IN CHINA
- 海洋与湖沼, 54(2): 472-482
- Oceanologia et Limnologia Sinica, 54(2): 472-482.
- http://dx.doi.org/10.11693/hyhz20220700180
文章历史
-
收稿日期:2022-07-05
收修改稿日期:2022-09-24
2. 江汉大学生命科学学院 湖北武汉 430056;
3. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室 山东青岛 266237;
4. 中国科学院海洋大科学研究中心 山东青岛 266071;
5. 中国科学院大学 北京 100049
2. School of Life Sciences, Jianghan University, Wuhan 430056;
3. Functional Laboratory for Marine Ecology and Environmental science, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China;
4. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China;
5. University of Chinese Academy of Sciences, Beijing 100049, China
棕囊藻属(Phaeocystis)于1893年首次描述, 包含10个物种, 其中3个物种波切棕囊藻(Phaeocystis pouchetii)、球形棕囊藻(Phaeocystis globosa)和南极棕囊藻(Phaeocystis antarctica)均具有复杂异型生活史, 兼具单细胞和由多细胞形成的囊体这两种形态, 常可形成大规模有害藻华(Schoemann et al, 2005)。其中, 球形棕囊藻赤潮自1997年在我国南海海域首次暴发以来, 陆续在我国的多个海域暴发, 造成了严重的生态危害(Wang et al, 2021)。
棕囊藻属物种的运动细胞均具两条长鞭毛及一条短的定鞭毛, 色素体1~4个。模式种波切棕囊藻的典型形态特征为囊体呈裂瓣状或云雾状(尽管新形成的囊体可能为球形), 囊体直径 < 2 mm, 囊体内往往有四个细胞或更多的细胞聚堆, 囊体的胶被较球形棕囊藻更为脆弱(Jacobsen, 2002)。南极棕囊藻的囊体一般也较小(直径 < 2 mm), 且更容易黏附于基质表面, 两根鞭毛的长度不相等(Medlin et al, 2007)。球形棕囊藻可以形成直径较大的球形囊体(直径 < 3 cm), 且囊体内细胞往往均匀分布(Medlin et al, 2007)。棕囊藻的其他物种为单细胞或可形成不定形群体, 心形棕囊藻(Phaeocystis cordata)细胞相对较小(直径3~4 μm), 具有2个叶绿体, 且未观察到非运动细胞, 均为运动细胞(Zingone et al, 1999)。扬棕囊藻(Phaeocystis jahnii)细胞直径3.5~5 μm, 具有2~4个叶绿体, 可形成不定形群体(Zingone et al, 1999)。蜂窝状棕囊藻(Phaeocystis scrobiculata)细胞为球形, 仅观察到运动细胞状态, 直径8 μm, 分泌物质由9根丝状物组成一组, 中心不规则(Medlin et al, 2007)。
冠状棕囊藻(Phaeocystis rex)是Andersen等(2015)首次从阿拉伯海分离到的单细胞棕囊藻物种, 迄今该种仅在阿拉伯海域有记录。该种为运动或不运动单细胞形态, 细胞较大, (6~10) μm×(8~15) μm, 运动细胞具有2根鞭毛和1根定鞭毛, 细胞外围覆盖有多层有机鳞片, 常见细胞一端增厚, 具2个色素体, 呈前伸手指状突起。系统进化分析表明冠状棕囊藻位于群体型种类分支的基部。
尽管基于野外样品的分析结果表明我国海域可能存在南极棕囊藻(P. antarctica)(Wu et al, 2015)、心形棕囊藻(P. cortada)和扬棕囊藻(P. jahnii)(Lin et al, 2014)等至少3个棕囊藻物种, 但这些均以“未培养克隆(uncultured clone)”的形式出现, 尚未分离到可以用于分类学鉴定的株系。迄今在我国海域仅明确鉴定到球形棕囊藻这一棕囊藻物种(陈菊芳等, 1999; 曲凌云等, 2008; 沈萍萍等, 2018, 2021)。我们在分析2021年的一次南海航次水样时, 观察到一种形态特征疑似棕囊藻的定鞭藻类。通过单细胞分离、培养, 获取了单克隆株系, 经形态特征及DNA分子标记的序列分析, 鉴定其为冠状棕囊藻, 这是该种在我国的首次记录。此外, 我们进一步构建了冠状棕囊藻的叶绿体基因组和线粒体基因组, 比较基因组分析表明, 与球形棕囊藻和南极棕囊藻相比, 冠状棕囊藻的细胞器基因组发生了显著的结构重排和序列变异。冠状棕囊藻在我国海域的发现及细胞器基因组的构建, 将为棕囊藻的地理分布和遗传进化研究提供基础参考资料。
1 材料与方法 1.1 样品采集、藻株分离和培养由“中国科学院任务/战略性先导科技专项(B类)印太交汇区海洋物质能量中心形成演化过程与机制”资助, 搭载“南海季风综合调查航次”的科考船“嘉庚”号, 进行样品采集。样品采集自我国南海海域, 坐标为118°E、18°N (图 1), 采集时间为2021年5月, 用采水器直接采集水体样本。
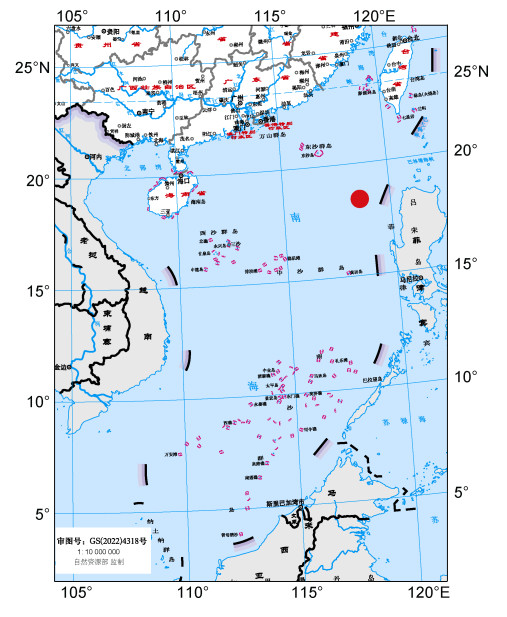 |
| 图 1 冠状棕囊藻株系CNS01077的地理位置信息 Fig. 1 Location of the sampling site (red dot) for the P. rex strain CNS01077 注: 红色圆圈标注采样点 |
藻株分离采用毛细管分离法: 用自制的毛细玻璃管于Nikon TS2倒置显微镜(日本东京)下挑取单细胞, 经无菌海水清洗后, 转移到装有L1培养基的24孔培养板中, 培养条件: 温度(19±1) ℃, 光照强度为30 μmol/(m2∙s), 光照周期为12h︰12h光照黑暗循环。
1.2 形态特征观察取培养状态良好的细胞, 采用ZEISS Axio Imager Z2 (Zeiss, Sliedrecht, 荷兰)电动正置显微镜进行显微形态观察及照片拍摄。
1.3 DNA提取、基因组测序取处于对数期的藻细胞, 离心收集藻体, 用细胞破碎仪(MP Biomedicals, Santa Ana, CA, 美国)以4 m/s的速度破碎细胞5 s, 利用试剂盒HP Plant DNA Kit (Omega, Norcross, GA, 美国)进行DNA提取, 完成后, 使用NanoDrop 2000 (Thermo Scientific, 美国)分光光度计测定DNA的浓度和质量, 使用1%琼脂糖凝胶检查DNA的完整性。确保合格后使用NEB Next® Ultra™DNA Library Prep Kit (NEB, 美国)进行DNA文库的制备: 首先使用超声仪(Covaris S220, Covaris, 美国)将基因组DNA破碎成350 bp, 之后进行末端修复, 并添加A尾和测序接头, 接着进行纯化和PCR扩增。使用安捷伦2100生物分析仪(Agilent, 美国)对文库进行检测和质控。使用NovaSeq PE150 (Illumina, San Diego, CA, 美国)平台对DNA文库进行测序, 生成长度为150 bp的reads, 测序工作由北京诺禾致源公司完成。
1.4 18S rDNA和细胞器基因组序列的筛选组装以近缘物种的目标片段序列为参考, 使用GetOrganelle (Jin et al, 2020)软件进行筛选组装, 得到18S rDNA和细胞器基因组序列, 针对有疑问的区域, 在两端设计引物, 采用PCR扩增和一代测序的方法进行验证。之后, 以组装出的结果为参考, 使用BWA (Li et al, 2009a)、SAMtools (Li et al, 2009b)和IGV (Thorvaldsdottir et al, 2013)进行检验。
1.5 细胞器基因组的注释及比较分析使用MFannot (https://github.com/BFL-lab/Mfannot) 在线网站进行细胞器基因组的基因注释, 使用sequin软件和Blast同源检索对结果进行完善。注释好的细胞器基因组通过在线软件OGDRAW (https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)进行图谱绘制。使用Mauve (Darling et al, 2010)软件进行细胞器基因组间的共线性分析。
1.6 系统发育分析从NCBI下载近缘物种的细胞器基因组序列。(1)18S rDNA序列矩阵经MEGA 7.0比对并手动剪切后, 使用IQ-TREE (Trifinopoulos et al, 2016)进行模型预测并构建系统发育树; (2)细胞器基因组序列: 使用PhyloSuite (Zhang et al, 2020)软件分别从叶绿体基因组和线粒体基因组提取共有基因的蛋白序列, 序列经比对、剪切和串联, 生成最终数据集。包括atp6, atp9, cox1, cox2, cox3, cob, nad1, nad2, nad3, nad4L, nad4, nad5和nad6等共计13个基因的蛋白质序列用于线粒体基因组系统发育分析。包括rpoC1, ccs1, rps2, rpoC2, rpl2, ycf3, rps3, rpl3, ycf4, rps4, rps5, rpl5, rps6, rpl6, rps7, rps8, rps9, rps10, rps11, rps12, rps13, rps14, rpl14, rps16, rpl16, rps17, rps18, ycf19, rps19, rpl19, rpl20, rpl21, rpl22, rpl23, rpl27, rpl31, rpl33, rpl34, rpl36, atpA, atpB, atpD, atpE, atpF, atpG, atpH, atpI, ccsA, clpC, dnaK, groEL, minD, petA, petB, petD, petG, petL, petM, petN, psaA, psaB, psaC, psaD, psaF, psaI, psaJ, psaL, psaM, psbA, psbB, psbC, psbD, psbE, psbF, psbH, psbI, psbJ, psbK, psbL, psbN, psbT, psbV, psbZ, rbcL, rbcS, rpoA, rpoB, secA, secY, tatC, tufA等91个基因的蛋白序列用于叶绿体基因组系统发育分析。使用IQ-TREE (Trifinopoulos et al, 2016)对每个基因进行模型预测、系统发育分析。
1.7 核苷酸多样性(nucleotide diversity, Pi)的计算利用PhyloSuite (Zhang et al, 2020)软件提取冠状棕囊藻CNS01077株系、球形棕囊藻CNS00066株系和南极棕囊藻CCMP1374株系的蛋白编码基因, 经MAFFT比对、trimAL (automated1)剪切, 利用Launch DnaSP6软件进行核苷酸多样性(nucleotide diversity, Pi)的计算。
2 结果 2.1 物种鉴定 2.1.1 CNS01077株系形态特征描述单细胞, 细胞运动或不运动, 运动细胞可明显观察到2根鞭毛和1根定鞭毛(图 2a, b), 营养细胞卵形、心形、椭圆形或球形(图 2a~2g), (4~10) μm×(5~16) μm, 非运动细胞常见一端增厚(图 2d~2h), 有时以增厚点为附着点附着生长, 叶绿体2个, 侧生, 每个叶绿体呈指状突起, 形成深杯状外观, 每个叶绿体具一个蛋白核。观察到包含2个子细胞的孢子囊(图 2h), 以孢子的形式进行繁殖。
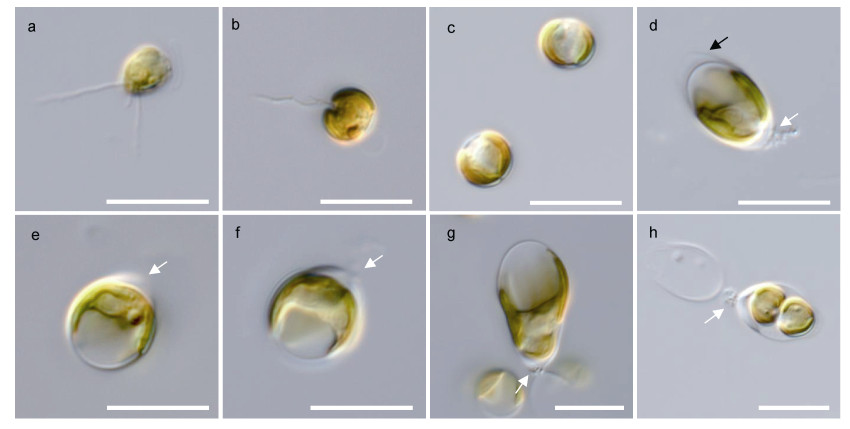 |
| 图 2 CNS01077株系的显微形态特征 Fig. 2 Morphological features of P. rex 注: a, b: 运动细胞, 可明显观察到2根鞭毛和1根定鞭毛; c~g: 非运动细胞; c: 心形细胞; d: 椭圆形细胞; e, f: 球形细胞; g: 不规则长椭圆形细胞; h: 孢子囊; 白色箭头: 细胞壁的增厚点; 黑色箭头: 指示类似细胞壁的覆盖物; 标尺: 10 μm |
通过筛选组装得到了CNS01077株系的18S rDNA全长序列(Genbank编号为ON860696), 基于Blastn同源比对初步确定其为棕囊藻属成员。从NCBI下载棕囊藻属成员的18S rDNA序列, 以进行系统发育分析(图 3), 结果表明, CNS01077株系的全长18S rDNA与冠状棕囊藻模式株系(CCMP 2000)的全长18S rDNA (KF422607)聚为一支, 二者的序列相似性为100%, 序列完全相同。冠状棕囊藻位于能够形成囊体的球形棕囊藻、波切棕囊藻和南极棕囊藻共同形成的支系的基部, 这与Andersen等(2015)的报道相符。
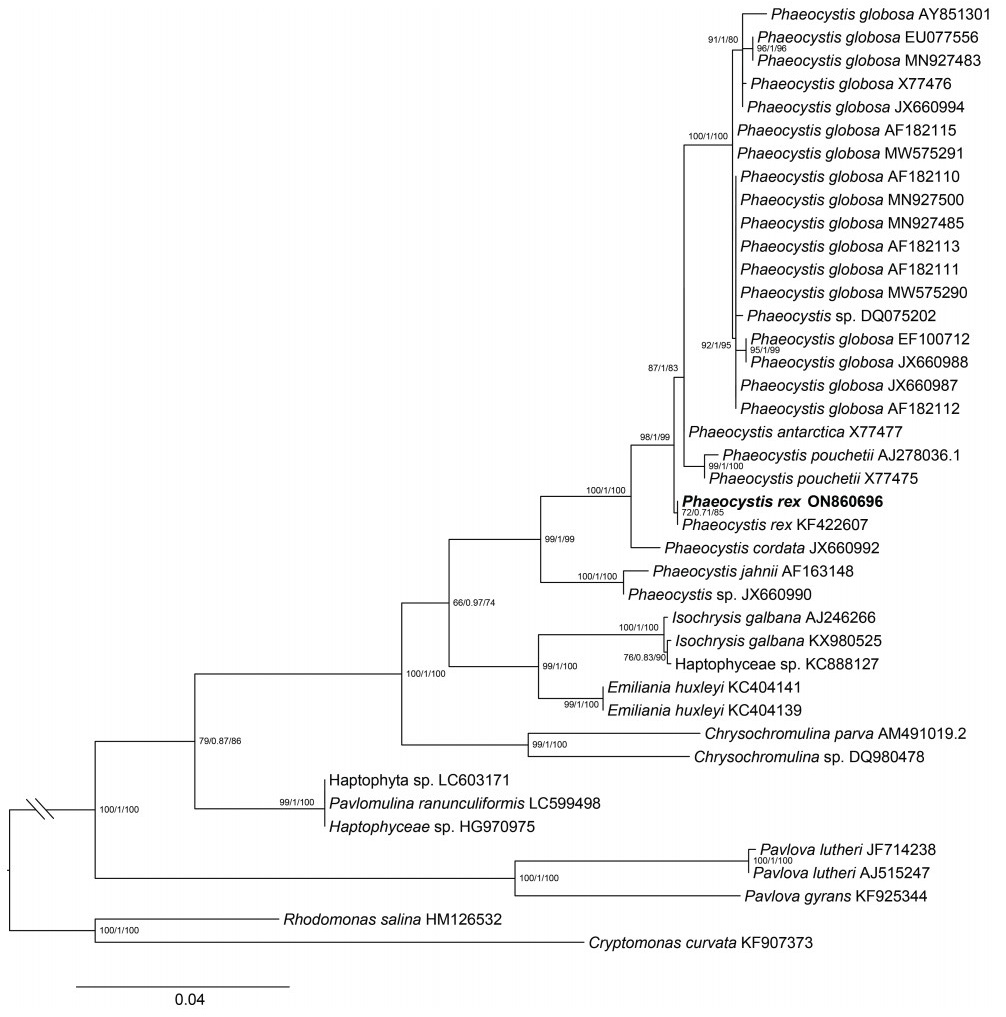 |
| 图 3 基于定鞭藻门18S rDNA序列的系统发育分析 Fig. 3 Phylogenetic analysis of the haptophyte species based on 18S rDNA sequences 注: 共用41条18S rDNA序列, 数据矩阵包含1640位点。利用W-IQ-TREE计算核苷酸最佳替代模型(TNe+I+G4), 并进行系统发育分析。支系上的数值分别代表SH-aLRT support (%) / aBayes support / ultrafast bootstrap support (%)。本研究获取的株系加粗显示 |
基于形态特征和系统发育位置, CNS01077株系鉴定为冠状棕囊藻。
2.2 冠状棕囊藻的遗传进化分析为进一步解析冠状棕囊藻特点及与其他棕囊藻物种之间的遗传进化关系, 本研究对其细胞器基因组进行了组装、注释和比较分析。
2.2.1 叶绿体基因组特征CNS01077株系的叶绿体基因组为110 250 bp, 包括108个蛋白编码基因、3个rRNA基因(rnl、rns和rrn5, 各2个拷贝)和27个tRNA基因(图 4)。相比球形棕囊藻和南极棕囊藻, CNS01077株系的叶绿体基因组未发现特有基因, 所有基因均在球形棕囊藻和南极棕囊藻叶绿体基因组中出现。但在基因的排列组成上, 三个物种的叶绿体基因组存在多处重排(图 5a)。为进一步探究叶绿体基因组在物种间的变异, 本研究计算了冠状棕囊藻、球形棕囊藻和南极棕囊藻的108个蛋白编码基因的Pi值, 其变化范围是0.019 (atpH)~0.26 (ycf80), 平均值为0.086, 在这108个基因中, atpH基因最为保守, ycf80基因的变异最高(图 5b)。基于叶绿体基因组的91个蛋白编码基因的蛋白序列的系统发育分析证明了CNS01077株系位于南极棕囊藻和球形棕囊藻形成的支系的基部(图 6)。
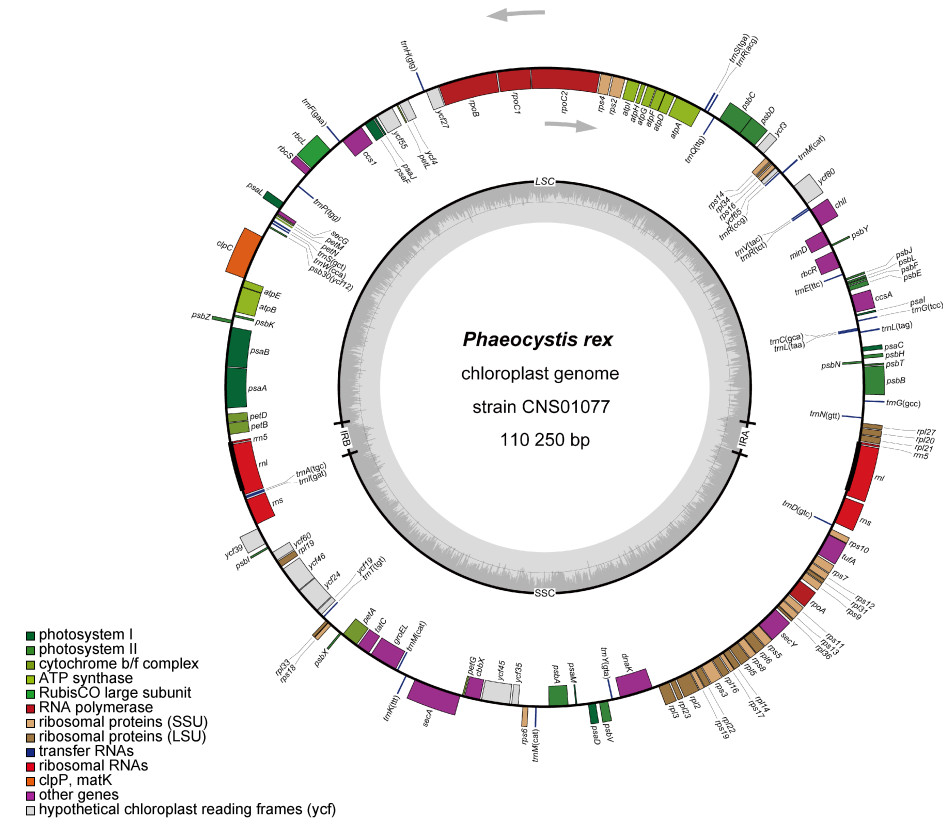 |
| 图 4 冠状棕囊藻CNS01077株系的叶绿体基因组图谱 Fig. 4 Circular map of the chloroplast genome of P. rex strain CNS01077 注: 基因的颜色根据基因的功能分类(注释见图谱下面的索引)。GC含量显示在内圈上。基因组包含两个反向重复序列(IRa和IRb)、一个大单拷贝区(LSC)和一个小单拷贝区(SSC) |
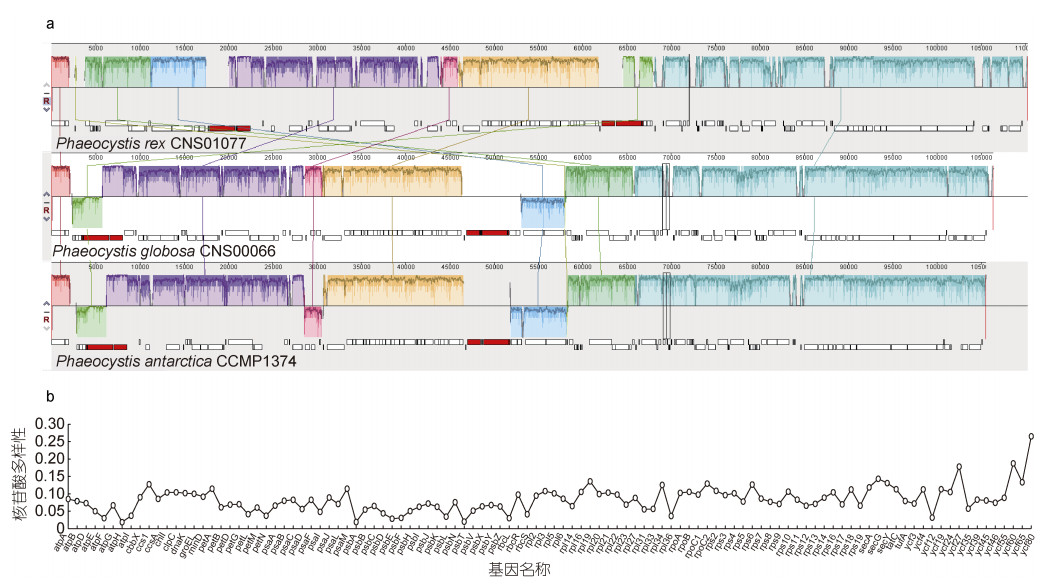 |
| 图 5 冠状棕囊藻、球形棕囊藻和南极棕囊藻叶绿体基因组的共线性分析(a)及其共有蛋白的核苷酸多样性分析(b) Fig. 5 Collinearity analysis (a) and nucleotide diversity (Pi) (b) analysis of the chloroplast genomes of Phaeocystis rex, P. globosa and P. antarctica 注: 每个彩色模块表示基因组间的共线性区域 |
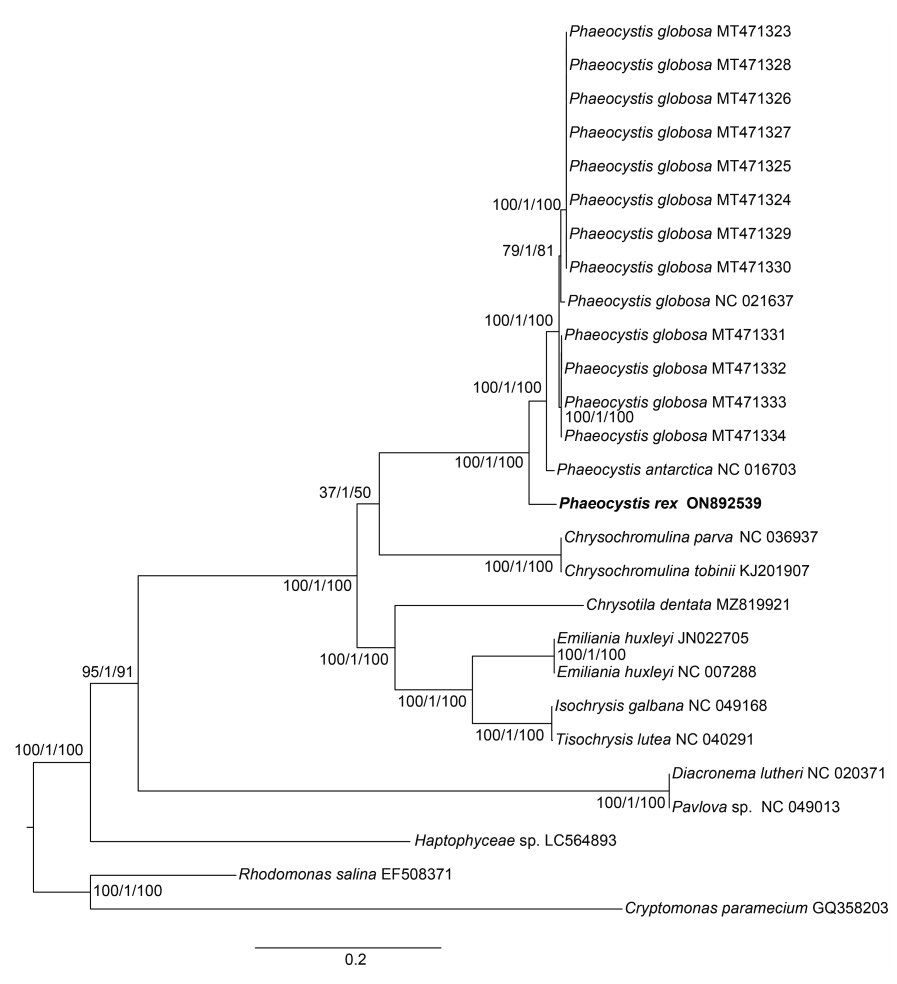 |
| 图 6 基于定鞭藻门叶绿体基因组91个蛋白序列的系统发育分析 Fig. 6 Phylogenetic tree of haptophyte species based on 91 chloroplast proteins 注: 利用W-IQ-TREE计算每个分区的核苷酸最佳替代模型, 并进行系统发育分析。支系上的数值分别代表SH-aLRT支持值(%) / aBayes支持值/ ultrafast支持值(%)。本研究获取的株系加粗显示 |
本研究得到了CNS01077株系的部分线粒体基因组序列(线粒体基因组具有一个长度未知的重复区), 其编码19个蛋白编码基因、2个rRNA基因(rnl和rns)和25个tRNA基因(图 7)。球形棕囊藻、南极棕囊藻和冠状棕囊藻三个物种拥有相同数量的蛋白编码基因和rRNA基因。相比南极棕囊藻, 冠状棕囊藻和球形棕囊藻缺失一个tRNA基因trnG (TCC)。在基因的排列组成上, 三个物种的线粒体基因组之间存在多处重排(图 8a)。为进一步探究线粒体基因组在物种间的变异性, 本研究计算了冠状棕囊藻、球形棕囊藻和南极棕囊藻的19个蛋白编码基因的Pi值, 其变化范围是0.11 (atp9)~0.35 (atp8), 平均值为0.23, 在这19个基因中, atp9基因最为保守, atp8基因的变异最高(图 8b)。基于线粒体基因组19个蛋白序列的系统发育分析再次证明了CNS01077株系位于南极棕囊藻和球形棕囊藻形成的支系的基部(图 9)。
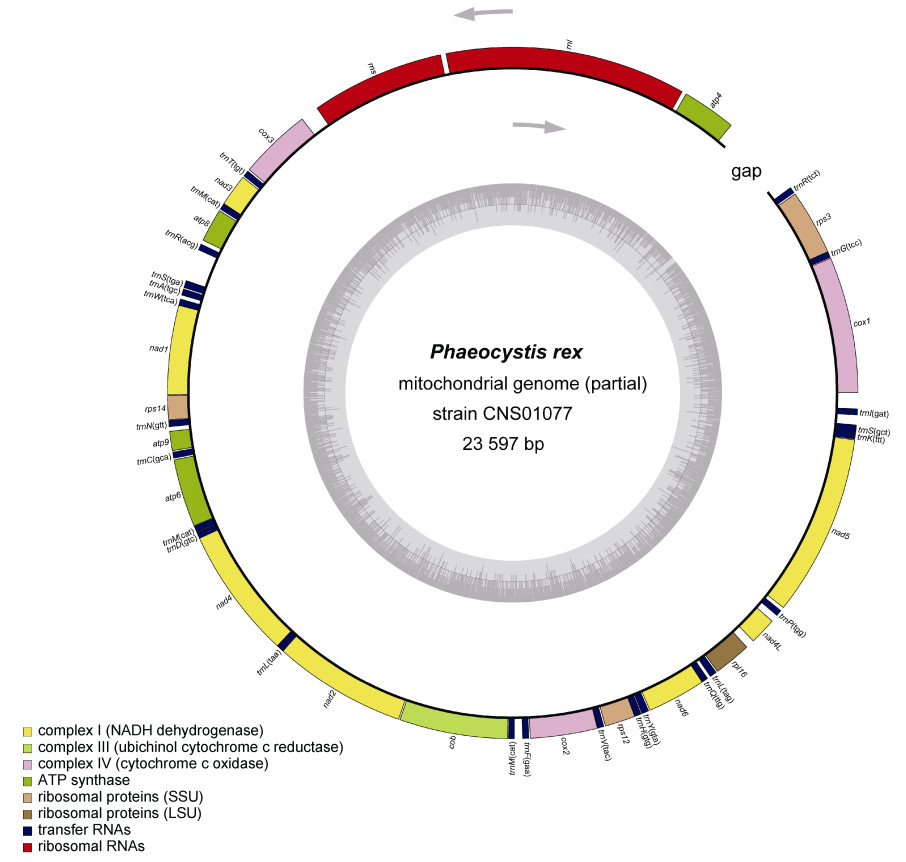 |
| 图 7 冠状棕囊藻CNS01077株系的线粒体基因组图谱 Fig. 7 The circular map of the mitochondrial genome of P. rex strain CNS01077 注: 基因的颜色根据基因的功能分类(图谱下面的索引), GC含量显示在内圈上 |
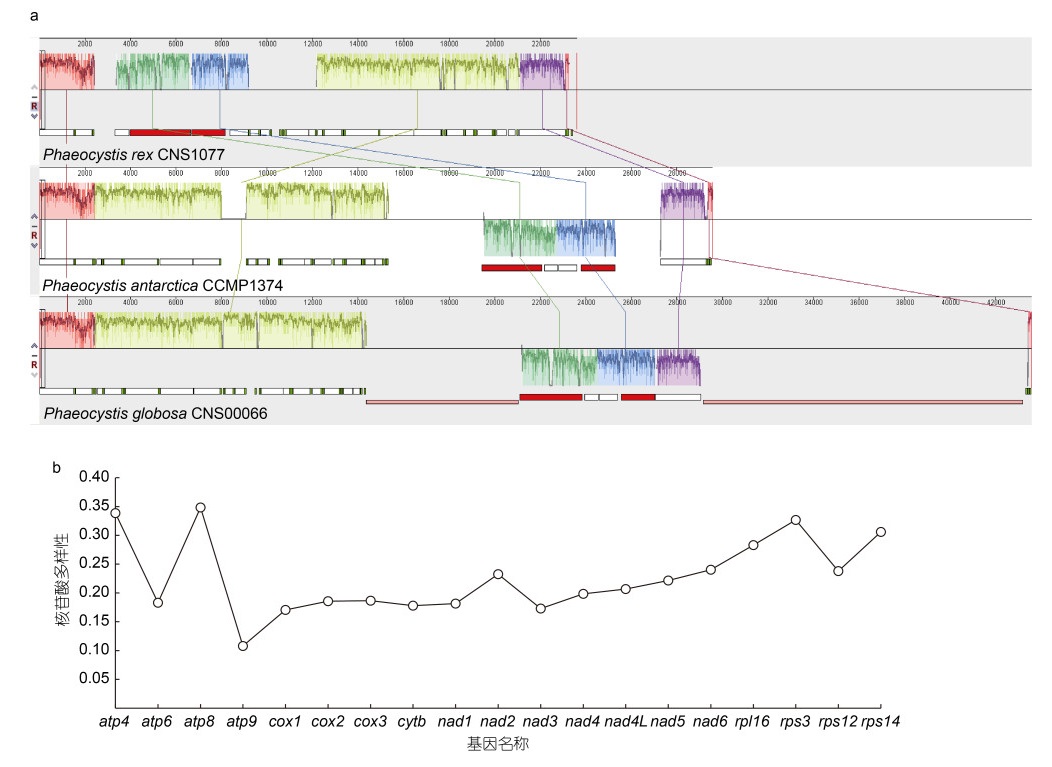 |
| 图 8 冠状棕囊藻、球形棕囊藻和南极棕囊藻线粒体基因组的共线性分析(a)及其共有蛋白的核苷酸多样性分析(b) Fig. 8 Collinearity analysis (a) and nucleotide diversity (Pi) analysis (b) among the mitochondrial genome of P. rex, P. globosa and P. antarctica 注: 每个彩色模块表示基因组间的共线性区域值 |
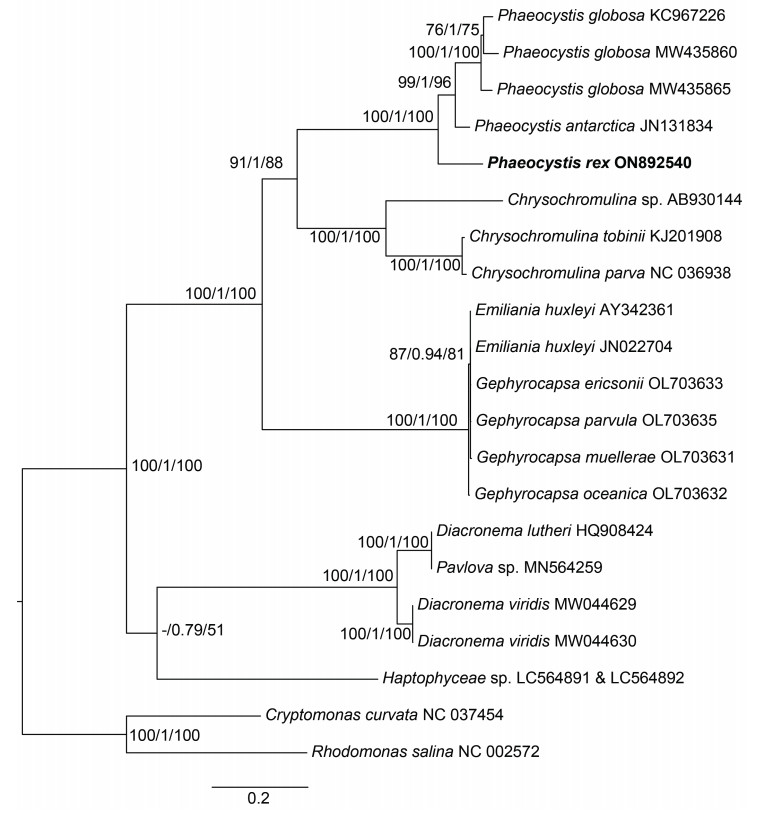 |
| 图 9 基于定鞭藻门线粒体基因组13个蛋白序列的系统发育分析 Fig. 9 Phylogenetic analysis of haptophyte species based on 13 mitochondrial proteins 注: 利用W-IQ-TREE计算每个分区的核苷酸最佳替代模型, 并进行系统发育分析。支系上的数值分别代表SH-aLRT支持值(%) / aBayes支持值/ ultrafast支持值(%)。本研究获取的株系加粗显示 |
CNS01077株系的运动细胞具有两根鞭毛和一根定鞭毛、非运动细胞外层偶见残留的细胞壁样覆盖物、常见细胞壁一端增厚、细胞在疑似细胞壁的覆盖物内进行细胞分裂, 随后子细胞释放、色素体为两个, 每个色素体指状突起, 形成深杯状外观, 这些特征符合冠状棕囊藻的典型形态特征。冠状棕囊藻的模式株CCMP2000的细胞直径为(6~10)×(8~15) μm, 而CNS01077株系的细胞大小为(4~10) μm×(5~16) μm, 略比模式株小, 表明冠状棕囊藻细胞大小变化范围更大, 基于此, 我们对该种的形态特征进行补充, 冠状棕囊藻的细胞大小为: (4~10) μm×(5~16) μm。
3.2 我国海域棕囊藻物种的多样性截止本研究开始前, 我国海域仅明确鉴定了一个棕囊藻物种, 即赤潮物种——球形棕囊藻。本研究首次分离、培养、分析了我国海域的新记录种——冠状棕囊藻。前人基于环境样品的分析表明我国海域极有可能存在其他棕囊藻种类, 如Lin等(2014)基于18S rRNA特异探针对环境样品开展研究时, 发现我国黄海的一次定鞭藻藻华主要由扬棕囊藻和心形棕囊藻引起。Wu等(2015)基于环境样品在我国南海发现的棕囊藻18S rDNA序列分别和南极棕囊藻、一个未定种Phaeocystis sp.聚为一支。这些研究表明我国海域棕囊藻多样性较高, 推测存在至少5种棕囊藻物种, 除了已经分离鉴定的球形棕囊藻和冠状棕囊藻(本研究), 极有可能还有尚待分离鉴定的南极棕囊藻、扬棕囊藻和心形棕囊藻。进一步对棕囊藻藻株分离培养, 并结合形态特征和系统发育研究等方法对其进行准确的物种鉴定将揭示更高的棕囊藻物种多样性。
3.3 冠状棕囊藻在棕囊藻进化上的重要意义基于18S rDNA数据、叶绿体基因组数据和线粒体基因组数据的系统发育研究表明, 冠状棕囊藻位于能够形成囊体的三个物种(球形棕囊藻、南极棕囊藻和波切棕囊藻)形成的支系的基部, 是已知与囊体形成物种亲缘关系最为密切的单细胞类群。冠状棕囊藻为单细胞形态, 常见其细胞一端的增厚是其典型的形态特征, 我们观察到该增厚点往往是其附着于其他基质或其他细胞的附着点, Andersen等(2015)也曾观察到少数几个细胞相互附着, 这或许为棕囊藻属内成员由单细胞进化为囊体的过渡形态。但位于冠状棕囊藻基部的扬棕囊藻, 偶见也可形成不定形的囊体形态(Zingone et al, 1999), 这说明棕囊藻的形态进化可能很复杂。对冠状棕囊藻乃至更多棕囊藻物种的进一步深入解析, 将对解析棕囊藻的系统进化和囊体出现的机制有重要意义。
3.4 棕囊藻属内细胞器基因组比较分析截止至本研究开始前, 仅有2个棕囊藻属物种的细胞器基因组数据被报道(Smith et al, 2014; Song et al, 2021a, 2021b), 即球形棕囊藻和南极棕囊藻。本研究首次构建了冠状棕囊藻的细胞器基因组。虽然这三个物种的蛋白编码基因均为共有基因, 但基因序列存在明显差异, 且在基因组结构上存在重排, 这表明三个物种存在明显的进化差异。核苷酸多样性分析表明, 冠状棕囊藻的线粒体基因组变异性高于叶绿体基因组, 而在球形棕囊藻和南极棕囊藻内, 也被证明线粒体基因组的变异率远高于叶绿体基因组(Smith et al, 2014), 这极有可能是棕囊藻属或更高分类阶元的共有现象, 背后的成因还有待进一步研究。
4 结论尽管我国海域棕囊藻物种的多样性较高, 但是迄今为止只有球形棕囊藻一个物种得到分离鉴定。基于形态特征和分子标记序列分析, 本研究对分离自中国南海的一个单细胞鞭毛类株系CNS01077株系进行物种鉴定, 确认其为冠状棕囊藻, 这是该物种在我国海域的首次报道。此外, 本研究构建了冠状棕囊藻首个叶绿体基因组和线粒体基因组, 比较基因组分析表明: 相比南极棕囊藻和球形棕囊藻, 冠状棕囊藻的细胞器基因组存在结构重排和序列变异。本研究将为棕囊藻的在我国海域的地理分布研究、遗传进化研究提供基础参考资料。
致谢 感谢中国科学院海洋研究所海洋大数据中心的支持。非常感谢两位匿名审稿人提出的极具建设性的意见和建议。
曲凌云, 吕颂辉, 高春蕾, 等, 2008. 棕囊藻渤海株核糖体18S rDNA和ITS基因结构序列分析. 海洋科学进展, 26(2): 200-206 DOI:10.3969/j.issn.1671-6647.2008.02.010 |
沈萍萍, 齐雨藻, 2021. 棕囊藻属(Phaeocystis)的种类多样性及地理分布特征研究进展. 海洋与湖沼, 52(1): 1-15 |
沈萍萍, 齐雨藻, 欧林坚, 2018. 中国沿海球形棕囊藻(Phaeocystis globosa)的分类、分布及其藻华. 海洋科学, 42(10): 146-162 DOI:10.11759/hykx20171225004 |
陈菊芳, 徐宁, 江天久, 等, 1999. 中国赤潮新记录种——球形棕囊藻(Phaeocystis globosa). 暨南大学学报(自然科学与医学版), 20(3): 124-129 |
ANDERSEN R A, BAILEY J C, DECELLE J, et al, 2015. Phaeocystis rex sp. nov. (Phaeocystales, Prymnesiophyceae): a new solitary species that produces a multilayered scale cell covering. European Journal of Phycology, 50(2): 207-222 DOI:10.1080/09670262.2015.1024287 |
DARLING A E, MAU B, PERNA N T, 2010. progressiveMauve: multiple genome alignment with gene gain, loss and rearrangement. PLoS One, 5(6): e11147 DOI:10.1371/journal.pone.0011147 |
JACOBSEN A, 2002. Morphology, relative DNA content and hypothetical life cycle of Phaeocystis pouchetii (Prymnesiophyceae); with special emphasis on the flagellated cell type. Sarsia, 87(5): 338-349 DOI:10.1080/0036482021000155795 |
JIN J J, YU W B, YANG J B, et al, 2020. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes. Genome Biology, 21(1): 241 DOI:10.1186/s13059-020-02154-5 |
LI H, DURBIN R, 2009a. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics, 25(14): 1754-1760 DOI:10.1093/bioinformatics/btp324 |
LI H, HANDSAKER B, WYSOKER A, et al, 2009b. The sequence alignment/map format and SAMtools. Bioinformatics, 25(16): 2078-2079 DOI:10.1093/bioinformatics/btp352 |
LIN Y C, CHUNG C C, GONG G C, et al, 2014. Diversity and abundance of haptophytes in the East China Sea. Aquatic Microbial Ecology, 72(3): 227-240 DOI:10.3354/ame01697 |
MEDLIN L, ZINGONE A, 2007. A taxonomic review of the genus Phaeocystis. Biogeochemistry, 83(1): 3-18 |
SCHOEMANN V, BECQUEVORT S, STEFELS J, et al, 2005. Phaeocystis blooms in the global ocean and their controlling mechanisms: a review. Journal of Sea Research, 53(1/2): 43-66 |
SMITH D R, ARRIGO K R, ALDERKAMP A C, et al, 2014. Massive difference in synonymous substitution rates among mitochondrial, plastid, and nuclear genes of Phaeocystis algae. Molecular Phylogenetics and Evolution, 71: 36-40 |
SONG H Y, CHEN Y, GIBSON K, et al, 2021a. High genetic diversity of the harmful algal bloom species Phaeocystis globosa revealed using the molecular marker COX1. Harmful Algae, 107: 102065 |
SONG H Y, CHEN Y, LIU F, et al, 2021b. Large differences in the haptophyte Phaeocystis globosa mitochondrial genomes driven by repeat amplifications. Frontiers in Microbiology, 12: 676447 |
THORVALDSDOTTIR H, ROBINSON J T, MESIROV J P, 2013. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in Bioinformatics, 14(2): 178-192 |
TRIFINOPOULOS J, NGUYEN L T, VON HAESELER A, et al, 2016. W-IQ-TREE: a fast online phylogenetic tool for maximum likelihood analysis. Nucleic Acids Research, 44(W1): W232-W235 |
WANG X D, SONG H Y, WANG Y, et al, 2021. Research on the biology and ecology of the harmful algal bloom species Phaeocystis globosa in China: progresses in the last 20 years. Harmful Algae, 107: 102057 |
WU W X, WANG L, LIAO Y, et al, 2015. Microbial eukaryotic diversity and distribution in a river plume and cyclonic eddy-influenced ecosystem in the South China Sea. Microbiologyopen, 4(5): 826-840 |
ZHANG D, GAO F, JAKOVLIĆ I, et al, 2020. PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies. Molecular ecology resources, 20(1): 348-355 |
ZINGONE A, CHRÉTIENNOT-DINET M J, LANGE M, et al, 1999. Morphological and genetic characterization of Phaeocystis cordata and P. jahnii (Prymnesiophyceae), two new species from the Mediterranean Sea. Journal of Phycology, 35(6): 1322-1337 |
 2023, Vol. 54
2023, Vol. 54


