
中国海洋湖沼学会主办。
文章信息
- 陈家鑫, 刘佳文, 法文龙, 李浩天, 类彦立. 2023.
- CHEN Jia-Xin, LIU Jia-Wen, FA Wen-Long, LI Hao-Tian, LEI Yan-Li. 2023.
- 黄海潮间带和浅海表层沉积物中植物DNA提取和PCR扩增的方法学探索
- METHODOLOGICAL EXPLORATION OF PLANT DNA EXTRACTION AND PCR AMPLIFICATION FROM INTERTIDAL AND SHALLOW SEA SEDIMENTS OF THE YELLOW SEA
- 海洋与湖沼, 54(3): 718-731
- Oceanologia et Limnologia Sinica, 54(3): 718-731.
- http://dx.doi.org/10.11693/hyhz20220900229
文章历史
-
收稿日期:2022-09-06
收修改稿日期:2022-10-13
2. 南方海洋科学与工程广东省实验室(珠海) 广东珠海 519082;
3. 中国科学院大学 北京 100049
2. Southern Marine Science and Engineering Guangdong Laboratory (Zhuhai), Zhuhai 519082, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
在古海洋学研究中, 海洋沉积物中记录的信息是反演海洋和陆地气候与环境变化的重要载体(Corinaldesi et al, 2008), 海洋环境的变化一般通过有孔虫进行指示(Chen et al, 2008; Muhong et al, 2008; Wang et al, 2018, 2019a, 2019b; Jian et al, 2019, 2020; Li et al, 2021a2, 3; Zhang et al, 2021; Zhou et al, 2021; Chen et al, 2022; Xiong et al, 2022), 陆地环境的变化则一般通过沉积物中的植物孢粉信息指示(Voldstad et al, 2020; Yang et al, 2020b2, 20213; Li et al, 2021b2)。植物是自然界中物质循环与能量交换的重要环节(张佳华等, 2000), 它们提供了许多宝贵的生态系统服务(Costanza et al, 2014)。植物群落的动态与气候变化有明显的相关性(Farrell et al, 2015), 这些植被由于气候变化的影响, 在全球范围内正处于不断下降的趋势(Kern et al, 1992; Erb et al, 2018; Cuni-Sanchez et al, 2021)。虽然植物群落对气候环境变化具有良好的指示作用, 但是到目前为止, 对于检测植被变化、评价人类活动影响的有效且高效的方法十分匮乏。
植被分布情况的获得一般依赖于野外调查(Chytrý et al, 2011)、分析沉积物中的孢粉(Yang et al, 2020b)3或遥感数据(Qi et al, 2000; Wang et al, 2002)。研究者可以通过遥感数据和野外调查来评估从米到千米的空间变化(Taubert et al, 2018), 对沉积物中的孢粉进行分析可以重建古植被的分布与变化(Yang et al, 2021)6。然而, 野外调查与遥感观测需要随着时间的推移进行多次调查和观测, 以生成用于变化检测的时间序列记录(Ahl et al, 2006; Adole et al, 2016; Zeng et al, 2020), 这种持续观察的成本高昂并且非常耗时(Danovaro et al, 2016), 孢粉分析同样耗时耗力, 样品处理与定种需要相当专业且丰富的经验(Hubbard et al, 1983)2~3。植物分子生物学的发展很好地解决了上述方法中存在的缺陷(Pagani et al, 20091~4; Erb et al, 20181~3)。Thompson等(2014)的研究指出, 分子分类学工具的成本比野外调查的成本低37%; 有研究提出在缺氧和有水存在的环境中可以较好地保存植物分子材料(Brown et al, 1993), 而海洋沉积物正是可以保存海相和陆相植物分子材料的极佳环境(Bálint et al, 2018; Estrada et al, 2018)。
到目前为止, 研究者已从大于55 ka BP的湖泊沉积物中提取并分析了植物DNA, 表明对湖泊沉积物的植物DNA进行的研究可能跨越晚更新世和全新世(Willerslev et al, 2003)。此外, 对从沉积物中提取并扩增的植物DNA进行分析可以反演周边植被情况(Willerslev et al, 2014; Yang et al, 2020b6, 20218; Li et al, 2021a4)。因此, 沉积物中的植物DNA是一种非常理想的长期监测植被情况的材料。大量关于沉积物中植物DNA提取及其应用的相关研究被报道(Willis et al, 1999; Estrada et al, 2018; Lézine et al, 2019), 然而, 多数关于沉积物中植物DNA的研究都集中在湖泊、冻土等沉积物类型中(林清等, 2002; 严东娜等, 2019; 马瑞丰等, 2020), 截至目前, 尚无从海洋沉积物中提取植物DNA的研究。
海洋沉积物中提取DNA的研究也存在一定局限性, 即便是在较为理想的环境下保存的DNA仍然会随着时间的推移受到严重的损伤和降解(Roberts et al, 2013), 因此, 如何对海洋沉积物中短片段、低浓度的DNA进行提取和PCR扩增是非常具有挑战性的课题。目前, 商用试剂盒被广泛应用于提取沉积物中的DNA (Larsen et al, 2015; Ramírez et al, 2018), 且聚合酶链反应(PCR)可以在大约3~4 h内实现超过100万倍的DNA扩增(Compton, 1991)。一般认为, 植物叶绿体基因具有非常保守的特点, 是一种理想的DNA条形码(Chung et al, 2003; Heinze, 2007)。因此, 基于植物叶绿体基因开发的引物在应用于扩增植物DNA时具有更好的效果(Li et al, 2011)。由于扩增出的条带质量除了与引物有关外(沈阳阳等, 2020; 法文龙等, 2022), 还与PCR体系中的2倍浓缩的PCR扩增预混合溶液(Mix)、退火温度、循环数以及加入DNA模板的量等因素有关(Saiki et al, 1988), 因此, 在方法探索过程中要根据需要对上述条件进行摸索优化。
基于上述背景, 本文着重探讨海洋沉积物中的植物DNA能否被成功提取和扩增?若能够成功扩增, 那么能否通过优化提取和扩增条件来实现高效提取和扩增?本实验对采自黄海潮间带和浅海的表层沉积物样品进行DNA提取方法探索, 并确定最适提取方法。此外, 使用不同的PCR特异性引物对植物叶绿体基因的部分序列进行PCR扩增方法探索并对结果进行比较分析, 最终探索出最适PCR扩增条件。本文旨在探索沉积物中植物DNA的提取方法并通过选择合适的引物验证海洋沉积物中植物叶绿体DNA的PCR扩增效能, 从而为涉及不同海区类型沉积物中植物DNA的提取和PCR扩增以及海洋沉积物中植物的分子多样性和分子生态学等研究提供更加可靠的实验方法和参考依据。
1 材料与方法 1.1 样品采集本实验采用的表层沉积物样品分别采自黄海潮间带和浅海(图 1)。首先, 选取潮间带未受外界扰动的区域, 根据离岸的远近选取3个站位, 3个站位均位于中潮带, 采样当日的最高潮为3.75 m, 最低潮为2.17 m。使用一次性采样勺刮取表层0~2 cm的沉积物, 再将沉积物样品分别装到不同的样品袋中并编号, 放入可携带式冰盒保温取样箱中。回到实验室后, 将样品铺平后放入−80 ℃冰箱中冷冻保存。2019年6月11日搭乘“科学三号”考察船, 使用箱式采泥器采集黄海南部区域(图 1)的沉积物样品。样品采集步骤如下: 使用一次性采样勺刮取表层0~2 cm的沉积物, 放入干净的密封袋中并标号记录站位信息, 将样品铺平并放置于−80 ℃冰箱中保存。本研究选取了浅海6个站位的样品(3500-04、3500-05、3500-06、3500-07、3500-08、3500-10)进行实验。
 |
| 图 1 采样站位区域分布图 Fig. 1 Distribution of the sampling sites 注: 图中“▲”是本实验采样站位 |
实验通过采用不同DNA提取试剂盒、不同品牌Mix、改变PCR退火温度、循环数及DNA模板量和使用不同引物(表 1, 2)来比较DNA提取效果和PCR扩增效率。在预实验中发现, gh引物的扩增效果较差, 而rbcL引物在初始条件下就可以扩增出质量较高的条带, 因此实验条件的摸索仅针对gh引物。
| 样品类型 | 引物 | 试剂盒 | Mix | 模板量 | 退火温度 | 循环数 | 变量 | |
| 方法A1 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 55 ℃ | 55 × | 试剂盒 |
| 方法A2 | 潮间带 | gh | DM | 2×High-Fidelity PCR Master Mix | 2 μL | 55 ℃ | 55 × | |
| 方法A3 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 50 ℃ | 55 × | 退火温度 |
| 方法A4 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 52 ℃ | 55 × | |
| 方法A5 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 54 ℃ | 55 × | |
| 方法A6 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 56 ℃ | 55 × | |
| 方法A7 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 58 ℃ | 55 × | |
| 方法A8 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 60 ℃ | 55 × | |
| 方法A9 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 62 ℃ | 55 × | |
| 方法A10 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 56 ℃ | 60 × | 循环数 |
| 方法A11 | 潮间带 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 56 ℃ | 55 × | |
| 方法A12 | 潮间带 | rbcL | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 60 ℃ | 45 × | 引物 |
| 注: 本研究设计的针对潮间带样品中植物DNA的提取和扩增方法, 其中DS代表DNeasy PowerSoil土壤微生物DNA提取试剂盒, DM代表DNeasy PowerMax土壤DNA提取试剂盒 | ||||||||
| 样品类型 | 引物 | 试剂盒 | Mix | 模板量 | 退火温度 | 循环数 | 变量 | |
| 方法B1 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 55 ℃ | 45 × | 试剂盒 |
| 方法B2 | 浅海陆架 | gh | DM | 2×High-Fidelity PCR Master Mix | 2 μL | 55 ℃ | 45 × | |
| 方法B3 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 52 ℃ | 45 × | 退火温度 |
| 方法B4 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 53 ℃ | 45 × | |
| 方法B5 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 54 ℃ | 45 × | |
| 方法B6 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 56 ℃ | 45 × | |
| 方法B7 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 58 ℃ | 45 × | |
| 方法B8 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 59 ℃ | 45 × | |
| 方法B9 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 60 ℃ | 45 × | |
| 方法B10 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 61 ℃ | 45 × | |
| 方法B11 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 59 ℃ | 45 × | 循环数 |
| 方法B12 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 59 ℃ | 50 × | |
| 方法B13 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 59 ℃ | 55 × | |
| 方法B14 | 浅海陆架 | gh | DS | DreamTaq Green PCR Master Mix | 2 μL | 59 ℃ | 45 × | Mix |
| 方法B15 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 59 ℃ | 45 × | |
| 方法B16 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 59 ℃ | 45 × | DNA模板量 |
| 方法B17 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 4 μL | 59 ℃ | 45 × | |
| 方法B18 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 6 μL | 59 ℃ | 45 × | |
| 方法B19 | 浅海陆架 | gh | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 59 ℃ | 45 × | 引物 |
| 方法B20 | 浅海陆架 | rbcL | DS | 2×High-Fidelity PCR Master Mix | 2 μL | 60 ℃ | 45 × | |
| 注: 本研究设计的针对浅海陆架样品中植物DNA的提取和扩增方法, 其中DS代表DNeasy PowerSoil土壤微生物DNA提取试剂盒, DM代表DNeasy PowerMax土壤DNA提取试剂盒 | ||||||||
使用两种试剂盒分别提取三个潮间带和六个浅海表层沉积物样品的环境DNA。每次称取约0.25 g沉积物样品, 每5个样品设置一个空白对照。此方法的所有提取步骤均按照说明书进行, 涡旋震荡时间为20 min, 洗脱液体积为100 μL。完成eDNA提取后, 利用Qubit @3.0荧光计(Thermo Scientific)检测eDNA的浓度。
1.4 PCR扩增将用不同试剂盒提取的两组DNA进行PCR扩增实验。PCR扩增实验采用的是25 μL的扩增体系, 该体系包括12.5 μL的Mix、0.5 μL的10 μmol/L引物(gh引物和rbcL引物的具体信息见表 3)、2 μL的DNA模板和9.5 μL的双蒸水(ddH2O)。实验中, 当改变DNA模板量时, 通过调整ddH2O的体积维持扩增体系体积的不变。优化前gh引物的PCR扩增程序(简称程序a)和rbcl引物的扩增程序(简称程序b)如表 4所示。将得到的PCR产物与1×loading buffer等体积混合, 并在浓度为1%的琼脂糖凝胶上进行电泳, 将凝胶放置在凝胶成像系统进行检测。每次实验前用核酸清洁剂彻底清洁实验台面和仪器周围空间, 喷洒核酸清洁剂到超净台面并开启紫外灯杀菌, 排除其他环境DNA对本实验的干扰。
| 引物名称 | 序列(5′~3′) | 目的片段长度 | 文献来源 |
| g | GGGCAATCCTGAGCCAA | 40 bp | Bradley et al, 2007 |
| h | CCATTGAGTCTCTGCACCTATC | ||
| rbcLZ1 | ATGTCACCACAAACAGAGACTAAAGCAAGT | 157 bp | Taberlet et al, 2007 |
| rbcL19b | CTTCTTCAGGTGGAACTCCAG |
| 引物 | 预变性 | 变性 | 退火 | 延伸 | 循环数 | 最后延伸 | 文献来源 | |
| 程序a | gh | 95 ℃/10 min | 95 ℃/30 s | 55 ℃/30 s | 72 ℃/1 min | 55 × | 72 ℃/10 min | Bradley et al, 2007 |
| 程序b | rbcL | 94 ℃/10 min | 92 ℃/15 s | 60 ℃/1 min | 72 ℃/1 min | 45 × | 72 ℃/10 min | Taberlet et al, 2007 |
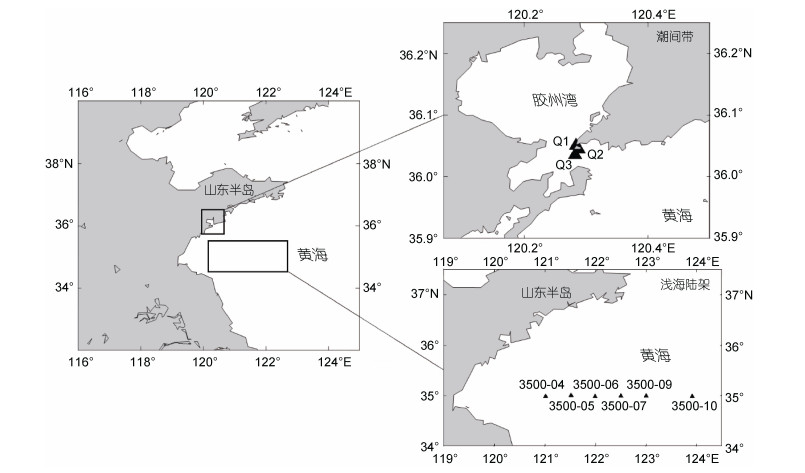
使用方法A1和方法A2分别提取潮间带3个站位的表层样品(0~2 cm), 共获得6组DNA序列, 其中, 使用方法A1提取的DNA为1~3号泳道, 使用方法A2提取的DNA为4~6号泳道。6组DNA序列使用程序a进行扩增。再分别使用方法B1和方法B2提取浅海3个站位(3500-04、3500-05、3500-06)的样品, 获得6组DNA (分别对应7~9号泳道和10~12号泳道)。在使用方法B1对浅海3500-4站位提取DNA时, 分别加入2和3 g的沉积物样品以比较加入不同样品克数时的提取与扩增质量, 样品泳道标号为13和14。使用程序a进行PCR扩增, 得到的条带如图 2所示。
 |
| 图 2 黄海潮间带和浅海沉积物样品使用不同试剂盒的扩增结果 Fig. 2 Amplification results of the Yellow Sea intertidal zone and shallow sea sediment samples using different kits 注: 图a为潮间带样品; 图b为浅海陆架样品 |
根据凝胶电泳图像, 可以观察到不同方法提取的DNA的PCR产物凝胶电泳条带存在亮度差异。为了进一步比较两者的差异, 使用Image J处理上述胶图, 并获得了各个条带的灰度值(表 5)。结果表明, 方法A1和方法B1明显优于方法A2和方法B2, 即DNeasy PowerSoil土壤微生物DNA提取试剂盒的效果好于DNeasy PowerMax土壤DNA提取试剂盒。
| 泳道编号 | 灰度值 | 泳道编号 | 灰度值 | 泳道编号 | 灰度值 | 泳道编号 | 灰度值 | |||
| 1 | 25 777 | 6 | 1 4656 | 11 | 19 887 | 16 | 27 819 | |||
| 2 | 22 361 | 7 | 25 698 | 12 | 25 587 | 17 | 19 461 | |||
| 3 | 23 019 | 8 | 26 461 | 13 | 24 235 | 18 | 28 736 | |||
| 4 | 18 736 | 9 | 23 186 | 14 | 21 568 | 19 | 26 844 | |||
| 5 | 14 584 | 10 | 20 132 | 15 | 13 077 | 20 | 27 656 | |||
| 注: 对无法直接观察到目的条带质量差别的泳道进行了灰度值分析以确定最优条件 | ||||||||||
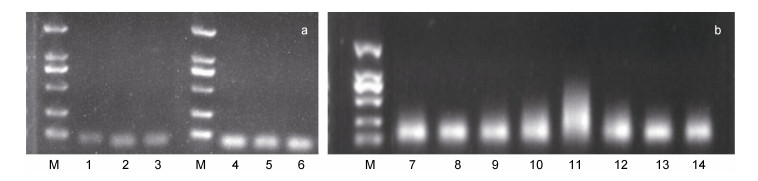
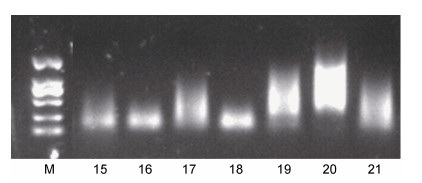
使用方法A3~A9对潮间带样品进行PCR扩增退火温度的探索, 不同退火温度下获得的凝胶电泳图如图 3所示。将所获得的电泳图用Image J图像软件分析, 得到各条带的灰度值(表 5)。实验结果表明, 方法A6明显优于其他方法, 即56 ℃为潮间带样品使用gh引物的最佳退火温度。使用方法B3~B10对浅海3500-04站位进行PCR扩增的退火温度探索, 27~29号泳道扩增效果无明显差别(图 4a), 因此为了进一步验证这三种方法的效果, 使用方法B8~B10对浅海其他5个站位(3500-5、6、7、8和3500-10)的样品进行实验。获得凝胶电泳图如图 4b所示。结果表明, 方法B8明显优于其他方法, 即59 ℃为浅海样品使用gh引物的最佳退火温度。
 |
| 图 3 黄海潮间带样品不同退火温度下的扩增结果 Fig. 3 Amplification results of intertidal samples from the Yellow Sea at different annealing temperatures 注: 15~21号泳道分别为退火温度为50~62 ℃时的扩增结果 |
 |
| 图 4 黄海浅海样品不同退火温度下的扩增结果 Fig. 4 Amplification results of shallow sea samples from the Yellow Sea at different annealing temperatures 注: 图a为3500-04站位样品, 退火温度依次为52、53、54、56、58、59、60和61 ℃; 图b为59、60、61 ℃的退火温度下其余5个站位的结果 |
使用方法A10对潮间带样品进行PCR循环数的探索。使用方法B11~B13对浅海4个站位的样品(3500-4、3500-5、3500-6、3500-7)进行PCR循环数的探索。获得凝胶电泳图如图 5所示。由结果可知, 方法A10出现了明显的拖带现象, 因此55次循环数为潮间带样品的最适循环数。对于浅海样品, 方法B11明显优于其他方法, 并得出最适循环数为45次的结论。
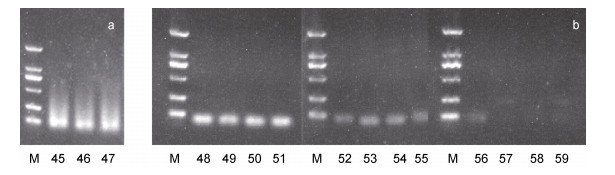 |
| 图 5 黄海潮间带样品和浅海样品不同循环数下的扩增结果 Fig. 5 Amplification results of intertidal samples and shallow sea samples from the Yellow Sea in different numbers of cycling 注: 图a为潮间带3个站位60次循环的扩增结果; 图b为浅海4个站位45、50、55次循环的扩增结果 |
分别使用方法B14、B15对浅海6个站位的样品进行PCR体系中Mix的探索, 获得的凝胶电泳条带如图 6a所示。由电泳结果可知, 方法B15明显优于方法B14, 因此2×High-Fidelity PCR Master Mix为最佳Mix。
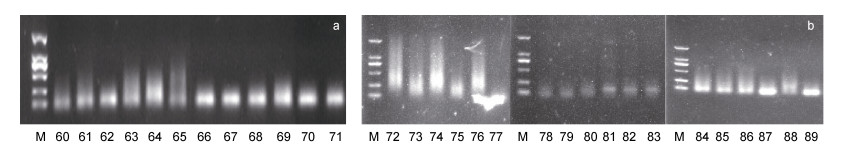 |
| 图 6 黄海浅海样品不同实验条件下的扩增结果 Fig. 6 Amplification results of shallow sea samples from the Yellow Sea under different experimental conditions 注: 图a为使用不同Mix扩增的结果, 60~71号; 图b为使用不同DNA模板加入量的扩增结果, 72~77号泳道为6 μL DNA模板, 78~83为4 μL DNA模板, 84~89为2 μL DNA模板 |
使用方法B16~B18对浅海6个站位的样品进行PCR体系中模板量的探索, 获得凝胶电泳图如图 6b所示。结果表明, 方法B16明显优于其他方法, 因此, 确定2 μL为最适模板量。
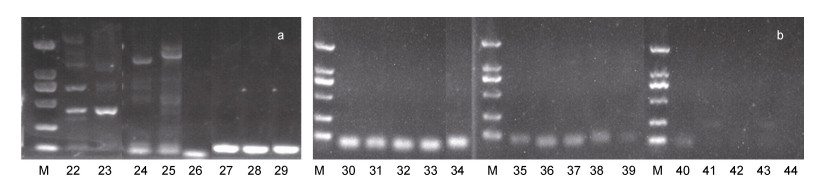
2.6 海洋沉积物中植物DNA的扩增方法探索——引物的比较使用方法A11、A12对潮间带样品进行引物效果的比较(图 7a, 图 8), 使用方法B19、B20对浅海样品进行引物效果的比较(图 7b, 图 8)。由实验结果可知, 方法A11明显优于方法A12, 表明优化条件后的gh引物为潮间带样品最适引物。同样地, 方法B19明显优于方法B20, 说明优化条件后的gh引物同样为浅海样品的最适引物。
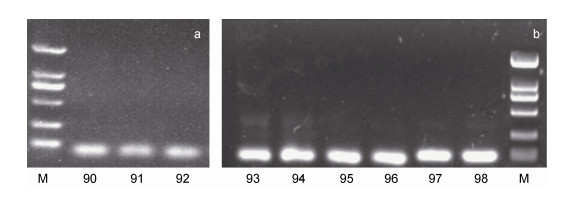 |
| 图 7 黄海潮间带和浅海样品使用优化后的组合条件下gh引物的扩增结果 Fig. 7 Amplification results of gh primers under optimized combination conditions for intertidal and shallow sea samples from the Yellow Sea 注: 图a为潮间带样品的扩增结果; 图b为浅海样品的扩增结果 |
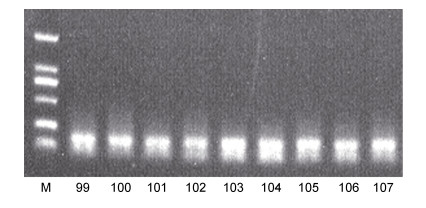 |
| 图 8 黄海潮间带和浅海样品使用rbcL引物的扩增结果 Fig. 8 Amplification results of rbcL primers for intertidal and shallow sea samples from the Yellow Sea 注: 99~101号泳道为潮间带样品的扩增结果, 102~107号泳道为浅海样品的扩增结果 |
到目前为止, 本课题组已经对海洋沉积物中的古海洋环境变化指示生物——有孔虫进行了较为系统的研究(Lei et al, 2014, 2015, 2017; Dong et al, 2020; Li et al, 2020), 同时也对海洋沉积物中的古代陆地环境变化指示生物——植物进行了孢粉分析研究(Yang et al, 2020b2~6, 20212~8)。但孢粉分析耗时耗力(Hubbard et al, 19832~3; Pagani et al, 20091~4; Erb et al, 20181~3)且目前国内外尚无从海洋沉积物中提取植物DNA的研究。基于此, 本实验对采自黄海潮间带和浅海表层沉积物样品进行植物DNA的提取和扩增方法探索, 从而为涉及不同海区类型沉积物中植物DNA的提取和PCR扩增以及海洋沉积物中植物的分子多样性和分子生态学等研究提供重要的实验方法和参考依据。
3.1 实验条件的探索一般地, 通过凝胶电泳可以有效判断DNA提取与扩增结果的好坏和引物的优劣(Zhang et al, 2003; Chatterjee et al, 2004; Sanchez et al, 2007; 王爱娜等, 2012; Shang et al, 2013; 徐鹏昊等, 2018)。基于此, 本研究通过对不同的实验条件进行优化、对比和选择, 将不同条件下提取的DNA以及使用gh引物进行PCR扩增得到的产物进行凝胶电泳分析, 进而得到提取DNA的最佳方法以及gh引物的最适条件。
3.1.1 海洋沉积物中植物DNA的提取方法探索由图 2a可知, 两种试剂盒(即DNeasy PowerSoil试剂盒与DNeasy PowerMax)均能成功提取黄海潮间带表层样品的植物DNA。其中, DNeasy PowerSoil试剂盒需要添加0.25 g样品(Evans et al, 2018; Lear et al, 2018; Mattei et al, 2019; Pearman et al, 2020a; Temesgen et al, 2020; 蔡杰等, 2022), 而DNeasy PowerMax试剂盒需要添加10 g样品(Lee et al, 2010; Jarosz et al, 2021)。对比可知, DNeasy PowerSoil试剂盒提取出的DNA的扩增条带更加明亮清晰, 所以DNeasy PowerSoil试剂盒的提取效能优于DNeasy PowerMax试剂盒。为了进一步验证这一结果是否具有通用性, 用上述两种试剂盒分别提取黄海浅海表层沉积物样品的DNA并进行比较, 得到的结果与黄海潮间带的规律一致(图 2b)。并且, 考虑到样品量的问题, 一般情况下没有必要使用10 g样品提取DNA (Tamarapu et al, 2001; 陈立等, 2004; Zhang et al, 2011; Lim et al, 2017), 因此, 推荐使用DNeasy PowerSoil试剂盒进行植物DNA提取。DNeasy PowerSoil试剂盒不仅能成功提取海洋沉积物中的植物DNA, 而且其操作流程简单且耗时更短(Song et al, 2021), 是一款理想的从海洋沉积物中提取植物DNA的试剂盒(Pearman et al, 2020b; van der Loos et al, 2021)。
3.1.2 海洋沉积物中植物DNA的扩增方法探索由图 3和表 4可知, 16和18号泳道的条带较亮且没有拖带, 且18号泳道的PCR产物条带最亮, 由此可得出, 56 ℃是针对潮间带沉积物样品扩增的最佳退火温度。由图 4a可知, 退火温度为59 ℃及以上时, 浅海沉积物样品的提取结果没有杂带和拖带现象, 且PCR扩增的特异性较好(Broude et al, 2001; Korbie et al, 2008; 李海阔等, 2009; 左丽娟等, 2011; Daher et al, 2014)。若退火温度过高, 则会引起引物与模板结合不完全, 从而影响引物的扩增效率, 导致出现的目的条带变暗甚至没有目的条带(Osborne, 1992; Hamajima et al, 2002; 冉姝等, 2008; 程远霞等, 2008)。因此, 本研究选择59 ℃作为扩增浅海沉积物样品的最佳退火温度。除此之外, 本文还针对潮间带沉积物样品进行了PCR循环数的比较, 由图 5a可知, 当把PCR循环数增加至60次, 出现了明显的拖带现象, 目的条带也不够明亮, 这可能是由非特异性产物增多造成的(Mishra et al, 2002; Gomez et al, 2005; Sipos et al, 2007; Ma et al, 2009, 2011a, 2011b; He et al, 2013; Shao et al, 2017), 因此仍然延续原程序中的55次循环数。而对于浅海沉积物样品, 分别对45次、50次和55次的PCR扩增循环数结果进行比较, 根据图 5b可知45次循环数的表现最好且最稳定, 因此将45次循环数确定为最适合浅海沉积物样品的PCR扩增条件。综上所述, 即使是同一对引物, 在扩增不同样品时的最佳退火温度和循环数等条件也可能不同(Quirós et al, 1998; Chaudhry et al, 1999; Omori et al, 2000; 龙平等, 2013; 王彦坤等, 2020), 因此在进行实验前需要进行相关预实验以探索适合实验样品的PCR扩增条件(Lemmer et al, 2004; Leli et al, 2019)。
此外, 还针对浅海沉积物样品进行了PCR扩增体系中DNA模板量以及Mix的比较, 由图 6a可知, 66~71号泳道的目的条带清晰且明亮, 无杂带和拖带现象, 因此, 使用2× TransTaq HiFi PCR SuperMixⅠ进行PCR扩增得到的产物明显优于使用DreamTaq Green PCR Master Mix(2X)进行PCR扩增得到的产物。由图 6b可知, DNA模板的添加量对PCR扩增也具有一定的影响(张部昌等, 2002; 李宗菊等, 2004; Stiller et al, 2012; Sun et al, 2014; 田孟祥等, 2015), 本研究中目的条带随DNA模板量增加而变浅, 甚至很多层位浅至没有条带, 与预期的模板量越多条带越亮(宋美茹等, 2012)不符。通过比较, 本研究选择加入2 μL DNA模板进行PCR扩增, 以达到最佳效果。
3.2 不同海区类型的最优实验条件 3.2.1 潮间带根据上述对各个实验条件的探索, 本文总结出了黄海潮间带沉积物表层样品的植物DNA最佳提取条件和使用gh引物时的最佳扩增条件, 即使用DNeasy PowerSoil土壤微生物DNA提取试剂盒, 使用25 μL PCR反应体系(Riera et al, 2010; Barrera et al, 2016; Carvalho et al, 2016)包括12.5 μL 2×Taq PCR Master Mix, 正、反引物各0.5 μL, DNA模板2 μL, 去离子水9.5 μL; 优化后的PCR扩增程序为: 95 ℃预变性10 min, 95 ℃变性30 s, 56 ℃退火30 s, 72 ℃延伸1 min, 共55个循环, 最后72 ℃延伸10 min。使用优化后的实验条件对黄海潮间带沉积物表层样品进行PCR扩增后的结果如图 7a所示, 潮间带三个站位的样品均能通过gh引物扩增出位置正确、清晰且明亮的条带, 证明gh引物能在上述实验条件下正确高效的扩增出潮间带沉积物样品的植物DNA片段(Tonooka et al, 2009)。
3.2.2 浅海由上述实验条件探索可知, 黄海浅海沉积物表层样品的植物DNA最佳提取条件和使用gh引物时的最佳扩增条件为: 使用DNeasy PowerSoil土壤微生物DNA提取试剂盒加入2 g样品进行提取, 使用25 μL PCR反应体系(Fishback et al, 1999; Moore, 2005; Lee, 2010; Pan et al, 2010; Saingam et al, 2018)包括12.5 μL 2× TransTaq HiFi PCR SuperMixⅠ, 正、反引物各0.5 μL, 2 μL DNA模板, 去离子水9.5 μL; 优化后的PCR扩增程序为: 95 ℃预变性10 min, 95 ℃变性30 s, 59 ℃退火30 s, 72 ℃延伸1 min, 共45个循环, 最后72 ℃延伸10 min。在优化的组合条件下对黄海浅海沉积物样品进行PCR扩增, 结果如图 7b所示。可以看出, 浅海6个站位的样品均能通过gh引物扩增出位置正确、清晰且明亮的条带, 表明gh引物在优化的实验条件下能够正确高效的扩增出浅海沉积物表层样品的植物DNA片段(张祥等, 2012)。
3.3 引物的对比使用优化后的实验条件对含有gh引物的PCR体系进行扩增, 并与rbcL引物的扩增结果进行比较。由图 7和图 8可以看出, 99~107号泳道虽然可以扩增出清晰明亮的条带, 但是有引物二聚体(Kolocheva et al, 1993; Kusumoto et al, 2004; Desmarais et al, 2012; Poritz et al, 2014; Satterfield, 2014; Chen et al, 2016; Meagher et al, 2018; Johnston et al, 2019; Hendling et al, 2021; Xie et al, 2022)产生, 尽管后期可以通过切胶回收去除引物二聚体(Brownie et al, 1997; Høgdall et al, 1999; 黄天谊等, 2005; Salimullah et al, 2009; Li et al, 2017; Tian et al, 2018; Soares et al, 2021), 但在增加了工作量的同时还容易引起不必要的误差(Buck et al, 1999; Nybo, 2011; Vandenbroucke et al, 2011; Karakuş et al, 2017; Yazdi et al, 2018; Xie et al, 2022); 而90~98号泳道的目的条带清晰、明亮且无杂带和拖带现象, 说明利用优化后的组合实验条件对含有gh引物的PCR体系进行扩增的结果明显优于rbcL引物的扩增结果。因此, gh引物是理想的扩增海洋沉积物中植物DNA的引物, 并且该引物具有较好的通用性, 适用于潮间带和浅海沉积物表层样品(彭程等, 2012)。
4 结论与展望本研究通过对比不同海区沉积物表层样品的DNA提取方法以及所选取的两组引物(即gh引物和rbcL引物)对沉积物中DNA进行的PCR扩增结果, 得出如下结论:
(1) 对于黄海潮间带表层沉积物样品, 建议使用DNeasy PowerSoil土壤微生物DNA提取试剂盒进行DNA提取, 使用2× TransTaq HiFi PCR SuperMixⅠ并加入2 μL DNA模板进行PCR扩增, 最佳退火温度为55 ℃, 最佳循环数为55个循环。
(2) 对于黄海浅海表层沉积物样品, 建议使用DNeasy PowerSoil土壤微生物DNA提取试剂盒进行DNA提取, 样品加入量为2 g, 使用2× TransTaq HiFi PCR SuperMixⅠ, 最佳退火温度为59 ℃, 最佳循环数为45个循环。
(3) 在引物选择方面, 与gh引物相比, rbcL引物虽然更容易扩增出目的条带, 但同时伴随有引物二聚体产生, 因此针对类似的潮间带和浅海样品, 本文推荐使用gh引物进行PCR扩增。
综上所述, 本研究探索并优化了海洋沉积物中植物DNA的提取和PCR扩增条件, 同时证实了海洋沉积物能够有效保存植物的DNA, 不仅为海洋沉积物中植物分子生物学研究提供了理论和技术支持, 更为陆地植被重建领域开拓了新思路。
马瑞丰, 张威, 刘亮, 等, 2020. 沉积植物古DNA技术在古植被重建中的应用. 沉积学报, 38(6): 1179-1191 |
王彦坤, 李天楚, 粘景梓, 等, 2020. 通用引物双重PCR在节肢动物物种鉴定中的应用. 昆虫学报, 63(1): 97-103 |
王爱娜, 王灏, 李殿荣, 等, 2012. 琼脂糖与聚丙烯酰胺凝胶电泳在SSR鉴定杂交油菜种子纯度中的比较. 西北农业学报, 21(8): 101-106 |
左丽娟, 高勇, 张庙见, 等, 2011. 植物高度同源重复基因的PCR检测. 四川大学学报(自然科学版), 48(3): 679-684 |
龙平, 崔占虎, 李虔全, 等, 2013. 基于位点特异性PCR的黄芪与红芪鉴别方法研究. 中国中药杂志, 38(16): 2581-2585 |
田孟祥, 张时龙, 余本勋, 等, 2015. 一种应用PCR缓冲液快速制备水稻DNA模板的方法. 分子植物育种, 13(2): 438-442 |
冉姝, 周国燕, 王景宇, 等, 2008. 聚合酶链式反应变性及退火温度与产物假阴性之间的关系. 中国生物制品学杂志, 21(11): 1016-1018, 1029 |
严东娜, 徐海, 侯卫国, 2019. 现代分子生物技术在湖泊沉积物古DNA研究中的应用. 生态学杂志, 38(7): 2200-2209 |
李宗菊, 熊丽, 桂敏, 等, 2004. 非洲菊基因组DNA提取及ISSR-PCR扩增模板浓度优化. 云南植物研究, 26(4): 439-444 |
李海阔, 曹雪雁, 周智磊, 等, 2009. 纳米金增强复杂体系中PCR扩增低拷贝基因的反应特异性初步研究. 中国生物工程杂志, 29(8): 75-80 |
沈阳阳, 类彦立, 李浩天, 等, 2020. 不同PCR引物对多种海洋生境沉积物中的有孔虫DNA扩增效能的比较研究. 微体古生物学报, 37(4): 368-380 |
宋美茹, 姚萍, 张跃新, 2012. QIAamp及Biomed粪便细菌基因组DNA提取试剂盒提取人肠道细菌基因组DNA质量的差异. 中国组织工程研究, 16(11): 2015-2018 |
张佳华, 姚凤梅, 2000. 陆地表面复杂过程中植物生态系统的作用. 水土保持学报, 14(4): 55-59 |
张部昌, 赵志虎, 于秀琴, 等, 2002. 富含GC DNA PCR扩增条件的优化. 军事医学科学院院刊, 26(4): 257-261 |
张祥, 李晓博, 谷素美, 等, 2012. 不同肠球菌基因组DNA提取方法对PCR鉴定的影响. 河南农业大学学报, 46(1): 67-71 |
陈立, 姜莉, 罗阳, 等, 2004. 一种从人血凝块中提取基因组DNA的方法. 细胞生物学杂志, 26(5): 548-549 |
林清, 施苏华, 彭平安, 等, 2002. 青藏高原多年冻土沉积物中埋藏植物DNA特征的初步分析. 冰川冻土, 24(1): 28-31 |
法文龙, 李浩天, 李青霞, 等, 2022. 黄海沉积物柱状样中有孔虫古DNA的提取和PCR扩增的方法学初探. 微体古生物学报, 39(1): 70-84 |
徐鹏昊, 罗武松, 何恩明, 等, 2018. 16S rDNA特异性引物设计优化及其在淞江鲈体表微生物鉴定中的应用. 复旦学报(自然科学版), 57(1): 59-67, 78 |
黄天谊, 王世海, 黎学铭, 等, 2005. 标签引物-套式/多重PCR检测恶性疟原虫和间日疟原虫的研究. 中国寄生虫学与寄生虫病杂志, 23(3): 140-142 |
彭程, 刘全兰, 王洪光, 等, 2012. 山东鹅观草SSR-PCR反应体系的优化和验证. 生物学杂志, 29(2): 91-94 |
程远霞, 刘宝林, 华泽钊, 等, 2008. 变性温度和时间对实时定量聚合酶链反应检测乙型肝炎病毒的影响. 中国组织工程研究与临床康复, 12(44): 8677-8680 |
蔡杰, 王博, 胡孙林, 等, 2022. 玻璃珠-涡旋振荡改良法用于硅藻DNA提取. 法医学杂志, 38(1): 119-126 |
ADOLE T, DASH J, ATKINSON P M, 2016. A systematic review of vegetation phenology in Africa. Ecological Informatics, 34: 117-128 |
AHL D E, GOWER S T, BURROWS S N, et al, 2006. Monitoring spring canopy phenology of a deciduous broadleaf forest using MODIS. Remote Sensing of Environment, 104(1): 88-95 |
BÁLINT M, PFENNINGER M, GROSSART H P, et al, 2018. Environmental DNA time series in ecology. Trends in Ecology & Evolution, 33(12): 945-957 |
BARRERA G, MURCIA J, CERÓN J, et al, 2016. PCR en tiempo real: una metodología útil para la detección y cuantificación de granulovirus. Revista Colombiana de Biotecnología, 18(2): 24-31 |
BRADLEY B J, STILLER M, DORAN-SHEEHY D M, et al, 2007. Plant DNA sequences from feces: potential means for assessing diets of wild primates. Am J Primatol, 69(6): 699-705 |
BROUDE N E, ZHANG L G, WOODWARD K, et al, 2001. Multiplex allele-specific target amplification based on PCR suppression. Proceedings of the National Academy of Sciences of the United States of America, 98(1): 206-211 |
BROWN T A, ALLABY R G, BROWN K A, et al, 1993. Biomolecular archaeology of wheat: past, present and future. World Archaeology, 25(1): 64-73 |
BROWNIE J, SHAWCROSS S, THEAKER J, et al, 1997. The elimination of primer-dimer accumulation in PCR. Nucleic Acids Research, 25(16): 3235-3241 |
BUCK G A, FOX J W, GUNTHORPE M, et al, 1999. Design strategies and performance of custom DNA sequencing primers. Biotechniques, 27(3): 528-536 |
CARVALHO I A, SCHWARZ D G G, PIETRALONGA P A G, et al, 2016. Presença de Mycobacterium avium subsp. paratuberculosis (MAP) em pacientes brasileiros com doença inflamatória intestinal e em controles. Sao Paulo Medical Journal, 134(1): 13-19 |
CHATTERJEE P K, BAKER J C JR, 2004. Preparing nested- deletion template DNA for field inversion gel electrophoresis analyses and position-specific end sequencing with transposon primers [M] // ZHAO S Y, STODOLSKY M. Bacterial Artificial Chromosomes. Totowa: Humana Press: 243-254.
|
CHAUDHRY U, SALUJA D, 1999. Detection of Neisseria Gonorrhoeae by polymerase chain reaction (PCR). Indian Journal of Clinical Biochemistry, 14(2): 135-142 |
CHEN M H, LI Q Y, ZHANG L L, et al, 2008. Systematic biotic responses to palaeoenvironmental change in the Late Pleistocene southern South China Sea: a preliminary study. Journal of Quaternary Science, 23(8): 803-815 |
CHEN H J, XU Z K, BAYON G, et al, 2022. Enhanced hydrological cycle during Oceanic Anoxic Event 2 at southern high latitudes: new insights from IODP Site U1516. Global and Planetary Change, 209: 103735 |
CHEN F, ZHANG D X, ZHANG Q, et al, 2016. Zero-background helicase-dependent amplification and its application to reliable assay of telomerase activity in cancer cell by eliminating primer-dimer artifacts. Chembiochem, 17(12): 1171-1176 |
CHUNG S M, STAUB J E, 2003. The development and evaluation of consensus chloroplast primer pairs that possess highly variable sequence regions in a diverse array of plant taxa. Theoretical and Applied Genetics, 107(4): 757-767 |
CHYTRÝ M, SCHAMINÉE J H J, SCHWABE A, 2011. Vegetation survey: a new focus for Applied Vegetation Science. Applied Vegetation Science, 14(4): 435-439 |
COMPTON J, 1991. Nucleic acid sequence-based amplification. Nature, 350(6313): 91-92 |
CORINALDESI C, BEOLCHINI F, DELL'ANNO A, 2008. Damage and degradation rates of extracellular DNA in marine sediments: implications for the preservation of gene sequences. Molecular Ecology, 17(17): 3939-3951 |
COSTANZA R, DE GROOT R, SUTTON P, et al, 2014. Changes in the global value of ecosystem services. Global Environmental Change, 26: 152-158 |
CUNI-SANCHEZ A, SULLIVAN M J P, PLATTS P J, et al, 2021. High aboveground carbon stock of African tropical montane forests. Nature, 596(7873): 536-542 |
DAHER R K, STEWART G, BOISSINOT M, et al, 2014. Isothermal recombinase polymerase amplification assay applied to the detection of group B streptococci in vaginal/anal samples. Clinical Chemistry, 60(4): 660-666 |
DANOVARO R, CARUGATI L, BERZANO M, et al, 2016. Implementing and innovating marine monitoring approaches for assessing marine environmental status. Frontiers in Marine Science, 3: 213 |
DESMARAIS S M, LEITNER T, BARRON A E, 2012. Quantitative experimental determination of primer-dimer Formation risk by free-solution conjugate electrophoresis. Electrophoresis, 33(3): 483-491 DOI:10.1002/elps.201100452 |
DONG S S, LEI Y L, LI T G, et al, 2020. Response of benthic foraminifera to pH changes: community structure and morphological transformation studies from a microcosm experiment. Marine Micropaleontology, 156: 101819 DOI:10.1016/j.marmicro.2019.101819 |
ERB K H, KASTNER T, PLUTZAR C, et al, 2018. Unexpectedly large impact of forest management and grazing on global vegetation biomass. Nature, 553(7686): 73-76 DOI:10.1038/nature25138 |
ESTRADA O, BREEN J, RICHARDS S M, et al, 2018. Ancient plant DNA in the genomic era. Nature Plants, 4(7): 394-396 DOI:10.1038/s41477-018-0187-9 |
EVANS J S, LÓPEZ-LEGENTIL S, ERWIN P M, 2018. Comparing two common DNA extraction kits for the characterization of symbiotic microbial communities from ascidian tissue. Microbes and Environments, 33(4): 435-439 DOI:10.1264/jsme2.ME18031 |
FARRELL C, SZOTA C, ARNDT S K, 2015. Urban plantings: 'living laboratories' for climate change response. Trends in Plant Science, 20(10): 597-599 DOI:10.1016/j.tplants.2015.08.006 |
FISHBACK A G, DANZMANN R G, SAKAMOTO T, et al, 1999. Optimization of semi-automated microsatellite multiplex polymerase chain reaction systems for rainbow trout (Oncorhynchus mykiss). Aquaculture, 172(3/4): 247-254 |
GOMEZ M, WU X M, WANG Y L, 2005. Detection of BCL2-IGH using single-round PCR assays. Diagnostic Molecular Pathology, 14(1): 17-22 DOI:10.1097/01.pas.0000142167.64629.29 |
HAMAJIMA N, SAITO T, MATSUO K, et al, 2002. Competitive amplification and unspecific amplification in polymerase chain reaction with confronting two-pair primers. The Journal of Molecular Diagnostics, 4(2): 103-107 DOI:10.1016/S1525-1578(10)60688-5 |
HE C Z, ZHANG K H, WANG T, et al, 2013. Single-primer- limited amplification: a method to generate random single- stranded DNA sub-library for aptamer selection. Analytical Biochemistry, 440(1): 63-70 DOI:10.1016/j.ab.2013.05.008 |
HEINZE B, 2007. A database of PCR primers for the chloroplast genomes of higher plants. Plant Methods, 3: 4 DOI:10.1186/1746-4811-3-4 |
HENDLING M, BARIŠIĆ I, 2021. Computational design of multiplex oligonucleotide-based assays [M] // MARCHISIO M A. Computational Methods in Synthetic Biology. 2nd ed. New York: Humana: 19-30.
|
HØGDALL E, BOYE K, VUUST J, 1999. Simple preparation method of PCR fragments for automated DNA sequencing. Journal of Cellular Biochemistry, 73(4): 433-436 DOI:10.1002/(SICI)1097-4644(19990615)73:4<433::AID-JCB1>3.0.CO;2-Q |
HUBBARD R N L B, BOULTER M C, 1983. Reconstruction of Palaeogene climate from palynological evidence. Nature, 301(5896): 147-150 DOI:10.1038/301147a0 |
JAROSZ W, DURANT J F, IRENGE L M W B, et al, 2021. Optimized DNA-based identification of Toxocara spp. eggs in soil and sand samples. Parasites & Vectors, 14(1): 426 |
JIAN Z M, JIANG S J, JORDAN R W, 2020. Progress of micropaleontological research in the western Pacific marginal seas. Marine Micropaleontology, 161: 101938 DOI:10.1016/j.marmicro.2020.101938 |
JIAN Z M, JIN H Y, KAMINSKI M A, et al, 2019. Discovery of the marine Eocene in the northern South China Sea. National Science Review, 6(5): 881-885 DOI:10.1093/nsr/nwz084 |
JOHNSTON A D, LU J, RU K L, et al, 2019. PrimerROC: accurate condition-independent dimer prediction using ROC analysis. Scientific Reports, 9(1): 209 DOI:10.1038/s41598-018-36612-9 |
KARAKUŞ M, YILMAZ B, ÖZBEL Y, et al, 2017. A new cost and time effective method for multilocus microsatellite typing (MLMT) studies: application of Leishmania tropica isolates and clinical samples from Turkey. Journal of Microbiological Methods, 141: 97-100 DOI:10.1016/j.mimet.2017.08.012 |
KERN R A, SCHLESINGER W H, 1992. Carbon stores in vegetation. Nature, 357(6378): 447-448 |
KOLOCHEVA T I, LOKHOVA I A, TANSYKBAEV T A, et al, 1993. Recognition of T-T cyclobutane pyrimidine dimers in the form of primers by DNA polymerases. Molekuliarnaia Biologiia, 27(6): 1386-1393 |
KORBIE D J, MATTICK J S, 2008. Touchdown PCR for increased specificity and sensitivity in PCR amplification. Nature Protocols, 3(9): 1452-1456 DOI:10.1038/nprot.2008.133 |
KUSUMOTO R, MASUTANI C, SHIMMYO S, et al, 2004. DNA binding properties of human DNA polymerase η: implications for fidelity and polymerase switching of translesion synthesis. Genes To Cells, 9(12): 1139-1150 DOI:10.1111/j.1365-2443.2004.00797.x |
LARSEN A M, MOHAMMED H H, ARIAS C R, 2015. Comparison of DNA extraction protocols for the analysis of gut microbiota in fishes. FEMS Microbiology Letters, 362(5): fnu031 |
LEAR G, DICKIE I, BANKS J, et al, 2018. Methods for the extraction, storage, amplification and sequencing of DNA from environmental samples. New Zealand Journal of Ecology, 42(1): 10(1A-50A) |
LEE D S, 2010. Real-time PCR machine system modeling and a systematic approach for the robust design of a real-time PCR-on-a-chip system. Sensors, 10(1): 697-718 DOI:10.3390/s100100697 |
LEE J H, PARK Y, CHOI J R, et al, 2010. Comparisons of three automated systems for genomic DNA extraction in a clinical diagnostic laboratory. Yonsei Medical Journal, 51(1): 104-110 DOI:10.3349/ymj.2010.51.1.104 |
LEI Y L, LI T G, BI H S, et al, 2015. Responses of benthic foraminifera to the 2011 oil spill in the Bohai Sea, PR China. Marine Pollution Bulletin, 96(1/2): 245-260 |
LEI Y L, LI T G, NIGAM R, et al, 2017. Environmental significance of morphological variations in the foraminifer Ammonia aomoriensis (Asano, 1951) and its molecular identification: a study from the Yellow Sea and East China Sea, PR China. Palaeogeography, Palaeoclimatology, Palaeoecology, 483: 49-57 DOI:10.1016/j.palaeo.2016.05.010 |
LEI Y L, STUMM K, WICKHAM S A, et al, 2014. Distributions and biomass of benthic ciliates, foraminifera and amoeboid protists in marine, brackish, and freshwater sediments. Journal of Eukaryotic Microbiology, 61(5): 493-508 DOI:10.1111/jeu.12129 |
LELI C, DI MATTEO L, GOTTA F, et al, 2019. Diagnostic accuracy of a commercial multiplex PCR for the diagnosis of meningitis and encephalitis in an Italian general hospital. Le Infezioni in Medicina, 27(2): 141-148 |
LEMMER K, MANTKE O D, BAE H G, et al, 2004. External quality control assessment in PCR diagnostics of dengue virus infections. Journal of Clinical Virology, 30(4): 291-296 DOI:10.1016/j.jcv.2003.11.002 |
LÉZINE A M, IZUMI K, KAGEYAMA M, et al, 2019. A 90 000-year record of Afromontane forest responses to climate change. Science, 363(6423): 177-181 DOI:10.1126/science.aav6821 |
LI Y, GAO L M, POUDEL R C, et al, 2011. High universality of matK primers for barcoding gymnosperms. Journal of Systematics and Evolution, 49(3): 169-175 DOI:10.1111/j.1759-6831.2011.00128.x |
LI S L, GU Y, LYU Y, et al, 2017. Integrated graphene oxide purification-lateral flow test strips (iGOP-LFTS) for direct detection of PCR products with enhanced sensitivity and specificity. Analytical Chemistry, 89(22): 12137-12144 DOI:10.1021/acs.analchem.7b02769 |
LI Q X, LEI Y L, MORARD R, et al, 2020. Diversity hotspot and unique community structure of foraminifera in the world's deepest marine blue hole-Sansha Yongle Blue Hole. Scientific Reports, 10(1): 10257 DOI:10.1038/s41598-020-67221-0 |
LI J R, SHI X F, LIU S F, et al, 2021a. Frequency of deep-sea turbidity as an important component of the response of a source-to-sink system to climate: a case study in the eastern Middle Bengal Fan since 32ka. Marine Geology, 441: 106603 DOI:10.1016/j.margeo.2021.106603 |
LI K, STOOF-LEICHSENRING K R, LIU S S, et al, 2021b. Plant sedimentary DNA as a proxy for vegetation reconstruction in eastern and northern Asia. Ecological Indicators, 132: 108303 DOI:10.1016/j.ecolind.2021.108303 |
LIM H J, CHOI J H, SON A, 2017. Necessity of purification during bacterial DNA extraction with environmental soils. Environmental Health and Toxicology, 32: e2017013 DOI:10.5620/eht.e2017013 |
MA J, GUAN S C, YAO D, et al, 2011a. Problems with and a system to eliminate single-primer PCR product contamination in simple sequence repeat molecular marker-assisted selection in soybean. Genetics and Molecular Research, 10(3): 1659-1668 DOI:10.4238/vol10-3gmr1366 |
MA L, HE S B, HUANG J, et al, 2009. Maximizing specificity and yield of PCR by the quantum dot itself rather than property of the quantum dot surface. Biochimie, 91(8): 969-973 DOI:10.1016/j.biochi.2009.04.020 |
MA J, WANG P W, YAO D, et al, 2011b. Single-primer PCR correction: a strategy for false-positive exclusion. Genetics and Molecular Research, 10(1): 150-159 DOI:10.4238/vol10-1gmr988 |
MATTEI V, MURUGESAN S, AL HASHMI M, et al, 2019. Evaluation of methods for the extraction of microbial DNA from vaginal swabs used for microbiome studies. Frontiers in Cellular and Infection Microbiology, 9: 197 DOI:10.3389/fcimb.2019.00197 |
MEAGHER R J, PRIYE A, LIGHT Y K, et al, 2018. Impact of primer dimers and self-amplifying hairpins on reverse transcription loop-mediated isothermal amplification detection of viral RNA. Analyst, 143(8): 1924-1933 DOI:10.1039/C7AN01897E |
MISHRA R N, SINGLA-PAREEK S L, NAIR S, et al, 2002. Directional genome walking using PCR. Biotechniques, 33(4): 830-834 DOI:10.2144/02334st07 |
MOORE P, 2005. Replicating success. Nature, 435(7039): 235 |
MUHONG C, LANLAN Z, LILI Z, et al, 2008. Preservation of radiolarian diversity and abundance in surface sediments of the South China Sea and its environmental implication. Journal of China University of Geosciences, 19(3): 217-229 DOI:10.1016/S1002-0705(08)60041-2 |
NYBO K, 2011. qPCR: avoiding signals in the no-template control. BioTechniques, 50(4): 213-215 DOI:10.2144/000113648 |
OMORI Y, TANIGAMI A, SUGANO S, 2000. Comparative PCR: a simple and sensitive method for quantifying low- abundance mRNA species. Genomics, 67(2): 140-145 DOI:10.1006/geno.2000.6240 |
OSBORNE B I, 1992. HyperPCR: a Macintosh Hypercard program for the determination of optimal PCR annealing temperature. Bioinformatics, 8(1): 83 DOI:10.1093/bioinformatics/8.1.83 |
PAGANI M, CALDEIRA K, BERNER R, et al, 2009. The role of terrestrial plants in limiting atmospheric CO2 decline over the past 24 million years. Nature, 460(7251): 85-88 DOI:10.1038/nature08133 |
PAN X Y, JIANG L, LIU K Y, et al, 2010. A microfluidic device integrated with multichamber polymerase chain reaction and multichannel separation for genetic analysis. Analytica Chimica Acta, 674(1): 110-115 DOI:10.1016/j.aca.2010.06.005 |
PEARMAN J K, KEELEY N B, WOOD S A, et al, 2020a. Comparing sediment DNA extraction methods for assessing organic enrichment associated with marine aquaculture. PeerJ, 8: e10231 DOI:10.7717/peerj.10231 |
PEARMAN W S, WELLS S J, SILANDER O K, et al, 2020b. Concordant geographic and genetic structure revealed by genotyping-by-sequencing in a New Zealand marine isopod. Ecology and Evolution, 10(24): 13624-13639 DOI:10.1002/ece3.6802 |
PORITZ M A, RIRIE K M, 2014. Getting things backwards to prevent primer dimers. The Journal of Molecular Diagnostics, 16(2): 159-162 DOI:10.1016/j.jmoldx.2014.01.001 |
QI J, MARSETT R C, MORAN M S, et al, 2000. Spatial and temporal dynamics of vegetation in the San Pedro River Basin area. Agricultural and Forest Meteorology, 105(1/2/3): 55-68 |
QUIRÓS E, PIÉDROLA G, MAROTO C, 1998. GBV-RNA detection by polymerase chain reaction with several primer pairs. Microbios, 95(381): 125-130 |
RAMÍREZ G A, GRAHAM D, D'HONDT S, 2018. Influence of commercial DNA extraction kit choice on prokaryotic community metrics in marine sediment. Limnology and Oceanography: Methods, 16(9): 525-536 DOI:10.1002/lom3.10264 |
RIERA M A, ROJAS M E, ZAPATA P D, 2010. Protocolo de extracción de DNA por salting-out para pequeños volúmenes de sangre. Revista de Ciencia y Tecnología, 12(14): 4-7 |
ROBERTS D A, BIRCHENOUGH S N R, LEWIS C, et al, 2013. Ocean acidification increases the toxicity of contaminated sediments. Global Change Biology, 19(2): 340-351 DOI:10.1111/gcb.12048 |
SAIKI R K, GELFAND D H, STOFFEL S, et al, 1988. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science, 239(4839): 487-491 DOI:10.1126/science.2448875 |
SAINGAM P, LI B, YAN T, 2018. Use of amplicon sequencing to improve sensitivity in PCR-based detection of microbial pathogen in environmental samples. Journal of Microbiological Methods, 149: 73-79 DOI:10.1016/j.mimet.2018.05.005 |
SALIMULLAH M, KATO S, MURATA M, et al, 2009. Tunable fractionation of nucleic acids. Biotechniques, 47(6): 1041-1043 DOI:10.2144/000113249 |
SANCHEZ O, GASOL J M, MASSANA R, et al, 2007. Comparison of different denaturing gradient gel electrophoresis primer sets for the study of marine bacterioplankton communities. Applied and Environmental Microbiology, 73(18): 5962-5967 DOI:10.1128/AEM.00817-07 |
SATTERFIELD B C, 2014. Cooperative primers: 2.5 million-fold improvement in the reduction of nonspecific amplification. The Journal of Molecular Diagnostics, 16(2): 163-173 DOI:10.1016/j.jmoldx.2013.10.004 |
SHANG Y, ZHU P Y, XU W T, et al, 2013. Single universal primer multiplex ligation-dependent probe amplification with sequencing gel electrophoresis analysis. Analytical Biochemistry, 443(2): 243-248 DOI:10.1016/j.ab.2013.09.012 |
SHAO K K, SHI X H, ZHU X J, et al, 2017. Construction and optimization of an efficient amplification method of a random ssDNA library by asymmetric emulsion PCR. Biotechnology and Applied Biochemistry, 64(2): 239-243 DOI:10.1002/bab.1467 |
SIPOS R, SZÉKELY A J, PALATINSZKY M, et al, 2007. Effect of primer mismatch, annealing temperature and PCR cycle number on 16S rRNA gene-targetting bacterial community analysis. FEMS Microbiology Ecology, 60(2): 341-350 DOI:10.1111/j.1574-6941.2007.00283.x |
SOARES R R G, AKHTAR A S, PINTO I F, et al, 2021. Sample-to-answer COVID-19 nucleic acid testing using a low-cost centrifugal microfluidic platform with bead-based signal enhancement and smartphone read-out. Lab on a Chip, 21(15): 2932-2944 DOI:10.1039/D1LC00266J |
SONG F C, KUEHL J V, CHANDRAN A, et al, 2021. A simple, cost-effective, and automation-friendly direct PCR approach for bacterial community analysis. mSystems, 6(5): e0022421 DOI:10.1128/mSystems.00224-21 |
STILLER M, FULTON T L, 2012. Multiplex PCR amplification of ancient DNA [M] // SHAPIRO B, HOFREITER M. Ancient DNA: Methods and Protocols. Totowa: Humana Press: 133-141.
|
SUN Z B, HUANG Y, WANG Y Z, et al, 2014. Potassium hydroxide-ethylene diamine tetraacetic acid method for the rapid preparation of small-scale PCR template DNA from actinobacteria. Molecular Genetics, Microbiology and Virology, 29(1): 42-46 DOI:10.3103/S089141681401008X |
TABERLET P, COISSAC E, POMPANON F, et al, 2007. Power and limitations of the chloroplast trnL (UAA) intron for plant DNA barcoding. Nucleic Acids Res, 35(3): e14 DOI:10.1093/nar/gkl938 |
TAMARAPU S, MCKILLIP J L, DRAKE M, 2001. Development of a multiplex polymerase chain reaction assay for detection and differentiation of Staphylococcus aureus in dairy products. Journal of Food Protection, 64(5): 664-668 DOI:10.4315/0362-028X-64.5.664 |
TAUBERT F, FISCHER R, GROENEVELD J, et al, 2018. Global patterns of tropical forest fragmentation. Nature, 554(7693): 519-522 DOI:10.1038/nature25508 |
TEMESGEN T T, BARLAAM A, TYSNES K R, et al, 2020. Comparative evaluation of UNEX-based DNA extraction for molecular detection of Cyclospora cayetanensis, Toxoplasma gondii, and Cryptosporidium parvum as contaminants of berries. Food Microbiology, 89: 103447 DOI:10.1016/j.fm.2020.103447 |
THOMPSON K A, NEWMASTER S G, 2014. RETRACTED ARTICLE: molecular taxonomic tools provide more accurate estimates of species richness at less cost than traditional morphology-based taxonomic practices in a vegetation survey. Biodiversity and Conservation, 23(6): 1411-1424 DOI:10.1007/s10531-014-0672-z |
TIAN J J, HUANG K L, LUO Y B, et al, 2018. Visual single cell detection of dual-pathogens based on multiplex super PCR (MS-PCR) and asymmetric tailing HCR (AT-HCR). Sensors and Actuators B: Chemical, 260: 870-876 DOI:10.1016/j.snb.2018.01.017 |
TONOOKA Y, FUJISHIMA M, 2009. Comparison and critical evaluation of PCR-mediated methods to walk along the sequence of genomic DNA. Applied Microbiology and Biotechnology, 85(1): 37-43 DOI:10.1007/s00253-009-2211-5 |
VAN DER LOOS L M, NIJLAND R, 2021. Biases in bulk: DNA metabarcoding of marine communities and the methodology involved. Molecular Ecology, 30(13): 3270-3288 DOI:10.1111/mec.15592 |
VANDENBROUCKE I, VAN MARCK H, VERHASSELT P, et al, 2011. Minor variant detection in amplicons using 454 massive parallel pyrosequencing: experiences and considerations for successful applications. Biotechniques, 51(3): 67-77 DOI:10.2144/000113733 |
VOLDSTAD L H, ALSOS I G, FARNSWORTH W R, et al, 2020. A complete Holocene lake sediment ancient DNA record reveals long-standing high Arctic plant diversity hotspot in northern Svalbard. Quaternary Science Reviews, 234: 106207 DOI:10.1016/j.quascirev.2020.106207 |
WANG P X, CLEMENS S, TADA R, et al, 2019a. Blowing in the monsoon wind. Oceanography, 32(1): 48-59 DOI:10.5670/oceanog.2019.119 |
WANG R H, FAN Z L, ZHANG H Z, et al, 2002. Remote sensing analysis of desert vegetation and its landscape changes: the case in Middle reaches of Tarim River Basin, Xinjiang, China. Science in China Series D: Earth Sciences, 45(1): 54-58 |
WANG P X, JIAN Z M, 2019b. Exploring the deep South China Sea: retrospects and prospects. Science China Earth Sciences, 62(10): 1473-1488 DOI:10.1007/s11430-019-9484-4 |
WANG R J, POLYAK L, XIAO W S, et al, 2018. Late-Middle Quaternary lithostratigraphy and sedimentation patterns on the Alpha Ridge, central Arctic Ocean: implications for Arctic climate variability on orbital time scales. Quaternary Science Reviews, 181: 93-108 DOI:10.1016/j.quascirev.2017.12.006 |
WILLERSLEV E, DAVISON J, MOORA M, et al, 2014. Fifty thousand years of Arctic vegetation and megafaunal diet. Nature, 506(7486): 47-51 DOI:10.1038/nature12921 |
WILLERSLEV E, HANSEN A J, BINLADEN J, et al, 2003. Diverse plant and animal genetic records from Holocene and Pleistocene sediments. Science, 300(5620): 791-795 DOI:10.1126/science.1084114 |
WILLIS K J, KLECZKOWSKI A, CROWHURST S J, 1999. 124 000-year periodicity in terrestrial vegetation change during the Late Pliocene Epoch. Nature, 397(6721): 685-688 DOI:10.1038/17783 |
XIE N G, WANG M X, SONG P, et al, 2022. Designing highly multiplex PCR primer sets with Simulated Annealing Design using Dimer Likelihood Estimation (SADDLE). Nature Communications, 13(1): 1881 DOI:10.1038/s41467-022-29500-4 |
XIONG Z F, LI T G, HÖNISCH B, et al, 2022. Monsoon- and ENSO-driven surface-water pCO2 variation in the tropical West Pacific since the Last Glacial Maximum. Quaternary Science Reviews, 289: 107621 DOI:10.1016/j.quascirev.2022.107621 |
YANG Z B, LEI Y L, ROSENTHAL Y, et al, 2021. Late Pleistocene climate induced changes in paleo-vegetation in Borneo: possible implications to human divergence. Quaternary Science Reviews, 267: 107109 DOI:10.1016/j.quascirev.2021.107109 |
YANG Z B, LI T G, LEI Y L, et al, 2020b. Vegetation evolution-based hydrological climate history since LGM in southern South China Sea. Marine Micropaleontology, 156: 101837 DOI:10.1016/j.marmicro.2020.101837 |
YAZDI S M H T, KIAH H M, GABRYS R, et al, 2018. Mutually uncorrelated primers for DNA-based data storage. IEEE Transactions on Information Theory, 64(9): 6283-6296 DOI:10.1109/TIT.2018.2792488 |
ZENG L L, WARDLOW B D, XIANG D X, et al, 2020. A review of vegetation phenological metrics extraction using time-series, multispectral satellite data. Remote Sensing of Environment, 237: 111511 DOI:10.1016/j.rse.2019.111511 |
ZHANG L S, BECQUET V, LI S H, et al, 2003. Optimization of multiplex PCR and multiplex gel electrophoresis in sunflower SSR analysis using infrared fluorescence and tailed primers. Acta Botanica Sinica, 45(11): 1312-1318 DOI:10.3321/j.issn:1672-9072.2003.11.009 |
ZHANG D, LI W, ZHANG S, et al, 2011. Evaluation of the impact of DNA extraction methods on BAC bacterial community composition measured by denaturing gradient gel electrophoresis. Letters in Applied Microbiology, 53(1): 44-49 DOI:10.1111/j.1472-765X.2011.03064.x |
ZHANG T L, WANG R J, XIAO W S, et al, 2021. Characteristics of terrigenous components of Amerasian Arctic Ocean surface sediments: implications for reconstructing provenance and transport modes. Marine Geology, 437: 106497 DOI:10.1016/j.margeo.2021.106497 |
ZHOU T C, SHI X F, HUANG M, et al, 2021. Genesis of REY-rich deep-sea sediments in the Tiki Basin, eastern South Pacific Ocean: evidence from geochemistry, mineralogy and isotope systematics. Ore Geology Reviews, 138: 104330 DOI:10.1016/j.oregeorev.2021.104330 |
 2023, Vol. 54
2023, Vol. 54


