中国海洋湖沼学会主办。
文章信息
- 陈锋, 胡朝阳, 孙雪, 徐年军. 2023.
- CHEN Feng, HU Chao-Yang, SUN Xue, XU Nian-Jun. 2023.
- microRNA介导雨生红球藻在高光胁迫下次级细胞壁形成和虾青素积累的分子机制
- THE MECHANISM UNDERLYING MICRORNA-MEDIATED SECONDARY CELL WALL FORMATION AND ASTAXANTHIN ACCUMULATION UNDER HIGH LIGHT STRESS IN HAEMATOCOCCUS PLUVIALIS
- 海洋与湖沼, 54(3): 763-772
- Oceanologia et Limnologia Sinica, 54(3): 763-772.
- http://dx.doi.org/10.11693/hyhz20220800205
文章历史
-
收稿日期:2022-08-01
收修改稿日期:2022-10-03
2. 浙江省海洋生物工程重点实验室 浙江宁波 315211
2. Key Laboratory of Marine Biotechnology of Zhejiang Province, Ningbo 315211, China
虾青素(3,3'-二羟基-β,β-1-胡萝卜素-4,4'-二酮)是一种天然存在的类胡萝卜素(Gong et al, 2016)。虾青素的基本结构由一个40碳不饱和骨架组成, 并由一个基本的异戊二烯结构进行生物合成(Ambati et al, 2014)。虾青素最先以赋予鲑科鱼类(鲑鱼和鳟鱼)、虾、龙虾粉红色肉质而闻名, 在它们的生长和繁殖过程中发挥着重要作用, 因此被广泛应用于水产养殖(Lim et al, 2018)。从营养的角度来看, 虾青素被认为是自然界中最强大的抗氧化剂, 抗氧化能力是维生素E的550倍。多项研究表明, 虾青素不仅可以有效地调节人体免疫力, 还可以预防肿瘤和心血管疾病, 治疗、缓解阿尔茨海默病和帕金森病相关的神经损伤, 因此在水产养殖、化妆品和医药等领域得到了广泛的应用, 具有巨大的市场价值(Miyawaki et al, 2008; Rao et al, 2013; Galasso et al, 2018)。
雨生红球藻(Haematococcus pluvialis)是一种单细胞绿藻, 在高光、氮饥饿和高盐等非生物胁迫下, 可以积累其干重4%以上的虾青素, 是目前自然界中最优的虾青素生产者(Fábregas et al, 2003)。胁迫条件会引发雨生红球藻细胞内活性氧的暴发, 它在调节雨生红球藻类胡萝卜素合成和虾青素积累中起着关键作用, 其中, 高光是诱导虾青素积累的最有效的条件(Hu et al, 2020)。雨生红球藻有着复杂的生活史, 根据形态和生理特征可以分为游动细胞和不动细胞(Zhang et al, 2019)。在适宜的条件下, 藻细胞呈绿色游动状态, 通过细胞增殖来增加生物量。在胁迫条件下, 藻细胞的增殖受到抑制, 其游动细胞的鞭毛开始脱落进而转变成不动细胞, 同时伴随着次级细胞壁的形成以及虾青素的大量积累(Hu et al, 2019)。因此, 利用雨生红球藻进行虾青素的生产需要两个阶段, 即在低光下积累生物量, 在高光下积累虾青素。然而, 在虾青素积累阶段, 藻细胞的厚壁结构导致下游加工的高能耗以及较低的虾青素提取效率, 这些极大地限制了雨生红球藻虾青素的商业化生产(Yu et al, 2021)。因此, 了解次级细胞壁和虾青素生物合成的调控机制对雨生红球藻虾青素产业具有重要意义。
MicroRNA (miRNA)是一类长度为18~30 nt的非编码小RNA (small RNA, sRNA), 通过互补mRNA调节基因的表达(Chen et al, 2011)。miRNA作为一种重要的转录后调控因子, 具有调节植物生长发育和响应环境胁迫的功能。近些年来, 研究人员利用高通量miRNA测序技术, 在高等植物和微藻中鉴定出大量保守和新的miRNA, 并对这些miRNA的靶基因进行了功能预测。例如, 在拟南芥(Arabidopsis)中发现miR319参与调控了花瓣、叶片的发育以及种子中油脂的积累(Todesco et al, 2010; Wang et al, 2020), 在棉花(Gossypium hirsutum)中miR160调控花药的发育和棉纤维伸长相关的miRNA (Liu et al, 2019)。在微藻中, 最早和最全面的miRNA研究是在莱茵衣藻(Chlamydomonas reinhardtii)中(Molnár et al, 2007; Lou et al, 2018), 在盐生杜氏藻(Dunaliella salina)中发现了111个响应高光胁迫的miRNAs(Lou et al, 2020)。此外, 只有一篇文献对雨生红球藻的miRNA进行了报道, 该研究发现了115个响应高光和醋酸钠胁迫的miRNAs (Wang et al, 2021)。因此, 可以看出目前和雨生红球藻miRNA相关的研究仍然十分匮乏。
在本研究中, 通过BGISEQ-500高通量测序技术, 我们鉴定了雨生红球藻在高光胁迫下从绿色游动细胞转变为红色不动细胞这一过程中差异表达的miRNA, 并对其表达模式和靶基因进行了分析和预测。结合我们之前的转录组研究, 阐明了这些差异表达的miRNA和其靶基因在高光胁迫诱导雨生红球藻次级细胞壁形成和虾青素合成过程中的调控作用。本研究不仅为理解雨生红球藻次级细胞壁形成和虾青素生物合成的潜在分子机制提供了新的思路, 也为深入开展雨生红球藻中miRNA的功能研究打下了基础。
1 材料与方法 1.1 雨生红球藻的培养和处理实验用雨生红球藻来自宁波大学藻种库, 藻株编号为NBU489。雨生红球藻采用两步法培养: 在第一阶段, 绿色藻细胞在含有150 mL无菌NBU3#培养基的250 mL锥形瓶中培养, 细胞初始接种量为0.25×105 cells/mL, 培养温度为24 ℃, 光强为25 μmol photon/(m2 s), 光暗周期为12 h: 12 h。第二阶段, 取60 mL培养至平台期的雨生红球藻藻液转移至新的100 mL锥形瓶中进行连续高光照强度培养, 光强为140~150 μmol photon/(m2 s)。每升NBU3#培养基由以下成分组成(单位: mg): NaNO3, 100; Na2EDTA, 20; K2HPO4, 10; FeSO4·7H2O, 2.5; MnSO4, 0.25; VB12, 5×10–4; VB1, 0.012 (Hu et al, 2020)。
分别在高光培养0 d (HL_0D)、1 d (HL_1D)和5 d (HL_5D)离心收集藻体, 将藻体沉淀先在液氮中冷冻后在–80 ℃保存, 每组样品设置三个生物学重复。
1.2 总RNA的提取与测序数据质控使用Trizol试剂(上海生工)从雨生红球藻样品中提取总RNA, 检测合格的RNA样品送至华大基因进行miRNA测序, 测序平台为BGISEQ-500。对测序所得原始序列进行去接头、去除低质量序列等数据过滤过程得到可信的有效标签序列, 并对其序列的数目和长度分布进行统计。
1.3 miRNA的鉴定与靶基因预测利用比对软件AASRA将有效标签序列比对到雨生红球藻mRNA数据库和其他sRNA数据库(Tang et al, 2021), 雨生红球藻小RNA按照miRbase (https://www.mirbase.org/) > pirnabank (http://pirnabank.ibab.ac.in/) > snoRNA (http://www.snorna.biotoul.fr/) > Rfam (http://rfam.xfam.org/)的顺序进行注释。基于miRNA的前体能够形成发夹二级结构, 我们使用miRA软件进行新的miRNA预测(Evers et al, 2015), 对得到的miRNA使用TPM标准化来进行表达水平的定量(Zhao et al, 2020)。使用psRobot (Wu et al, 2012)和TargetFinder软件(Fahlgren et al, 2010)对得到的miRNA进行靶基因的预测, 结合自由能、得分值等条件对靶基因进行过滤, 最终取两个软件的交集作为最终的靶基因预测结果。
1.4 差异表达miRNA及其GO和KEGG富集分析采用DESeq方法进行负二项分布的差异表达分析(Wang et al, 2010)。以Q值< 0.001和差异倍数的绝对值> 2作为两个阈值筛选不同对照组的差异表达miRNA (Differentially expressed miRNA, DEmiRNA), 根据DEmiRNA筛选结果, 使用R软件中的pheatmap函数对其进行层次聚类分析。
利用KEGG数据库和GO数据库对DEmiRNA对应的靶基因进行KEGG通路和GO富集分析(He et al, 2018), 来研究miRNA调控次级细胞壁合成和虾青素生物合成的分子机制。
2 结果与分析 2.1 sRNA-seq的数据质控和分析我们对高光处理0 d (HL_0D)、1 d (HL_1D)和5 d (HL_5D)的雨生红球藻样品构建了9个sRNA文库, 用于高通量测序, 样品和之前用于转录组分析的样品是一致的(Hu et al, 2019)。如表 1所示, HL_0D、HL_1D和HL_5D三组雨生红球藻样品测序共产生了2.5×108条原始数据, 去除低质量数据后, 从三个时间点的样品中分别得到了25 594 806、25 049 571和23 165 827条有效标签序列, 这些干净序列比对上参考基因组的平均比对率分别达到86.87%、87.14%和88.35%。上述9个样品的Q20值均超过98.6%, 说明本研究中sRNA-seq数据质量良好, 可满足后续分析需要。
| 样品 | 原始读段 | 有效标签序列 | 比对率/% | Q20值 |
| HL_0D_1 | 27 784 020 | 25 575 038 | 88.76 | 99.0 |
| HL_0D_2 | 29 896 729 | 28 339 719 | 83.97 | 98.8 |
| HL_0D_3 | 29 845 259 | 28 376 865 | 87.87 | 99.0 |
| HL_1D_1 | 28 571 881 | 27 147 825 | 86.65 | 98.7 |
| HL_1D_2 | 27 846 889 | 26 413 804 | 86.89 | 98.8 |
| HL_1D_3 | 30 123 818 | 28 756 826 | 87.06 | 98.7 |
| HL_5D_1 | 20 150 462 | 19 058 074 | 85.46 | 98.6 |
| HL_5D_2 | 29 241 738 | 27 225 145 | 89.43 | 98.7 |
| HL_5D_3 | 28 342 432 | 26 397 593 | 90.17 | 98.8 |
我们对测序所得的高光胁迫下雨生红球藻的miRNA序列与miRBase、snoRNA、Rfam、siRNA等非编码数据库进行比对, 共鉴定出342个已知miRNA, 其中在高光0 d、1 d和5 d处理组中分别鉴定到310、188和188个已知miRNA, 这些miRNA序列与植物中已知的miRNA序列高度一致。基于miRNA的前体能够形成发夹二级结构这一原理, 利用miRA软件(Evers et al, 2015)预测出283个新miRNA, 其中199个位于DNA的正义链上, 84个位于DNA的负义链上, 这些新miRNA的前体都具有典型的茎环发夹二级结构。
植物中miRNA序列的长度主要集中在21 nt, 上述625个miRNA的长度分布介于18~30 nt之间, 长度为21 nt (206个)的miRNA数量最多, 与莱茵衣藻(Chlamydomonas reinhardtii)、玉米(Zea mays L)类似(Molnár et al, 2007; 王琪月等, 2018), 其次是30 nt (85个)和22 nt (78个), 其中, 已知的miRNA的长度分布在18~24 nt, 长度为25~30 nt的miRNA全部为预测的新miRNA, 这些miRNA序列长度分布范围与Dicer酶切割的特征一致(Wang et al, 2021)。此外, 19~23 nt的miRNA的首位碱基分布偏向为U, 而25~ 30 nt的miRNA偏向C, 24 nt的miRNA偏向A (图 1)。
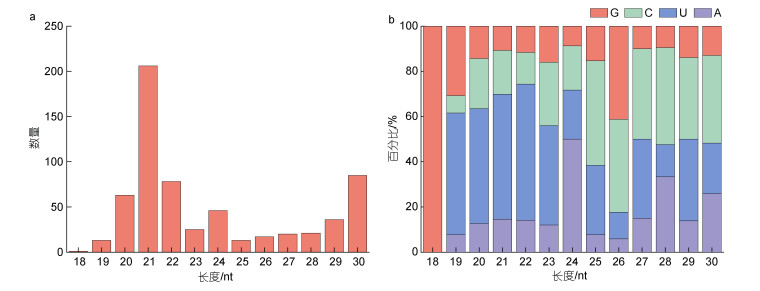 |
| 图 1 雨生红球藻在高光胁迫下的miRNA长度分布(a)和首位碱基偏好(b) Fig. 1 The miRNA length distribution (a) and the first nucleotide bias (b) in H. pluvialis under high light stress 注: G: 鸟嘌呤; C: 胞嘧啶; U: 尿嘧啶; A: 腺嘌呤 |
为了研究雨生红球藻在高光胁迫下miRNA的响应变化, 根据Q < 0.001和|变化倍数|≥2条件对DEmiRNA进行了筛选(图 2)。在HL_0D vs HL_1D、HL_0D vs HL_5D和HL_1D vs HL_5D这三个比较组中分别发现了123、172和78个DEmiRNA, 其中显著上调的miRNA分别为15、48和54个, 显著下调的miRNA分别为108、124和24个(图 2a)。与其他两组不同, HL_1D vs HL_5D组的DEmiRNA数量最少, 且上调的DEmiRNA多于下调的DEmiRNA。对三个比较组的DEmiRNA进行差异倍数聚类分析发现, miRNA在雨生红球藻响应高光胁迫过程中呈动态表达, HL_0D vs HL_1D和其他两组被聚类在不同分支上, 这些结果说明雨生红球藻在高光1 d时的初始miRNA表达模式与其他时间点有着较大差异(图 2b)。
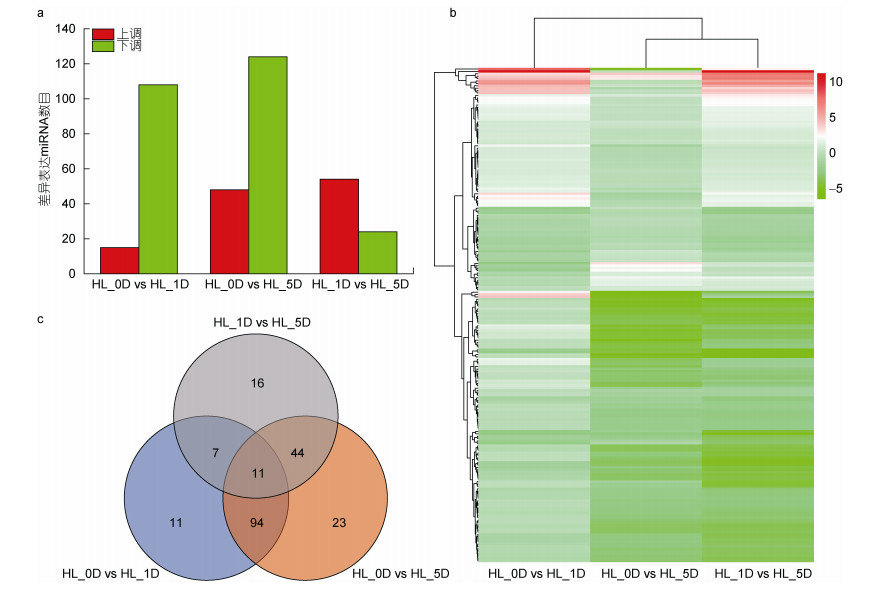 |
| 图 2 各个高光比较组中miRNA的差异表达分析 Fig. 2 Differential expression analysis of miRNAs in each comparison group 注: a. 各个比较组中差异表达的miRNA; b. 各个比较组中miRNA表达水平的热图分析; c. 各个比较组中miRNA的维恩分析 |
从维恩图(图 2c)中可见, 分别有11个、23个和16个miRNA在三个比较组中特异性表达, 有11个miRNA在三组中均具有显著差异。对这11个DEmiRNA进一步分析发现, 和高光第0天相比, novel_mir51、novel_mir253、novel_mir187、novel_ mir143这四个miRNA在第1天和第5天都是显著上调的, novel_mir106和miR319a_1这两个miRNA在第1天和第5天都是显著下调的, 而miR171b-3p、novel_mir259、novel_mir260、novel_mir66和novel_ mir29这5个miRNA在高光第1天时显著下调的而在第5天时又显著上调了。miR319a是一类在植物叶片发育和抗逆有着重要调控作用的miRNA (Bresso et al, 2018), 最新的研究表明, 在拟南芥中过表达miR319a会抑制脂肪酸的积累(Wang et al, 2020)。雨生红球藻在高光胁迫下会快速积累大量油脂, 用于储存虾青素酯和抵抗逆境, 与第0天相比, miR319a的表达在高光1 d和5 d时是一直显著下调的, 这说明雨生红球藻在高光胁迫下可能通过下调miR319a_1的表达量来减弱对油脂合成相关的基因的抑制, 从而促进了油脂的生物合成, 来缓解高光胁迫对藻细胞的伤害。miR171b-3p也是一种在植物中和代谢过程相关的miRNA, 在乌龙茶(Camellia sinensis)中它通过抑制类黄酮物质的生物合成, 从而增强了萜类化合物的生物合成(Zhu et al, 2020)。萜类化合物是虾青素生物合成的前体, 这类物质的含量变化与雨生红球藻中虾青素的合成密切相关, 该miRNA在雨生红球藻响应高光胁迫过程中表现出先下调后上调的趋势, 说明它可能参与调控了和虾青素合成相关前体萜类物质的代谢过程。这些结果表明, 雨生红球藻在高光胁迫下的miRNA有着高度阶段性或时期特异性表达的特点。此外, 上述11个在三个时间段均显著差异表达miRNA中, 有9个为新鉴定出的miRNA, 数量远大于已知miRNA (2个), 推测这些差异表达的新miRNA在雨生红球藻响应高光胁迫过程中可能发挥了重要作用, 值得进一步研究, 这些新miRNA的序列信息如表 2所示。
| miRNA ID | miRNA序列 | TPM值 | ||
| HL_0D | HL_1D | HL_5D | ||
| novel_mir29 | GTGAGGTTCATTGGAGGCAGGGTGGGGCCA | 4.41 | 1.67 | 15.50 |
| novel_mir51 | AATGGCAAAGCGCTTCTCGGTCCACTGGCC | 1.41 | 16.52 | 234.19 |
| novel_mir66 | TGGACAGTTAGGTCCGCGT | 38.17 | 13.04 | 95.61 |
| novel_mir72 | CAAACCTTGTCCGATTGGGCTG | 1 035.28 | 628.39 | 477.28 |
| novel_mir106 | TGACACAGCAATCTGAAGGAGG | 15.92 | 5.23 | 1.58 |
| novel_mir147 | CGCGTACCTCCGACACTCAACTGACCGCCG | 1 923.06 | 2 709.80 | 4 133.34 |
| novel_mir155 | GTTGGTGTCAGAGAGGTAGCGGGCTATTGC | 430.67 | 411.33 | 238.29 |
| novel_mir119 | CGCCGTCAGAGTCCAGGGTGTA | 5 966.52 | 3 606.68 | 2345.90 |
| novel_mir121 | TCAGGGCGATCATTGGGACACGACCTCAC | 5 157.95 | 5 190.84 | 4313.60 |
| novel_mir143 | CAAGGCGGCAGCGGTCAGGCCGG | 1.03 | 5.31 | 105.62 |
| novel_mir187 | CTGCCGGTCATCAAAGTCCCGGGTG | 34.20 | 57.07 | 260.42 |
| novel_mir253 | CCAGCAGGGCATGGGCGTGTTTGCAGCC | 2.22 | 11.60 | 345.27 7 |
| novel_mir259 | CCGACCTTGCCTTCCTTTGTGC | 0.40 | 0.000 1 | 2.31 |
| novel_mir260 | GTGTGGTGAAACTCCGGTGCGGCGACG | 9.93 | 2.55 | 37.49 |
此外, DEmiRNA的表达谱结果显示, 在三个时间点下表达量前三的均为新miRNA, 高光处理0 d时表达量最高的是novel_mir119 (TPM=5 966.52)、novel_ mir147 (TPM=1 923.06)、novel_mir72 (1 035.28); 高光处理1 d时表达量最高的是novel_mir121 (TPM=5 190.84)、novel_mir119 (TPM=3 606.68)、novel_mir155 (411.33); 高光处理5 d时表达量最高的是novel_ mir121 (TPM=5 190.84)、novel_mir147 (TPM=4 133.33)、novel_mir119 (2 345.90)。这些高表达的新miRNA的序列信息也列在表 2。
2.4 DEmiRNA的靶基因预测及功能分析由于miRNA通过结合下游靶基因来发挥其生物学功能, 因此我们利用TargetFinder和psRobot软件对雨生红球藻在高光胁迫下的具有显著差异的miRNA进行了靶基因预测。结果显示, HL_0D vs HL_1D、HL_0D vs HL_5D、HL_1D vs HL_5D三个比较组共有274个DEmiRNA对应3018个靶基因。不同miRNA的靶基因数目差距很大, 大部分miRNA都有靶向两个以上基因, 靶基因数量最多的是miR160a- 5p, 有58个靶基因, 而miR482a_1、miR159a_ 1和novel_mir106等35个miRNA都仅有1个靶基因。研究表明, 与miR160a-5p高度同源的miR160a_A05参与调控了棉花中棉纤维的合成(Liu et al, 2019)。在miR160a-5p的靶基因中发现它可以靶向内切葡聚糖酶(纤维素酶的一种)基因, 推测miR160a-5p也可能参与了雨生红球藻中纤维素的代谢。
为了进一步研究高光胁迫下雨生红球藻DEmiRNA的功能, 对上述三组DEmiRNA的靶基因分别进行GO注释分类分析。GO显著性富集结果所图 3所示, HL_0D vs HL_1D、HL_0D vs HL_5D、HL_1D vs HL_5D比较组分别注释到38、41和36个GO条目, 包括生物过程, 细胞组分和分子功能三大类。在HL_0D vs HL_1D组中分别有447、755、363条靶基因被富集到生物学过程、细胞组分和分子功能, 其中富集靶基因数最多的是催化活性(165个); 在HL_0D vs HL_5D组中分别有802、1 015、508条靶基因被富集到生物学过程、细胞组分和分子功能, 其中富集靶基因数最多的也是催化活性(223个); 在HL_1D vs HL_5D组中分别有454、455、244条靶基因被富集到生物学过程、细胞组分和分子功能, 其中富集靶基因数最多的是结合功能(121个)。
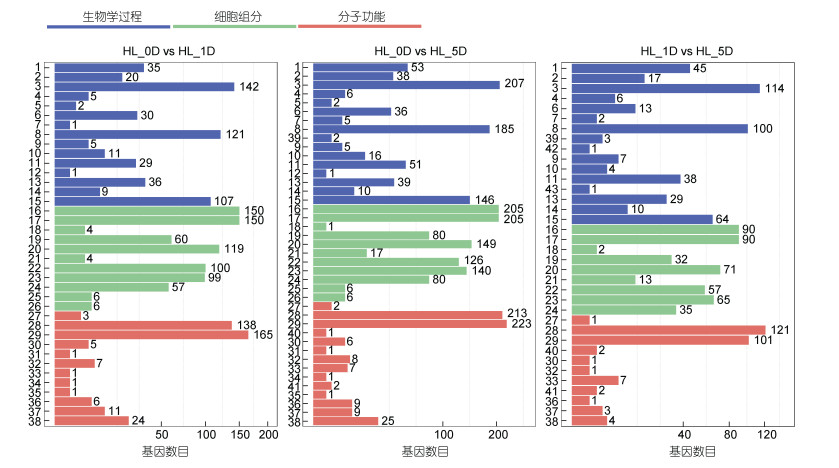 |
| 图 3 各个比较组中DEmiRNA靶基因的GO数据库注释 Fig. 3 The GO database annotation of DEmiRNA's target genes in each comparison group 注: 1. 生物调控; 2. 细胞组织部分或生物合成; 3. 细胞过程; 4. 发育过程; 5. 生长; 6. 定位; 7. 运动; 8. 生物代谢进程; 9. 生物过程的负调控; 10. 生物过程的正调控; 11. 生物过程调控; 12. 繁殖; 13. 对刺激的反应; 14. 信号传导; 15. 单一机体过程; 16. 细胞; 17. 细胞部分; 18. 细胞外组分; 19. 大分子复合物; 20. 膜结构; 21. 膜关闭内腔; 22. 膜部分; 23. 细胞器; 24. 细胞器部分; 25. 病毒粒子; 26. 病毒粒子部分; 27. 抗氧化活性; 28. 结合功能; 29. 催化活性; 30. 酶调节活性; 31. 鸟苷-核苷酸交换因子活性; 32. 分子转导活性; 33. 核酸结合转录因子活性; 34. 营养存储库活性; 35. 蛋白标签; 36. 受体活性; 37. 结构分子活性; 38. 转运活性; 39. 多细胞生物过程; 40. 电子载体活性; 41. 蛋白质结合转录因子活性; 42. 有机体进程; 43. 生殖过程 |
KEGG注释分类结果显示, 共有1 381条靶基因被注释到20个二级KEGG通路, 其中被注释到靶基因数量最多的是代谢一级通路, 共有736个(53.3%)靶基因。结果如图 4所示, 在代谢一级通路中靶基因数量前三的通路分别为全局和总览图、其他氨基酸的代谢和碳水化合物代谢, 其中碳水化合物代谢通路中的部分靶基因被富集到淀粉与蔗糖代谢、果糖与甘露糖代谢、糖酵解和TCA循环这四个通路, 这些靶基因可能调控了雨生红球藻次级细胞壁的形成和虾青素碳骨架的合成。此外, 萜类和聚酮类化合物代谢中的部分靶基因被富集到萜类骨架生物合成通路, 可能参与调控了虾青素前体物质的生物合成(Du et al, 2021a)。脂质代谢通路中的相关靶基因可能会参与到油脂和脂肪酸的合成, 雨生红球藻在高光胁迫下, 虾青素和油脂是同步积累的并且虾青素经过酯化反应后储存在三酰甘油中(Du et al, 2021b)。
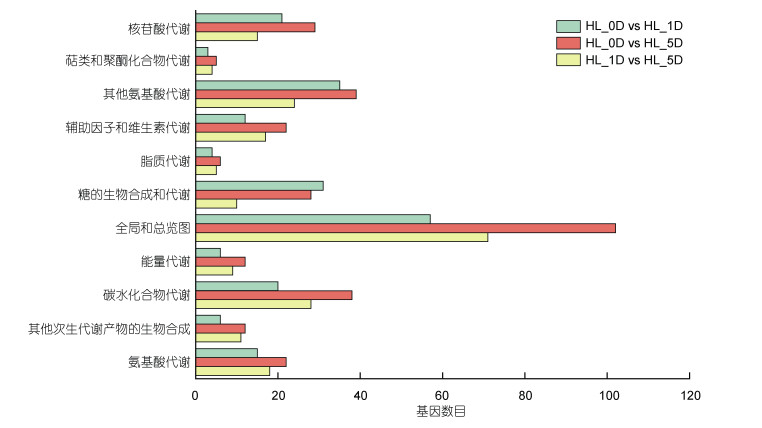 |
| 图 4 各个比较组中DEmiRNA靶基因的KEGG数据库注释 Fig. 4 The KEGG database annotation of DEmiRNA's target genes in each comparison group |
根据靶基因的KEGG数据库注释信息和前期研究中的转录组结果, 我们针对性分析了雨生红球藻中次级细胞壁生物合成以及虾青素生物合成这两个生理过程中的DEmiRNA和靶基因之间的调控作用。本文主要找到4条和这两个生理过程有关的KEGG通路, 包括蔗糖和淀粉代谢、果糖和甘露糖代谢、糖酵解、萜类骨架合成。这4条通路中共涉及13个DEmiRNA, 其中8个DEmiRNA结合9个蔗糖和淀粉代谢通路相关靶基因, 2个DEmiRNA结合2个果糖和甘露糖代谢通路相关靶基因, 5个DEmiRNA结合5个糖酵解通路相关靶基因, 2个DEmiRNA结合2个萜类骨架生物合成通路相关靶基因。
甘露糖和纤维素是雨生红球藻红色不动细胞次级细胞壁的主要组成成分, 其中甘露糖占所有细胞壁糖类的80% (Lin et al, 2021)。对于植物来说, GDP-甘露糖是主要的核苷酸糖之一, 它不仅参与调控细胞壁的形成, 还为甘露糖的生物合成提供底物(Figueroa et al, 2021)。DEmiRNA和转录组结果(图 5a)共同显示, 与第0天相比, 高光处理组中和次级细胞壁合成相关通路上的葡萄糖磷酸变位酶(PGM)、内切葡聚糖酶(EG)和甘露聚糖内切-1,4-β-甘露糖苷酶(MAN)基因的表达均显著上调。其中, novel_mir167结合两个靶基因调控了PGM基因的表达, miR160a- 5p、novel_mir146、miR172b-5p_5和novel_mir281这4个miRNA分别结合对应的靶基因共同调控了EG基因的表达, novel_mir66结合靶基因调控了MAN基因的表达。在雨生红球藻次生细胞壁的生物合成过程中, 纤维素合成酶和纤维素酶基因的表达都会明显上调, 前者可以催化纤维素的生物合成, 为细胞壁提供刚性结构, 后者可以水解纤维素, 为GDP-甘露糖的生物合成提供原料(Guo et al, 2021)。因此, 在高光条件下, 上述纤维素合成和代谢相关的miRNA可能通过上调次级细胞壁底物合成相关靶基因的表达水平, 来调控雨生红球藻次级细胞壁的生物合成。值得一提的是, 在4个调控内切葡聚糖酶的miRNA中, miR160a-5p和miR172b-5p_5的在高光下的表达显著下调, 而novel_mir146和novel_mir281的表达显著上调, 调控甘露聚糖内切-1, 4-β-甘露糖苷酶的novel_ mir66在高光第1天的表达显著下调, 但是在第5天时的表达显著上调, 虽然这些miRNA的表达模式不同, 但是最终都上调了靶基因的表达。这些结果不仅说明了这5个miRNA在雨生红球藻次级细胞壁形成中存在着重要调控作用, 还体现了miRNA和mRNA相互作用的复杂性。
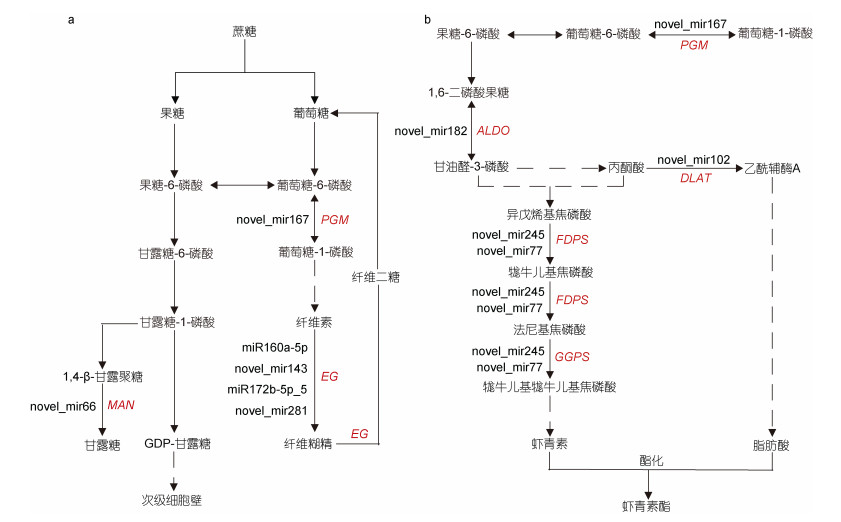 |
| 图 5 雨生红球藻高光下DEmiRNA-mRNA在次级细胞壁形成(a)和虾青素生物合成(b)的调控作用 Fig. 5 The regulatory role of DEmiRNA-mRNA on secondary cell wall formation (a) and astaxanthin biosynthesis (b) under high light in H. pluvialis |
糖酵解是生物体中一个基本的代谢途径, 该过程将葡萄糖分解成2个三碳代谢物并释放能量(Yu et al, 2022)。淀粉和蔗糖代谢途径中产生的6-磷酸葡萄糖和6-磷酸果糖大部分直接进入糖酵解途径, 部分进入磷酸戊糖代谢途径, 这两个代谢途径都为虾青素的合成提供前体和能量(Du et al, 2021a)。DEmiRNA和转录组结果共同显示(图 5b), 糖酵解途径中的果糖-1, 6-二磷酸醛缩酶基因(ALDO)和丙酮酸脱氢酶E2组分基因(DLAT)在高光处理下是显著上调的, 这两个基因分别是novel_mir182和novel_mir102的靶基因。ALDO基因负责催化裂解1, 6-二磷酸-果糖生成甘油醛-3磷酸, 随后和丙酮酸进入萜类骨架生物合成途径形成虾青素前体物质异戊烯焦磷酸(isopentenyl pyrophosphate, IPP), 在萜类骨架生物合成途径中, novel_mir245和novel_mir77共同上调了该途径中法尼基焦磷酸合酶基因(FDPS)和关键酶牻牛儿基牻牛儿基焦磷酸合酶基因(GGPS)的表达, 为虾青素的合成提供了更多的萜类骨架。丙酮酸也可以通过丙酮酸脱氢酶复合体转化为乙酰CoA, 该复合体主要由丙酮酸脱氢酶(E1, EC 1.2.4.1)、二氢硫辛酸转乙酰化酶(E2, EC 2.3.1.12)和二氢硫辛酸脱氢酶(E3, EC 1.8.1.4)组成, 乙酰CoA是脂肪酸生物合成的底物(Gwak et al, 2014), 研究表明雨生红球藻和小球藻(Chlorella zofingiensis)中乙酰CoA含量还和虾青素的生物合成具有很强的相关性(Zhang et al, 2016)。这些结果表明, novel_mir182、novel_mir102、novel_mir245和novel_mir77可能通过调控糖酵解和萜类骨架生物合成途径来增加虾青素和脂肪酸生物合成的底物, 从而促进了雨生红球藻在高光下虾青素的合成。上述雨生红球藻在高光下次级细胞壁形成和虾青素生物合成过程中起重要调控作用的DEmiRNA的详细信息如表 3所示。
| miRNA ID | miRNA序列 | 靶基因 |
| novel_mir167 | AGGTGTAGGCCATGCATGCGT | 葡萄糖磷酸变位酶 |
| miR160a-5p | TGCCTGGCTCCCTGTATGCCA | 内切葡聚糖酶 |
| novel_mir143 | CAAGGCGGCAGCGGTCAGGCCGG | 内切葡聚糖酶 |
| miR172b-5p_5 | GCAGCACCATTAAGATTCAC | 内切葡聚糖酶 |
| novel_mir281 | CACCTTGGCCAGCCAGATCTT | 内切葡聚糖酶 |
| novel_mir66 | TGGACAGTTAGGTCCGCGT | 甘露聚糖内切-1, 4-β-甘露糖苷酶 |
| novel_mir182 | CCATGGTGACGCCAGGACCCTG | 果糖-二磷酸醛缩酶 |
| novel_mir102 | AACCATGCGAGCCTTGATGTTGTGGTCAGC | 丙酮酸脱氢酶E2组分 |
| novel_mir245 | GCGTTGGTAAACGTTGCCATGGTCGTCAT | 法尼基焦磷酸合成酶/牻牛儿基牻牛儿基焦磷酸合成酶 |
| novel_mir77 | ATCGGACAAGGTTGGCCGAAGG | 法尼基焦磷酸合成酶/牻牛儿基牻牛儿基焦磷酸合成酶 |
从高光胁迫下的雨生红球藻的sRNA高通量测序结果中, 我们共鉴定出342个已知miRNA和283个新miRNA。对这些miRNA进行了差异表达分析和靶基因预测, 并结合转录组结果, 本文揭示了高光下雨生红球藻次级细胞壁和虾青素合成相关的DEmiRNA及其靶基因的潜在调控关系。结果发现包括miR160-5p、novel_mir143、novel_mir102、novel_ mir245在内的10个miNRA参与了高光胁迫下雨生红球藻次级细胞壁的形成和虾青素的生物合成。本研究不仅丰富了雨生红球藻miRNA相关的信息, 还从miRNA层面增加了高光下雨生红球藻次级细胞壁形成和虾青素的生物合成机制的新的见解。
王琪月, 孟淑君, 张柯, 等, 2018. 玉米雌穗发育杂种优势相关miRNA的研究. 作物学报, 44(6): 796-813 |
AMBATI R R, PHANG S M, RAVI S, et al, 2014. Astaxanthin: sources, extraction, stability, biological activities and its commercial applications-a review. Marine Drugs, 12(1): 128-152 DOI:10.3390/md12010128 |
BRESSO E G, CHOROSTECKI U, RODRIGUEZ R E, et al, 2018. Spatial control of gene expression by miR319- regulated TCP transcription factors in leaf development. Plant Physiology, 176(2): 1694-1708 DOI:10.1104/pp.17.00823 |
CHEN C J, LIU Q, ZHANG Y C, et al, 2011. Genome-wide discovery and analysis of microRNAs and other small RNAs from rice embryogenic callus. RNA Biology, 8(3): 538-547 DOI:10.4161/rna.8.3.15199 |
DU F C, HU C Y, SUN X, et al, 2021a. Transcriptome analysis reveals pathways responsible for the promoting effect of sucrose on astaxanthin accumulation in Haematococcus pluvialis under high light condition. Aquaculture, 530: 735757 DOI:10.1016/j.aquaculture.2020.735757 |
DU F C, HU C Y, SUN X, et al, 2021b. Transcriptome analysis reveals the promoting effect of trisodium citrate on astaxanthin accumulation in Haematococcus pluvialis under high light condition. Aquaculture, 543: 736978 DOI:10.1016/j.aquaculture.2021.736978 |
EVERS M, HUTTNER M, DUECK A, et al, 2015. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics, 16: 370 DOI:10.1186/s12859-015-0798-3 |
FÁBREGAS J, DOMÍNGUEZ A, MASEDA A, et al, 2003. Interactions between irradiance and nutrient availability during astaxanthin accumulation and degradation in Haematococcus pluvialis. Applied Microbiology and Biotechnology, 61(5): 545-551 |
FAHLGREN N, CARRINGTON J C, 2010. miRNA target prediction in plants [M] // MEYERS B C, GREEN P J. Plant MicroRNAs. New York: Humana Press: 51-57.
|
FIGUEROA C M, LUNN J E, IGLESIAS A A, 2021. Nucleotide-sugar metabolism in plants: the legacy of Luis F. Leloir. Journal of Experimental Botany, 72(11): 4053-4067 DOI:10.1093/jxb/erab109 |
GALASSO C, OREFICE I, PELLONE P, et al, 2018. On the neuroprotective role of astaxanthin: new perspectives?. Marine Drugs, 16(8): 247 DOI:10.3390/md16080247 |
GONG M Y, BASSI A, 2016. Carotenoids from microalgae: a review of recent developments. Biotechnology Advances, 34(8): 1396-1412 DOI:10.1016/j.biotechadv.2016.10.005 |
GUO C L, MEI R, ANWAR M, et al, 2021. The functionally characterization of putative genes involved in the formation of mannose in the aplanospore cell wall of Haematococcus pluvialis (Volvocales, Chlorophyta). Metabolites, 11(11): 725 DOI:10.3390/metabo11110725 |
GWAK Y, HWANG Y S, WANG B B, et al, 2014. Comparative analyses of lipidomes and transcriptomes reveal a concerted action of multiple defensive systems against photooxidative stress in Haematococcus pluvialis. Journal of Experimental Botany, 65(15): 4317-4334 DOI:10.1093/jxb/eru206 |
HE B X, HOU L L, DONG M M, et al, 2018. Transcriptome analysis in Haematococcus pluvialis: astaxanthin induction by high light with acetate and Fe2+. International Journal of Molecular Sciences, 19(1): 175 DOI:10.3390/ijms19010175 |
HU C Y, CUI D D, SUN X, et al, 2019. Transcriptomic analysis unveils survival strategies of autotrophic Haematococcus pluvialis against high light stress. Aquaculture, 513: 734430 DOI:10.1016/j.aquaculture.2019.734430 |
HU C Y, CUI D D, SUN X, et al, 2020. Primary metabolism is associated with the astaxanthin biosynthesis in the green algae Haematococcus pluvialis under light stress. Algal Research, 46: 101768 DOI:10.1016/j.algal.2019.101768 |
LIM K C, YUSOFF F M, SHARIFF M, et al, 2018. Astaxanthin as feed supplement in aquatic animals. Reviews in Aquaculture, 10(3): 738-773 DOI:10.1111/raq.12200 |
LIN J, YAN H L, ZHAO L, et al, 2021. Interaction between the cell walls of microalgal host and fungal carbohydrate- activate enzymes is essential for the pathogenic parasitism process. Environmental Microbiology, 23(9): 5114-5130 DOI:10.1111/1462-2920.15465 |
LIU G Y, LIU J, PEI W F, et al, 2019. Analysis of the MIR160 gene family and the role of MIR160a_A05 in regulating fiber length in cotton. Planta, 250(6): 2147-2158 DOI:10.1007/s00425-019-03271-7 |
LOU S L, SUN T, LI H, et al, 2018. Mechanisms of microRNA-mediated gene regulation in unicellular model alga Chlamydomonas reinhardtii. Biotechnology for Biofuels, 11: 244 DOI:10.1186/s13068-018-1249-y |
LOU S L, ZHU X L, ZENG Z Y, et al, 2020. Identification of microRNAs response to high light and salinity that involved in beta-carotene accumulation in microalga Dunaliella salina. Algal Research, 48: 101925 DOI:10.1016/j.algal.2020.101925 |
MIYAWAKI H, TAKAHASHI J, TSUKAHARA H, et al, 2008. Effects of astaxanthin on human blood rheology. Journal of Clinical Biochemistry and Nutrition, 43(2): 69-74 DOI:10.3164/jcbn.2008048 |
MOLNÁR A, SCHWACH F, STUDHOLME D J, et al, 2007. miRNAs control gene expression in the single-cell alga Chlamydomonas reinhardtii. Nature, 447(7148): 1126-1129 DOI:10.1038/nature05903 |
RAO A R, SINDHUJA H N, DHARMESH S M, et al, 2013. Effective inhibition of skin cancer, tyrosinase, and antioxidative properties by astaxanthin and astaxanthin esters from the green alga Haematococcus pluvialis. Journal of Agricultural and Food Chemistry, 61(16): 3842-3851 DOI:10.1021/jf304609j |
TANG C, XIE Y M, GUO M, 2021. AASRA: An anchor alignment-based small RNA annotation pipeline. Biology of Reproduction, 105(1): 267-277 DOI:10.1093/biolre/ioab062 |
TODESCO M, RUBIO-SOMOZA I, PAZ-ARES J, et al, 2010. A collection of target mimics for comprehensive analysis of microRNA function in Arabidopsis thaliana. PLoS Genetics, 6(7): e1001031 DOI:10.1371/journal.pgen.1001031 |
WANG L K, FENG Z X, WANG X, et al, 2010. DEGseq: an R package for identifying differentially expressed genes from RNA-seq data. Bioinformatics, 26(1): 136-138 DOI:10.1093/bioinformatics/btp612 |
WANG X D, MIAO X X, CHEN G, et al, 2021. Identification of microRNAs involved in astaxanthin accumulation responding to high light and high sodium acetate (NaAC) stresses in Haematococcus pluvialis. Algal Research, 54: 102179 DOI:10.1016/j.algal.2020.102179 |
WANG F L, ZHENG T, HU Z H, et al, 2020. Overexpression of miR319a altered oil body morphogenesis and lipid content in Arabidopsis seeds. Plant Molecular Biology Reporter, 38(4): 531-537 DOI:10.1007/s11105-020-01217-y |
WU H J, MA Y K, CHEN T, et al, 2012. PsRobot: a web-based plant small RNA meta-analysis toolbox. Nucleic Acids Research, 40(W1): W22-W28 DOI:10.1093/nar/gks554 |
YU C L, WANG H P, QIAO T S, et al, 2021. A fed-batch feeding with succinic acid strategy for astaxanthin and lipid hyper- production in Haematococcus pluviualis. Bioresource Technology, 340: 125648 DOI:10.1016/j.biortech.2021.125648 |
YU W J, ZHANG L T, ZHAO J, et al, 2022. Enhancement of astaxanthin accumulation in Haematococcus pluvialis by exogenous oxaloacetate combined with nitrogen deficiency. Bioresource Technology, 345: 126484 DOI:10.1016/j.biortech.2021.126484 |
ZHANG Z, SUN D Z, MAO X M, et al, 2016. The crosstalk between astaxanthin, fatty acids and reactive oxygen species in heterotrophic Chlorella zofingiensis. Algal Research, 19: 178-183 DOI:10.1016/j.algal.2016.08.015 |
ZHANG C H, ZHANG L T, LIU J G, 2019. Exogenous sodium acetate enhances astaxanthin accumulation and photoprotection in Haematococcus pluvialis at the non-motile stage. Journal of Applied Phycology, 31(2): 1001-1008 DOI:10.1007/s10811-018-1622-z |
ZHAO S R, YE Z, STANTON R, 2020. Misuse of RPKM or TPM normalization when comparing across samples and sequencing protocols. RNA, 26(8): 903-909 DOI:10.1261/rna.074922.120 |
ZHU C, ZHANG S T, ZHOU C Z, et al, 2020. Integrated transcriptome, microRNA, and phytochemical analyses reveal roles of phytohormone signal transduction and ABC transporters in flavor Formation of oolong tea (Camellia sinensis) during solar withering. Journal of Agricultural and Food Chemistry, 68(45): 12749-12767 DOI:10.1021/acs.jafc.0c05750 |
 2023, Vol. 54
2023, Vol. 54


