
中国海洋湖沼学会主办。
文章信息
- 刘焘, 陈冰洁, 史会来, 平洪领, 张涛, 瞿印权, 高天翔. 2023.
- LIU Tao, CHEN Bing-Jie, SHI Hui-Lai, PING Hong-Ling, ZHANG Tao, QU Yin-Quan, GAO Tian-Xiang. 2023.
- 基于基因组survey的横带髭鲷(Hapalogenys analis)微卫星位点筛选与特征分析
- ISOLATION AND ANALYSIS OF HAPALOGENYS ANALIS MICROSATELLITES BY GENOME SURVEY
- 海洋与湖沼, 54(3): 848-855
- Oceanologia et Limnologia Sinica, 54(3): 848-855.
- http://dx.doi.org/10.11693/hyhz20221000281
文章历史
-
收稿日期:2022-10-30
收修改稿日期:2022-12-06
2. 汕头大学长江新闻与传播学院 广东汕头 515063;
3. 浙江省海洋水产研究所 浙江省海水增养殖重点实验室 浙江舟山 316021
2. Cheung Kong School of Journalism and Communication, Shantou University, Shantou 515063, China;
3. Zhejiang Province Key Laboratory of Mariculture and Enhancement, Zhejiang Marine Fisheries Research Institute, Zhoushan 316021, China
横带髭鲷(Hapalogenys analis)属于鲈形目(Perciformes)、石鲈科(Pomadasyidae)、髭鲷属(Hapalogenys), 俗称“十六枚”, 系浅海中下层岩礁性鱼类, 在我国沿海均有分布, 是极具发展潜力的增养殖品种, 也是名贵的海水观赏鱼类(赵盛龙等, 2016)。作为重要的海水经济鱼类, 其野生资源匮乏, 渔获量不大, 在市场上供不应求, 近年来已经开始人工养殖。关于横带髭鲷的基础研究已有一些报道, 包括生理生化(史海东等, 2004)、基础生物学(许文军等, 2002; 姚海富等, 2006; 张涛等, 2020; 平洪领等, 2021)、遗传学(喻子牛等, 1994, 1995; 王世锋等, 2008; 徐田军等, 2010)等方面的研究。迄今尚未见组学方面的研究报道, 这极大地制约了后续横带髭鲷的繁养和保护工作。因此, 为了更好地开展横带髭鲷的增养殖研究, 探究其种质资源情况以及群体的遗传多样性和遗传结构尤为重要。
微卫星分子标记在研究物种种质情况及遗传信息方面有很大优势, 因微卫星DNA具有高度多态性与共显性、数量丰富且易于检测的特点(Tautz, 1989; Madesis et al, 2013), 已被广泛应用到遗传多样性检测(Cheng et al, 2015)和遗传种质鉴定(Lü et al, 2013)等多个领域。横带髭鲷微卫星标记开发未见有相关研究, 截至目前, GenBank登录的横带髭鲷微卫星序列为0条, 其数量严重不足, 亟需开发横带髭鲷微卫星多态性标记。在过去的研究中, 微卫星标记的筛选开发主要依靠构建微卫星富集文库, 其操作复杂、成本高且效率低, 不够便利(Zane et al, 2002)。近年来, 随着高通量测序技术的日渐成熟, 动植物微卫星标记的批量开发拥有了基因组水平的全新开发策略(Csencsics et al, 2010; Capobianchi et al, 2013)。基因组survey测序为非模式生物基因组范围内微卫星位点的检测提供了技术支持, 同时也提供了生物体基因组基础信息, 包括其基因组大小、杂合度水平和重复率等信息的评估。目前, 由于基因组中含有大量的微卫星序列且检出率高, 基于该测序方法开发微卫星位点已经在多种水生生物(Lu et al, 2017; Chen et al, 2020; Xu et al, 2020)中得到应用。
本研究旨在使用基因组survey技术评估横带髭鲷基因组大小, 解析基因组范围内微卫星序列的组成和分布特征, 从中筛选开发具有多态性的微卫星位点, 并利用一个供试群体检测所开发的微卫星位点的适用性。新开发的微卫星位点可应用于今后横带髭鲷遗传育种和群体遗传学的研究。
1 材料与方法 1.1 材料用于基因组survey测序的1尾横带髭鲷样品采自浙江舟山海域。用于多态性微卫星位点分离与鉴定及群体遗传多态度检测的横带髭鲷供试群体为采自浙江舟山海域的野生群体, 共32尾。将肌肉等组织液氮速冻, 放置于–80 ℃超低温冰箱中冷冻保存。
1.2 基因组DNA提取和survey测序使用常规的酚-氯仿方法抽提基因组DNA, 总DNA经过RNase A处理获得高纯度、无RNA污染的DNA。利用超微量分光光度计测定基因组DNA, 由D260/D280和浓度(ng/μL)判定DNA的纯度和浓度。构建350 bp基因组survey测序文库, 采用Illumina HiSeq 2000高通量测序仪进行双末端测序, 由武汉古奥基因科技有限公司完成。
1.3 微卫星序列的获取将测序所得clean data进行基因组de nove组装并获得拼接序列, 使用微卫星识别工具MISA软件(Beier et al, 2017)搜索微卫星位点, 筛选参数设置为: 筛选单核苷酸(mononucleotide)、二核苷酸(dinucleotide)、三核苷酸(trinucleotide)、四核苷酸(tetranucleotide)、五核苷酸(pentanucleotide)和六核苷酸(hexanucleotide)的微卫星位点, 对应的最低重复次数分别为10次、6次、5次、5次、5次和5次, 侧翼长度≥10 bp, 两个微卫星序列间的间隔大于12 bp。参考Weber(1990)评价微卫星的标准, 将所有微卫星位点区分为三种类型: 完美型、非完美型和复合型。
1.4 多态性微卫星位点分析基于微卫星位点所在序列, 采用Primer3 v2.37软件(Koressaar et al, 2007; Untergasser et al, 2012; Kõressaar et al, 2018)设计微卫星引物, 参数设置为默认值。从设计出的引物中随机挑选150对合成, 以横带髭鲷基因组DNA为模板进行PCR扩增。引物合成由上海生工生物公司完成。PCR扩增反应体系为25 μL, 包括: 1 μL模板DNA(50 ng), 2.5 μL 10*buffer, 2 μL dNTPs, 正反引物各1 μL (10 μmol/L), 17.5 μL ddH2O, 0.15 μL Easy Taq DNA聚合酶(TaKaRa, 北京)。PCR扩增程序设置为: 94 ℃预变性5 min、94 ℃变性30 s、最佳退火温度退火30 s、72 ℃延伸30 s、共32个循环, 最后72 ℃延伸10 min。所有引物均进行了最佳退火温度的筛选, 退火温度梯度设为52~60 ℃。PCR扩增产物通过8%非变性聚丙烯酰胺凝胶电泳和银染法检测。
1.5 数据分析与统计测序所得原始数据经质控, 过滤低质量数据后, 保留clean data使用Jellyfish v2.01 (Marçais et al, 2011)和GenomeScopev1.0软件(Vurture et al, 2017)进行K-mer分析, 以评估基因组大小、杂合率以及重复序列信息。横带髭鲷基因组草图通过使用SOAPdenovo v2.2.10软件(Luo et al, 2015)基于de Bruijin算法组装获得。基因组大小估算公式为: 基因组大小G=K-mer数量/峰值深度(Marçais et al, 2011)。
微卫星序列特征分析与统计: 使用Excel Microsatelite Toolkit (Shaibi et al, 2008)计算等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)以及多态性信息含量(PIC)等遗传参数; 使用Micro-Checker (Van Oosterhout et al, 2004)检验无效等位基因; Genepop v4.0 (Rousset, 2008)对位点进行哈迪-温伯格平衡检测。
2 结果 2.1 横带髭鲷基因组组装与基因组大小预测基因组survey测序共获得clean data83.02 G。Q20、Q30和GC含量分别为97.54%、93.1%和43.65%, 测序深度约158×。对过滤后的高质量数据随机抽取10 000对Reads数据, 进行NT库比对, 前80%的物种成功比对到横带髭鲷的近缘物种DNA上, 证明该文库数据中不含有明显的外源污染。碱基频率分布检查无AT、GC分离现象。拼接共获得Contigs总长度644 630 724 bp, 组装后获得Scaffolds总长度675 336 059 bp, Scaffolds N50长度为1 589 bp (表 1)。K-mer值设为17进行分析, 17-mer深度为114, 对应K-mer数量为64 483 008 615, 预估横带髭鲷基因组大小约为543 Mb。分析显示横带髭鲷基因组杂合率为0.45%, 重复率为26.78%。综上表明, 横带髭鲷基因组文库测序质量正常, 可用于后续信息分析。
| 类别 | 总长度/bp | 序列总数 | ≥2 kb | 最大长度/bp | N50/bp | GC含量/% |
| Contigs | 644 630 724 | 1 645 244 | 37 450 | 19 715 | 682 | 42.8 |
| Scaffolds | 675 336 059 | 1 115 882 | 59 651 | 137 823 | 1 589 | 40.9 |
在横带髭鲷基因组组装获得的Scaffolds数据中共检测到280 378个完美型微卫星位点。微卫星出现频率为25.13%, 微卫星序列相对丰度为415.17个/Mb, 即平均每2.4 Kb出现一个微卫星位点。微卫星总长度为5 824 206 bp, 平均长度为20.77 bp。对微卫星的组成类型和数量进行分析, 结果表明: 二核苷酸重复是横带髭鲷基因组最丰富的微卫星类型, 占比56.05%; 其次为单核苷酸重复与三核苷酸重复, 分别占比29.20%和10.28%, 上述三种重复类型占微卫星总量的95.53%。而其余重复类型占比很低, 四核苷酸(2.99%)、五核苷酸(1.03%)、六核苷酸(0.45%)这三种重复仅占微卫星总量的4.47% (表 2)。
| 重复类型 | 数量 | 比例/% | 相对丰度/(个/Mb) | 平均长度/bp | 总长度/bp | 平均距离/(kb/个) |
| 单核苷酸 | 81 867 | 29.20 | 121.22 | 12.37 | 1 012 772 | 8.25 |
| 二核苷酸 | 157 144 | 56.05 | 232.69 | 24.24 | 3 808 858 | 4.30 |
| 三核苷酸 | 28 828 | 10.28 | 42.69 | 20.96 | 604 320 | 23.42 |
| 四核苷酸 | 8 378 | 2.99 | 12.41 | 29.55 | 247 624 | 80.61 |
| 五核苷酸 | 2 886 | 1.03 | 4.27 | 32.79 | 94 640 | 234.00 |
| 六核苷酸 | 1 275 | 0.45 | 1.89 | 43.91 | 55 992 | 529.67 |
| 总计 | 280 378 | 100.00 | 415.17 | 20.77 | 5 824 206 | 2.40 |
横带髭鲷基因组survey测序共检测到重复单元1 253种, 五、六核苷酸重复单元种类最丰富, 分别为484和515种。在单核苷酸重复中A/T为优势重复单元, 达到了62 714个, 占全部微卫星位点数的22.37%; 二核苷酸以TG/CA为主, 占全部微卫星位点数的21.98%; 三核苷酸重复中占比较高的重复单元有AAT/ATT; 四核苷酸重复中出现频率较高的是AATG/ CATT; 五核苷酸重复以ATCTT/AAGAT为主, 六核苷酸重复中CCTAAC/GTTAGG重复单元数量较多(表 3)。
| 重复类型 | 种类 | 最多重复单元 | 最少重复单元 | |||||
| 单元类型 | 位点数量 | 比例/% | 单元类型 | 位点数量 | 比例/% | |||
| 单核苷酸 | 2 | A/T | 62 714 | 22.37 | C/G | 19 153 | 6.83 | |
| 二核苷酸 | 8 | TG/CA | 61 641 | 21.98 | GC/CG | 164 | 0.06 | |
| 三核苷酸 | 30 | AAT/ATT | 2 725 | 0.97 | ACG/CGT | 22 | ||
| 四核苷酸 | 214 | AATG/CATT | 577 | 0.20 | ||||
| 五核苷酸 | 484 | ATCTT/AAGAT | 328 | 0.12 | ||||
| 六核苷酸 | 515 | CCTAAC/GTTAGG | 131 | 0.04 | ||||
各核苷酸类型的微卫星在重复次数变异上也存在巨大差别。如图 1所示, 10次重复和6次重复的微卫星位点数量最多, 分别占横带髭鲷基因组微卫星总数的14.60%和12.46%。从重复次数与对应微卫星位点数量变化趋势看, 低重复次数的微卫星序列数量较多, 高重复次数的微卫星序列数量较少。随着重复次数的增多, 核苷酸重复类型的位点数在重复次数为6时达到第一次峰值, 重复次数为10时达到第二次峰值, 后呈递减趋势。
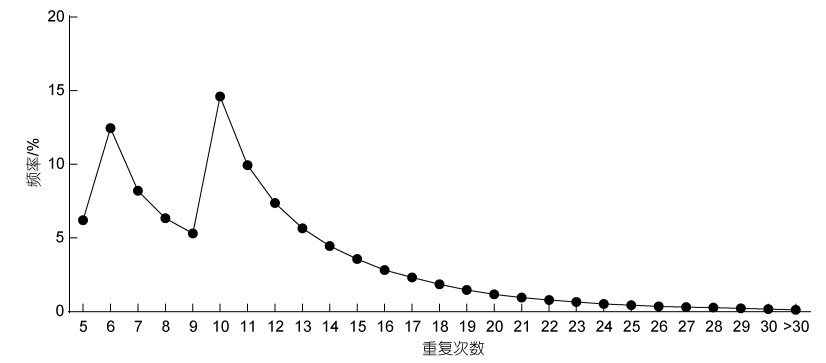 |
| 图 1 横带髭鲷基因组微卫星重复次数频率分布 Fig. 1 Frequency distribution of genome microsatellite repeats in H. analis |
本研究共筛选150对微卫星引物, 其中有84对可稳定扩增出清晰明亮的条带, 具有多态性的微卫星标记共20对, 包括14个二核苷酸重复、3个三核苷酸重复和3个四核苷酸重复位点。图 2展示供试群体在HDZD-46位点的扩增结果。20个微卫星位点共检测到等位基因175个, 各位点等位基因数从5~13不等, 平均等位基因数为8.75个, 观测杂合度(Ho)范围为0.500~0.917, 平均值为0.808; 期望杂合度(He)范围为0.719~0.918, 平均值为0.863; 多态信息含量(PIC)在0.660~0.890之间, 平均值为0.826。经Bonferroni校正后有5个位点(HDZD-16、HDZD-66、HDZD-68、HDZD-117和HDZD-119)显著偏离哈迪-温伯格平衡。有4个位点(HDZD-16、HDZD-68、HDZD-117和HDZD-119)存在无效等位基因(表 4)。
 |
| 图 2 横带髭鲷在HDZD-46位点的聚丙烯酰胺电泳图谱 Fig. 2 Polyacrylamide gel electrophoresis at HDZD-46 in H. analis |
| 位点 | 核心序列 | 引物序列(5′-3′) | 最适退火温度/℃ | 片段区间/bp | 等位基因数 | 观测杂合度 | 期望杂合度 | 多态信息含量 | 哈迪-温伯格平衡 |
| HDZD-2 | (AC)6 | F: CCACCGCATCATTTATTCCT R: CTTCGGAGATGGATGAGAGC |
59.4 | 270~278 | 7 | 0.542 | 0.859 | 0.822 | 0.005 |
| HDZD-16 | (CT)9 | F: TTGTCTACGAGCGTCCTGTG R: GGGTGGAGACAGATGAAAGC |
52.5 | 231~233 | 9 | 0.917 | 0.882 | 0.848 | 0.000* |
| HDZD-42 | (CT)12 | F: GCGACTTCAGCAGCAATATG R: TGTAAATGCTGGTGCGAGAG |
56.9 | 188~201 | 8 | 0.625 | 0.849 | 0.809 | 0.002 |
| HDZD-43 | (TA)6 | F: CACAGCTGACGCTAAACCAC R: CCATCTCTCCACTCCTCTCG |
56.9 | 176~279 | 5 | 0.500 | 0.719 | 0.660 | 0.033 |
| HDZD-46 | (TGTA)5 | F: AAATGAACGGGAGACACGAG R: GTGTTTCTTGAGCCTCTGGC |
55.1 | 145~229 | 7 | 0.542 | 0.812 | 0.766 | 0.003 |
| HDZD-47 | (GT)20 | F: GGCTCTACAGGCCTCTTCCT R: TCTGACAGCATTGGCAGTTC |
55.1 | 194~223 | 10 | 0.958 | 0.893 | 0.862 | 0.081 |
| HDZD-53 | (GT)16 | F: AATTCATGCAGAAGGGATGC R: CCATCATGAGACCAATGTGC |
53.5 | 225~261 | 10 | 0.958 | 0.901 | 0.871 | 0.433 |
| HDZD-66 | (TG)10 | F: CCGCCATTTAACACCTGTCT R: TACTCCTTGTCCGTGCTGTG |
53.5 | 204~213 | 8 | 0.875 | 0.866 | 0.829 | 0.000* |
| HDZD-68 | (AC)14 | F: ACTACTGCTGGCCATTTGCT R: CTCTGCACCAAGACGTGTGT |
53.5 | 226~245 | 11 | 0.875 | 0.918 | 0.890 | 0.000* |
| HDZD-79 | (AC)12 | F: AACGTGCAGACACAGAAACG R: CGAGGGTTTGGATTATGCAG |
55.1 | 142~180 | 10 | 0.750 | 0.871 | 0.837 | 0.006 |
| HDZD-87 | (AC)21 | F: ATTATGTCCAGGTTGCAGGC R: GCAACGATACTGCTCCCTTC |
55.1 | 247~260 | 9 | 0.958 | 0.858 | 0.823 | 0.016 |
| HDZD-102 | (GT)17 | F: ATCCTGAGAATCCCAAGCCT R: GGCCATCTCCTAATCCCAGT |
55.1 | 200~260 | 7 | 0.833 | 0.850 | 0.809 | 0.176 |
| HDZD-103 | (TCTG)7 | F: GGAGGGAAAGAGTGGAGGTC R: CAGCGCTTCCTATGGAGAAC |
58.4 | 240~269 | 9 | 0.833 | 0.885 | 0.853 | 0.354 |
| HDZD-104 | (TTG)7 | F: TTGCAAAGTGACGGAGACTG R: GGGAGAGAGAGTGATGCTGG |
55.1 | 168~257 | 6 | 0.917 | 0.803 | 0.754 | 0.003 |
| HDZD-106 | (TG)27 | F: TCTCGAGATGTTTGCCCTCT R: CACGCCTACACAAGCAGATG |
59.4 | 268~275 | 11 | 0.917 | 0.895 | 0.865 | 0.015 |
| HDZD-109 | (GT)12 | F: TGCTGGCAGTGTCTCAGAGT R: ACTGACAGTCATGCAGTGGG |
55.1 | 243~273 | 8 | 0.833 | 0.873 | 0.838 | 0.011 |
| HDZD-116 | (AAC)5 | F: CTATAGCCTGCTCCACTGCC R: CTCCTCGGAGTTTGAGTTGC |
58.4 | 244~266 | 9 | 0.917 | 0.859 | 0.823 | 0.018 |
| HDZD-117 | (AC)19 | F: ACTACCTGGTCAGGTGGCAG R: TTAGTGGGCTTGGTGTCCTC |
58.4 | 212~264 | 9 | 0.792 | 0.878 | 0.844 | 0.000* |
| HDZD-119 | (TATC)5 | F: AACTGCAGGATGTTCTTGGG R: CTCGTCCTTGCATCAGGAAT |
56.9 | 267~271 | 9 | 0.875 | 0.889 | 0.861 | 0.000* |
| HDZD-135 | (AAT)8 | F: CCATTGATGGGAGGTGAATC R: TGTATTTCTGCTGGTGGCTG |
56.9 | 156~182 | 13 | 0.750 | 0.897 | 0.868 | 0.003 |
| 注: *经Bonferroni校正显著偏离哈迪-温伯格平衡(P < 0.001) | |||||||||
高通量测序的发展为横带髭鲷这样的非模式生物的遗传学研究提供了很大的便利, 基于基因组筛选微卫星位点逐渐成为开发微卫星位点的重要手段之一。本研究首次对横带髭鲷基因组进行survey测序, K-mer分析结果预测横带髭鲷的基因组大小为543 Mb。截至目前统计, 硬骨鱼类中红鳍东方鲀(Takifugu rubripes)的基因组最小, 为322.5 Mb (Aparicio et al, 2002); 基因组最大的为非洲肺鱼(Protopterus annectens)达到了40 G (Wang et al, 2021)。而大部分鱼类的基因组大小均小于1 G, 如半滑舌鳎(Cynoglossus semilaevis) 477 Mb (Chen et al, 2014)、大黄鱼(Larimichthys crocea) 728 Mb (Wu et al, 2014)、中国鱚(Sillago sinica) 534 Mb (Xu et al, 2018)和罗非鱼(Oreochromis niloticus) 815 Mb (Brawand et al, 2014)等。而基因组大小的增加与简单重复序列的数量增加有关(Hancock, 1996), 横带髭鲷基因组小于1 G, 与大部分鱼类无太大差异, 推测其简单重复序列的数量应该与基因组大小相近的物种无明显差异。除基因组大小外, 本研究组装得到的其他横带髭鲷基因组草图信息, 如GC含量、Scaffolds长度等也为以后的基因组方面的深入研究奠定了基础。
利用de novo基因组高通量测序技术, 组装得到Scaffolds数据中共检测到280 378个完美型微卫星位点, 低于在斑尾刺虾虎鱼(Acanthogobius ommaturus)和多鳞鱚(Sillago sihama)中检测到微卫星位点的数量(Chen et al, 2020; Qiu et al, 2020), 分别为349 138个和630 028个; 其出现频率为25.13%, 低于龙头鱼(Harpadon nehereus) (40.47%), 相对丰度为415.17个/Mb, 低于龙头鱼(753.12/Mb) (黄新芯等, 2021), 高于褐菖鲉(Sebastiscus marmoratus) (314.6/Mb) (Xu et al, 2018)。其中, 龙头鱼(1 315 Mb) (Yang et al, 2021)、斑尾刺虾虎鱼(928.01 Mb)和褐菖鲉(800 Mb)的基因组大于横带髭鲷, 多鳞鱚(522.6 Mb)的基因组小于横带髭鲷, 有学者认为, 基因组越大其微卫星的大小或者相对丰度也随之增大(Crollius et al, 2000; Schlötterer et al, 2000), 但经过对比, 我们认为这样的推论欠妥。横带髭鲷微卫星组成类型结果分析表明, 随着核苷酸数量增多, 微卫星数量呈递减现象。其中二核苷酸重复最为丰富, 这与真核生物中二核苷酸重复类型的微卫星最为常见的普遍规律一致(罗文永等, 2003); 单核苷酸重复数量仅次于二核苷酸重复, 但是单核苷酸重复作为分子标记的用途不大, 一般在设计引物时不纳入考虑范围; 其余核苷酸重复数量相对较少。此外, 核苷酸重复类型的数量占比与物种进化水平有紧密的联系, 低级重复类型数量多表明物种进化水平高, 高级重复类型占比大则表明物种具较短的进化时间或者变异频率低(Harr et al, 2000; Li et al, 2017; Lei et al, 2021)。在横带髭鲷的微卫星组成中, 一、二、三核苷酸重复类型占比达微卫星总量的95.53%, 我们认为这可能是由于横带髭鲷的进化历程长造成的。这提示我们在大批量开发微卫星时, 二、三核苷酸重复类型应当是主要的筛选对象, 这些类型的微卫星位点具较高的分子标记开发潜力。
在横带髭鲷基因组中, 所有微卫星位点中共有1 253种重复单元, 随着核苷酸数量增多, 各类型的重复单元种类呈递增趋势, 但其数量呈递减趋势。单核苷酸重复中A/T含量丰富, C/G较少; 二核苷酸重复中TG/CA含量较多, GC/CG含量较少; 三核苷酸重复中ACG/CGT含量极少。三种重复类型中GC含量均为少数, 这一特点是由于脊椎动物基因组中GC碱基重复较为罕见, 可能与基因组中CpG岛甲基化, 胞苷酸C易通过脱氨基作用转变为胸腺嘧啶T有关(Schorderet et al, 1992)。此外, 在比较各重复单元的重复次数时, 我们发现重复次数为6时, 微卫星数量达到第一次峰值; 重复次数为10时的微卫星数量最多, 达到第二次峰值, 在第二次峰值过后, 微卫星位点数量随着碱基重复单元的重复次数的增加而减少。这一结果不符合大多数真核生物微卫星数目随碱基重复单元的重复次数增加呈指数下降趋势的一般规律。结合重复单元与重复次数, 在横带髭鲷微卫星位点开发中, 优先选择优势重复单元与重复次数较高的微卫星是提高微卫星多态性有效的方法, 这在筛选多态性微卫星位点的结果中也得到了印证。
本研究共筛选出20对具多态性的微卫星位点, 并应用于供试群体遗传多样性检测。对于微卫星多态性的评价通常根据Botstein等(1980)提出的标准进行, 即当PIC > 0.5时, 位点为高度多态; 当0.25 < PIC < 0.5时, 位点为中度多态; 当PIC < 0.25时, 位点为低度多态。据此, 本研究中的20个微卫星位点均呈现出较高的多态性。杂合度同样是反映物种遗传多样性水平的重要指标, 当杂合度介于0.5~0.8之间时, 可认为供试群体具有较高的遗传多样性。本研究中的20个微卫星位点平均期望杂合度与观测杂合度均达到了0.8, 说明供试群体杂合性高, 遗传多样性高。检测20个微卫星位点中有5个位点显著偏离哈迪-温伯格平衡, 其中有4个位点检测到无效等位基因, 推测无效等位基因存在可能是导致偏离哈迪-温伯格平衡的重要原因。但微卫星位点扩增出现无效等位基因是不能通过一个群体的遗传分析得以解决的, 还需通过家系分析来确认, 以获取更精确的微卫星位点信息(宋忠魁等, 2013)。
4 结论本研究首次使用基因组survey测序方法构建了横带髭鲷的基因组草图, 评估了基因组大小, 并在基因组范围内识别开发了20对具有较高多态性的微卫星位点。研究结果可用于今后横带髭鲷群体遗传学和遗传育种的研究。
王世锋, 杜佳莹, 苏永全, 等, 2008. 厦门近海横带髭鲷野生群体遗传结构分析. 厦门大学学报(自然科学版), 47(1): 88-93 DOI:10.3321/j.issn:0438-0479.2008.01.020 |
平洪领, 张涛, 史会来, 等, 2021. 横带髭鲷早期生长发育特征. 中国水产科学, 28(3): 276-287 |
史海东, 毛国民, 王海岳, 2004. 温度和盐度对横带髭鲷胚胎发育的影响. 上海水产大学学报, 13(3): 230-234 |
许文军, 辛剑, 张学舒, 等, 2002. 横带髭鲷Hapalogeny mucronatus (Eydoux et Souleyet)白点虫病的防治研究. 现代渔业信息, 17(11): 24-25, 13 DOI:10.3969/j.issn.1004-8340.2002.11.007 |
宋忠魁, 聂振平, 谢达, 等, 2013. 拟穴青蟹(Scylla paramamosain)微卫星位点开发——基于(AG)12探针杂交. 海洋与湖沼, 44(1): 171-176 |
张涛, 平洪领, 史会来, 等, 2020. 舟山海域不同规格横带髭鲷营养成分分析及品质评价. 浙江海洋大学学报(自然科学版), 39(6): 502-508 DOI:10.3969/j.issn.1008-830X.2020.06.005 |
罗文永, 胡骏, 李晓方, 2003. 微卫星序列及其应用. 遗传, 25(5): 615-619 DOI:10.3321/j.issn:0253-9772.2003.05.027 |
赵盛龙, 徐汉祥, 钟俊生, 等, 2016. 浙江海洋鱼类志. 杭州: 浙江科学技术出版社, 660-661
|
姚海富, 毛国民, 史海东, 2006. 横带髭鲷Hapalogenys mucronatus (Eydoux et Souleyet)亲鱼培育、产卵和孵化的初步试验. 现代渔业信息, 21(4): 15-17 DOI:10.3969/j.issn.1004-8340.2006.04.005 |
徐田军, 王健鑫, 孙悦娜, 等, 2010. 基于髭鲷属鱼类Cyt b基因全序列探讨髭鲷属在鲈总科的分类地位. 动物分类学报, 35(3): 530-536 |
黄新芯, 蒋艳琳, 蒋小姿, 等, 2021. 基于高通量转录组测序技术的龙头鱼微卫星信息分析. 浙江海洋大学学报(自然科学版), 40(3): 189-197 DOI:10.3969/j.issn.1008-830X.2021.03.001 |
喻子牛, 孔晓瑜, 徐文武, 等, 1994. 斜带髭鲷Hapalogenys nitens (Richardson)和横带髭鲷H. mucronatus (Eydoux et Souleyet)的核型. 青岛海洋大学学报, 24(2): 175-180 |
喻子牛, 孔晓瑜, 谢宗墉, 1995. 山东近海21种经济鱼类的核型研究. 中国水产科学, 2(2): 1-6 DOI:10.3321/j.issn:1005-8737.1995.02.010 |
APARICIO S, CHAPMAN J, STUPKA E, et al, 2002. Whole-Genome shotgun assembly and analysis of the genome of Fugu rubripes. Science, 297(5585): 1301-1310 DOI:10.1126/science.1072104 |
BEIER S, THIEL T, MÜNCH T, et al, 2017. MISA-web: a web server for microsatellite prediction. Bioinformatics, 33(16): 2583-2585 DOI:10.1093/bioinformatics/btx198 |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al, 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 32(3): 314-331 |
BRAWAND D, WAGNER C E, LI Y I, et al, 2014. The genomic substrate for adaptive radiation in African cichlid fish. Nature, 513(7518): 375-381 DOI:10.1038/nature13726 |
CAPOBIANCHI M R, GIOMBINI E, ROZERA G, 2013. Next-generation sequencing technology in clinical virology. Clinical Microbiology and Infection, 19(1): 15-22 DOI:10.1111/1469-0691.12056 |
CHEN B J, SUN Z C, LOU F R, et al, 2020. Genomic characteristics and profile of microsatellite primers for Acanthogobius ommaturus by genome survey sequencing. Bioscience Reports, 40(11): BSR20201295 DOI:10.1042/BSR20201295 |
CHEN S L, ZHANG G J, SHAO C W, et al, 2014. Whole-genome sequence of a flatfish provides insights into ZW sex chromosome evolution and adaptation to a benthic lifestyle. Nature Genetics, 46(3): 253-260 DOI:10.1038/ng.2890 |
CHENG J, YANAGIMOTO T, SONG N, et al, 2015. Population genetic structure of chub mackerel Scomber japonicus in the Northwestern Pacific inferred from microsatellite analysis. Molecular Biology Reports, 42(2): 373-382 DOI:10.1007/s11033-014-3777-2 |
CROLLIUS H R, JAILLON O, DASILVA C, et al, 2000. Characterization and repeat analysis of the compact genome of the freshwater pufferfish Tetraodon nigroviridis. Genome Research, 10(7): 939-949 DOI:10.1101/gr.10.7.939 |
CSENCSICS D, BRODBECK S, HOLDEREGGER R, 2010. Cost-effective, species-specific microsatellite development for the endangered Dwarf Bulrush (Typha minima) using next-generation sequencing technology. Journal of Heredity, 101(6): 789-793 DOI:10.1093/jhered/esq069 |
HANCOCK J M, 1996. Simple sequences and the expanding genome. BioEssays, 18(5): 421-425 DOI:10.1002/bies.950180512 |
HARR B, SCHLÖTTERER C, 2000. Long microsatellite alleles in Drosophila melanogaster have a downward mutation bias and short persistence times, which cause their genome-wide underrepresentation. Genetics, 155(3): 1213-1220 DOI:10.1093/genetics/155.3.1213 |
KÕRESSAAR T, LEPAMETS M, KAPLINSKI L, et al, 2018. Primer3_masker: integrating masking of template sequence with primer design software. Bioinformatics, 34(11): 1937-1938 DOI:10.1093/bioinformatics/bty036 |
KORESSAAR T, REMM M, 2007. Enhancements and modifications of primer design program Primer3. Bioinformatics, 23(10): 1289-1291 DOI:10.1093/bioinformatics/btm091 |
LEI Y, ZHOU Y, PRICE M, et al, 2021. Genome-wide characterization of microsatellite DNA in fishes: survey and analysis of their abundance and frequency in genome- specific regions. BMC Genomics, 22(1): 421 DOI:10.1186/s12864-021-07752-6 |
LI Z G, CHEN F, HUANG C H, et al, 2017. Genome-wide mapping and characterization of microsatellites in the swamp eel genome. Scientific Reports, 7(1): 3157 DOI:10.1038/s41598-017-03330-7 |
LU X, LUAN S, KONG J, et al, 2017. Genome-wide mining, characterization, and development of microsatellite markers in Marsupenaeus japonicus by genome survey sequencing. Chinese Journal of Oceanology and Limnology, 35(1): 203-214 DOI:10.1007/s00343-016-5250-7 |
LÜ Z M, LI H M, LIU L Q, et al, 2013. Rapid development of microsatellite markers from the large yellow croaker (Pseudosciaena crocea) using next generation DNA sequencing technology. Biochemical Systematics and Ecology, 51: 314-319 DOI:10.1016/j.bse.2013.09.019 |
LUO R B, LIU B H, XIE Y L, et al, 2015. SOAPdenovo2: an empirically improved memory-efficient short-read de novo assembler. GigaScience, 4(1): 30 |
MADESIS P, GANOPOULOS I, TSAFTARIS A, 2013. Microsatellites: evolution and contribution [M] // KANTARTZI S K. Microsatellites: Methods and Protocols. Totowa: Humana Press: 1-13.
|
MARÇAIS G, KINGSFORD C, 2011. A fast, lock-free approach for efficient parallel counting of occurrences of k-mers. Bioinformatics, 27(6): 764-770 DOI:10.1093/bioinformatics/btr011 |
QIU B X, FANG S B, IKHWANUDDIN M, et al, 2020. Genome survey and development of polymorphic microsatellite loci for Sillago sihama based on Illumina sequencing technology. Molecular Biology Reports, 47(4): 3011-3017 DOI:10.1007/s11033-020-05348-z |
ROUSSET F, 2008. GENEPOP' 007: a complete re-implementation of the GENEPOP software for Windows and Linux. Molecular Ecology Notes, 8(1): 103-106 |
SCHLÖTTERER C, HARR B, 2000. Drosophila virilis has long and highly polymorphic microsatellites. Molecular Biology and Evolution, 17(11): 1641-1646 DOI:10.1093/oxfordjournals.molbev.a026263 |
SCHORDERET D F, GARTLER S M, 1992. Analysis of CpG suppression in methylated and nonmethylated species. Proceedings of the National Academy of Sciences of the United States of America, 89(3): 957-961 DOI:10.1073/pnas.89.3.957 |
SHAIBI T, LATTORFF H, MORITZ R, 2008. A microsatellite DNA toolkit for studying population structure in Apis mellifera. Molecular Ecology Resources, 8(5): 1034-1036 DOI:10.1111/j.1755-0998.2008.02146.x |
TAUTZ D, 1989. Hypervariability of simple sequences as a general source for polymorphic DNA markers. Nucleic Acids Research, 17(16): 6463-6471 DOI:10.1093/nar/17.16.6463 |
UNTERGASSER A, CUTCUTACHE I, KORESSAAR T, et al, 2012. Primer3—new capabilities and interfaces. Nucleic Acids Research, 40(15): e115 DOI:10.1093/nar/gks596 |
VAN OOSTERHOUT C, HUTCHINSON W F, WILLS D P M, et al, 2004. Micro-checker: software for identifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes, 4(3): 535-538 DOI:10.1111/j.1471-8286.2004.00684.x |
VURTURE G W, SEDLAZECK F J, NATTESTAD M, et al, 2017. GenomeScope: fast reference-free genome profiling from short reads. Bioinformatics, 33(14): 2202-2204 DOI:10.1093/bioinformatics/btx153 |
WANG K, WANG J, ZHU C L, et al, 2021. African lungfish genome sheds light on the vertebrate water-to-land transition. Cell, 184(5): 1362-1376 |
WEBER J L, 1990. Informativeness of human (dC-dA)n·(dG-dT)n polymorphisms. Genomics, 7(4): 524-530 |
XU S Y, SONG N, XIAO S J, et al, 2020. Whole genome survey analysis and microsatellite motif identification of Sebastiscus marmoratus. Bioscience Reports, 40(2): BSR20192252 |
XU S Y, XIAO S J, ZHU S L, et al, 2018. A draft genome assembly of the Chinese sillago (Sillago sinica), the first reference genome for Sillaginidae fishes. GigaScience, 7(9): giy108 |
WU C W, ZHANG D, KAN M Y, et al, 2014. The draft genome of the large yellow croaker reveals well-developed innate immunity. Nature Communications, 5: 5227 |
YANG T Y, HUANG X X, NING Z J, et al, 2021. Genome-wide survey reveals the microsatellite characteristics and phylogenetic relationships of Harpadon nehereus. Current Issues in Molecular Biology, 43(3): 1282-1292 |
ZANE L, BARGELLONI L, PATARNELLO T, 2002. Strategies for microsatellite isolation: a review. Molecular Ecology Notes, 11(1): 1-16 |
 2023, Vol. 54
2023, Vol. 54


