
中国海洋湖沼学会主办。
文章信息
- 徐敏慧, 颜成瑞, 顾忠旗, 黄继, 李继姬, 叶莹莹, 夏立萍. 2023.
- XU Min-Hui, YAN Cheng-Rui, GU Zhong-Qi, HUANG Ji, LI Ji-Ji, YE Ying-Ying, XIA Li-Ping. 2023.
- 厚壳贻贝(Mytilus coruscus)Hox基因家族的鉴定、进化与表达分析
- IDENTIFICATION, EVOLUTION, AND EXPRESSION PATTERN OF HOX FAMILY IN MYTILUS CORUSCUS
- 海洋与湖沼, 54(3): 895-906
- Oceanologia et Limnologia Sinica, 54(3): 895-906.
- http://dx.doi.org/10.11693/hyhz20221000279
文章历史
-
收稿日期:2022-10-28
收修改稿日期:2023-01-03
2. 嵊泗县海洋科技研究所 浙江嵊泗 202450
2. Shengsi Marine Science and Technology Institute, Shengsi 202450, China
同源异型盒基因(homeobox gene)包含一条约180个碱基的保守DNA序列, 约编码60 ~ 61个氨基酸的同源结构域(homedomain, HD) (Bürglin, 2011)在控制脊椎动物生长和发育, 体轴发育、中枢神经系统和肢体发育中均发挥重要的作用(陈远, 2013)。从植物和酵母细胞到动物和人类组织均存在同源异型盒基因的多个拷贝, 其中最有代表性的是Hox基因家族(He et al, 2018)。作为一种含有同源结构域的基因, 它通常在染色体中呈簇状分布, 保守性较高。并且, 它在对躯体和组织特征的决定以及胚胎发育中器官的形成方面起着重要的调节作用(王师等, 2017)。
高度保守的Hox基因家族是生物成体形态的主要架构者, 为生物体正常的身体模式所必需的基因时空表达提供了基础。Hox基因自被发现以来, 一直被认为是动物界中形态进化的主要驱动因素(Pearson et al, 2005)。在软体动物Hox基因的来源及组成上, De Rosa等(1999)首先推测Hox基因在进化过程中, 数目由原口动物共同祖先中的8个变为冠轮动物共同祖先中的10个; Winnepenninckx等(1996)分析了分子数据中大量的信息, 并认为, 软体动物是单源起源的; Callaerts等(2002)推测软体动物基因组中仅含有一个Hox基因簇; 此后, Iijima等(2006)分离鉴定了Hox基因在7个物种中的分布情况, 并认为软体动物最初至少含有12个Hox基因(Hox1、Hox2、Hox3、Hox4、Hox5a、Hox5b、Hox6a、Hox6b、Hox7、Hox8、Post1和Post2); 同时, 基于冠轮动物和蜕皮动物Hox基因调控中后区的明显区别, 将两者的Hox基因分别命名, 前者为Lox5、Lox2、Lox4、Post1、Post2, 而后者为ftz、Antp、Ubx、Abd-A、Abd-B (Balavoine et al, 2002); 在笠贝(Lottia gigantea)中, 完整的Hox基因簇首先被发现和提及。软体动物一个完整的Hox基因簇包含Hox1、Hox2、Hox3、Hox4、Hox5、Antp、Lox2、Lox4、Lox5、Post1和Post2总共11个基因, 王师等(2017)研究发现双壳贝类物种的Hox基因簇也含有11个Hox基因, 并且Hox基因的转录方向与L. gigantea的一致。在后生动物胚胎发育过程中, Hox基因参与了前后轴身体结构的调控。有人认为, 不同物种类群之间表现出的丰富的形态多样性可能与Hox基因簇的变化有关, 尤其是关于基因簇复制, 基因复制、基因丢失、基因表达变异和调控相互作用(Garcia-Fernàndez, 2005)。事实上, 已经存在较多对于软体动物Hox基因的研究和调查。Hinman从腹足类物种驴耳鲍(Haliotins asinine)中提取出Hox1、Hox2、Hox3、Hox4和Hox5基因。Hox2、Hox3和Hox4在转子腹侧的神经外胚层细胞中表达, 与Hox5一起在发育中的神经节中表达; Has-Hox1和Has-Hox4在壳形成区域表达, 表明这两个基因参与腹足动物壳的形态发生(Samadi et al, 2009)。在腹足类中, Samadi等(2009)报告了Gibbula varia物种中Hox1、Post1和Post2参与胚胎、幼虫和成虫壳的形成。Brooke等(1998)认为Hox基因簇最初可能以单一基因簇的形式存在, 通常称为magacluster类ANTP。在后生动物进化的早期阶段, 单一基因簇的ANTP通过串联复制产生了其他的旁系同源基因簇, 例如ParaHox基因簇。Carpintero等(2004)第一次从双壳贝类Pecten maximus中分离得到了ParaHox基因caudal和ANT-C中的Pb基因, 说明了Hox基因在双壳贝类中的存在; 在对菲律宾蛤仔(Ruditapes philippinarum)Hox基因研究发现, 11个Hox基因完整存在, 研究结果发现Hox最早在原肠胚时期开始表达, 在功能上, Hox1、Hox4、Post1和Post2基因可能与初生壳形成有关; Antp与Hox5基因可能与菲律宾蛤仔底栖生活适应有关; Hox2和Hox3基因可能与菲律宾蛤仔足形成有关(牟政强, 2015)。总体来说, Hox基因表现出多样性的表达, 表明其在成体或幼体特定组织中具有不同的功能。
软体动物作为原口动物的第二大类群, 存在于海洋、淡水以及陆地等多种环境中。目前已知的软体动物化石类群有6万种以上, 是动物界中仅次于节肢动物的第二大门类。厚壳贻贝(Mytilus coruscus)作为双壳纲代表性的物种之一, 在水生环境中发挥着重要的生态作用, 包括栖息地创造悬浮有机废物过滤和海岸线保护等(Ekstrom et al, 2015)。厚壳贻贝作为沿海重要的经济物种, 含有相对较高的粗蛋白和粗脂肪, 氨基酸种类丰富, 是补充人体营养的理想食品。软体动物在进化史上占据重要的地位, Hox基因除了在软体动物发育过程中所起到的作用, 在成体各组织中也会表达并执行一定的生物学功能。然而当前Hox基因在软体动物各组织中的表达模式尚未有太多研究, 软体动物Hox基因在成体中的表达数据需要被挖掘。同时, Hox作为一类重要的调控基因家族, 其基因数目, 基因排列顺序, 相关调控因子都直接与软体动物体态结构的多样性有关。因此, 需要对Hox基因进行更进一步的研究, 来对Hox的表达以及对应形成的机制进行修正和阐释。而软体动物作为原口动物中最重要的类群, 研究其中Hox基因的多种变化, 也有助于研究理解双侧对称动物的进化与遗传。因此, 本研究通过荧光定量PCR技术探究Hox基因在厚壳贻贝鳃、性腺、外套膜、闭壳肌、足、消化腺六个组织中的表达模式, 以探究Hox基因在厚壳贻贝组织中可能起到的功能和作用。
1 材料与方法 1.1 数据来源为了鉴定厚壳贻贝中含有homeodomain同源保守结构域的基因, 从NCBI数据库(http://www.ncbi.nlm.nih.gov/)中下载了厚壳贻贝全基因组数据(NCBI登录号: PRJEB33342)以及相关注释文件, 同时结合本课题组已完成的厚壳贻贝基因组解析(SRA数据库登录号: PRJNA635756)构建厚壳贻贝全基因组蛋白数据库。此外, 下载了NCBI数据库中软体动物的Hox蛋白序列(16个物种, 109条序列, 具体登录号见表 1)。
| 物种名 | 基因名 | 登录号 | 物种名 | 基因名 | 登录号 | |
| Acanthochitona rubrolineata | Hox1 | QCF47205 | Gymnomenia pellucida | Antp | APD15669 | |
| Acanthochitona crinita | Hox1 | AKV16302 | Acanthochitona crinita | Antp | AKV16307 | |
| Gymnomenia pellucida | Hox1 | APD15663 | Wirenia argentea | Lox2 | APD15716 | |
| Nucula tumidula | Hox1 | APD15698 | Gymnomenia pellucida | Lox2 | APD15670 | |
| Antalis entalis | Hox1 | APD15651 | Scutopus ventrolineatus | Lox2 | APD15709 | |
| Plakobranchus ocellatus | Hox1 | GFO20336 | Antalis entalis | Lox2 | APD15657 | |
| Lottia goshimai | Hox1 | QCF47216 | Acanthochitona rubrolineata | Lox2 | QCF47213 | |
| Steromphala varia | Hox2 | ADJ18233 | Acanthochitona crinita | Lox2 | APD15647 | |
| Mytilus edulis | Hox2 | CAG2258121 | Lottia goshimai | Lox2 | QCF47224 | |
| Plakobranchus ocellatus | Hox2 | GFO48376 | Steromphala varia | Lox2 | ADJ18238 | |
| Elysia marginata | Hox2 | GFS11912 | Acanthochitona crinita | Lox2 | AMT82759 | |
| Steromphala varia | Hox2 | ADJ18233 | Acanthochitona crinita | Lox4 | APD15648 | |
| Elysia marginata | Hox2 | GFN88056 | Acanthochitona rubrolineata | Lox4 | QCF47212 | |
| Xipholeptos notoides | Hox3 | APD15688 | Nucula tumidula | Lox4 | APD15703 | |
| Antalis entalis | Hox3 | APD15653 | Wirenia argentea | Lox4 | APD15717 | |
| Acanthochitona crinita | Hox3 | APD15643 | Gymnomenia pellucida | Lox4 | APD15671 | |
| Acanthochitona rubrolineata | Hox3 | QCF47207 | Plakobranchus ocellatus | Lox4 | GFO19732 | |
| Mytilus edulis | Hox3 | CAG2258122 | Elysia marginata | Lox4 | GFR63780 | |
| Nucula tumidula | Hox3 | APD15700 | Antalis entalis | Lox4 | APD15658 | |
| Wirenia argentea | Hox3 | APD15711 | Lottia goshimai | Lox4 | QCF47223 | |
| Gymnomenia pellucida | Hox3 | APD15665 | Steromphala varia | Lox4 | ADJ18237 | |
| Lottia goshimai | Hox3 | QCF47218 | Acanthochitona crinita | Lox4 | AMT82758 | |
| Steromphala varia | Hox3 | ADJ18232 | Plakobranchus ocellatus | Lox5 | GFO19711 | |
| Acanthochitona crinita | Hox3 | AKV16304 | Elysia marginata | Lox5 | GFR91347 | |
| Mytilus edulis | Hox3 | CAG2234314 | Acanthochitona crinita | Antp | APC93964 | |
| Mytilus edulis | Hox3 | CAG2234315 | Wirenia argentea | Antp | APD15715 | |
| Mytilus edulis | Hox3 | CAG2235200 | Wirenia argentea | Lox5 | APD15714 | |
| Mytilus edulis | Hox3 | CAG2234320 | Nucula tumidula | Lox5 | APD15702 | |
| Nucula tumidula | Hox4 | APD15701 | Acanthochitona crinita | Lox5 | APD15646 | |
| Acanthochitona crinita | Hox4 | APD15644 | Acanthochitona rubrolineata | Lox5 | QCF47210 | |
| Acanthochitona rubrolineata | Hox4 | QCF47208 | Acanthochitona crinita | Lox5 | AMT82757 | |
| Acanthochitona crinita | Hox4 | AKV16305 | Steromphala varia | Lox5 | ADJ18236 | |
| Mytilus edulis | Hox4 | CAG224127 | Wirenia argentea | Post1 | APD15718 | |
| Lottia goshimai | Hox4 | QCF47219 | Gymnomenia pellucida | Post1 | APD15672 | |
| Antalis entalis | Hox4 | APD15654 | Antalis entalis | Post1 | APD15659 | |
| Wirenia argentea | Hox4 | APD15712 | Nucula tumidula | Post1 | APD15704 | |
| Gymnomenia pellucida | Hox4 | APD15666 | Lottia goshimai | Post1 | QCF47226 | |
| Scutopus ventrolineatus | Hox5 | APD15708 | Steromphala varia | Post1 | ACX84673 | |
| Acanthochitona rubrolineata | Hox5 | QCF47209 | Acanthochitona rubrolineata | Post2 | QCF47214 | |
| Acanthochitona crinita | Hox5 | AKV16306 | Acanthochitona crinita | Post2 | AKV16308 | |
| Plakobranchus ocellatus | Hox5 | GFO39532 | Wirenia argentea | Post2 | APD15719 | |
| Elysia marginata | Hox5 | GFR79648 | Gymnomenia pellucida | Post2 | APD15673 | |
| Lottia goshimai | Hox5 | QCF47220 | Xipholeptos notoides | Post2 | APD15690 | |
| Steromphala varia | Hox5 | ADJ18234 | Nucula tumidula | Post2 | APD15705 | |
| Xipholeptos notoides | Hox5 | APD15689 | Antalis entalis | Post2 | APD15660 | |
| Antalis entalis | Hox5 | APD15655 | Plakobranchus ocellatus | Post2 | GFN88054 | |
| Mytilus edulis | Hox5 | CAG2241278 | Elysia marginata | Post2 | GFS11817 | |
| Acanthochitona crinita | Hox5 | APD15645 | Acanthochitona crinita | Post2 | APD15649 | |
| Xipholeptos notoides | Antp | APD15687 | Lottia goshimai | Post2 | QCF47225 | |
| Lottia goshimai | Antp | QCF47222 | Steromphala varia | Post2 | ACX84674 | |
| Mytilus edulis | Antp | CAG224128 | Mizuhopecten yessoensis | Genome | PRJNA747524 | |
| Acanthochitona rubrolineata | Antp | QCF47211 | Crassostrea gigas | Genome | PRJEB35351 | |
| Gymnomenia pellucida | Lox5 | APD15668 | Elysia chlorotica | Genome | PRJNA29229 | |
| Antalis entalis | Lox5 | APD15656 | Mytlius coruscus | Genome | PRJEB33342 | |
| Lottia goshimai | Lox5 | QCF47221 |
使用TBtools软件对厚壳贻贝蛋白数据库和软体动物Hox蛋白序列(虾夷扇贝Mizuhopecten yessoensis, 太平洋牡蛎Crassostrea gigas, 绿叶海蜗牛Elysia chlorotica, 厚壳贻贝M. coruscus)进行blast比对, 结合NCBI中Protein Blast功能, 对目的Hox基因家族序列进行进一步的鉴定和确认。使用Expasy网站在线工具(http://web.expasy.org/compute_pi/)预测了厚壳贻贝Hox序列的分子量(MW), 等电点(pI)和疏水值(GRAVY); 运用Euk-mPLoc2.0在线网站(http://www.csbio.sjtu.edu.cn/bioinf/euk-multi-2/)推测Hox基因家族成员的亚细胞定位。
1.3 厚壳贻贝Hox基因系统进化树构建为了获得贻贝中Hox基因的系统发育关系, 对所有鉴定出含有homeodomain的101条序列, 采用最大似然法(Maximum Likelihood)并利用MEGA 7.0 (Tamura et al, 2011)软件构建系统发育树, bootstrap设置为2 000个重复。另一方面, 对所有从NCBI上下载的部分软体动物Hox蛋白序列进行同样的多序列比以及构建系统发育树, 最后应用ITOL在线网站(https://itol.embl.de/)完成系统发育树的可视化和美化。相关序列的NCBI登录号如表 1所示。
1.4 Hox基因家族结构及染色体定位分析利用获得的厚壳贻贝Hox基因家族注释信息, 提取厚壳贻贝11个Hox基因的染色体定位信息, 通过TBtools软件可视化。
1.5 Hox基因家族保守基序及基因结构分析通过SMART (http://smart.embl-heidelberg.de/)和NCBI中的CD-search功能(https://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml)找出目的基因功能域的结构。利用MEME (http://meme-suite.org/index.html)在线工具对厚壳贻贝Hox基因家族成员进行Motif分析, 输出结构域数目最大值设置为10, 所得结果与使用MEGA7.0软件构建的厚壳贻贝Hox基因进化树在TBtools软件中进行可视化。
1.6 实时荧光定量PCR分析和表达分析厚壳贻贝成贝来自浙江省舟山市嵊泗列岛周围海域(122°46′E, 30°7′N), 选取贝体完整、健康、大小均一的厚壳贻贝作为样本, 解剖获取鳃、性腺、外套膜、闭壳肌、足、消化腺六个组织, 解剖获得组织后当场放入含有RNAlater溶液的2 mL冻存管中, 置于–80 ℃冰箱保存以便用于RNA的提取。厚壳贻贝不同成体组织按照RNA提取试剂RNAiso Plus (TaKaRa, Japan)提取方法提取RNA, 而后测定各样RNA浓度, 依据PrimerScript RTreagent Kit (Thermo Fisher Scientific)试剂盒提供方法进行反转录, 反转录cDNA用于后续qRT-PCR分析。基于厚壳贻贝全基因组中筛选到的11条Hox基因序列, 用软件Primer Primers5设计荧光定量PCR引物, 并以EF1a基因为内参(龙治海等, 2018)。由上海生工生物公司提供引物合成技术服务, 引物浓度10 μmol/L (表 2), 用TB Green Premix Ex Taq (Tli RNase H Plus) RR820A (TaKaRa, Japan)试剂盒进行荧光定量PCR反应, 使用Applied Biosystems 7500型荧光定量PCR仪(ABI, 美国)进行Hox基因在厚壳贻贝成体不同组织的相对定量表达分析。荧光定量PCR反应采用10 μL反应体系: TB Green Premix Ex Taq (Tli RNaseH Plus) (2X), 5 μL; PCR Forward Primer (10 μmol/L), 0.2 μL; PCR Reverse Primer (10 μmol/L), 0.2 μL; ROX Reference Dye II (50X)×2, 0.2 μL; cDNA, 1 μL; H2O, 3.4 μL。反应程序为: 50 ℃, 2 min; 95 ℃, 30 s; 95 ℃, 40个15 s循环; 58 ℃, 45 s; 95 ℃, 15 s; 72 ℃, 5 min。EF1a作为内参, 对目的基因的表达水平进行标准化。2–ΔΔCt法用来计算目的基因的相对表达量, 其中也包括表达量的均值±标准误(Mean±SE)。用TBtools软件可视化分析厚壳贻贝Hox基因家族在不同组织内的表达谱。
| 引物名称 | 引物序列5'~3' |
| Hox1F | ATGAATACGGAGAGTGATTACACAC |
| Hox1R | CATTGCTGTCATTGAAAGTCCAT |
| Hox2F | AGGGAAGAGAGTCTGGCTTTATTA |
| Hox2R | CTTGGTATTCCGGCTGTTGT |
| Hox3F | CAAACGCCGAGATGCATAA |
| Hox3R | TGAACTGATACTATTAGAACACGGA |
| Hox4F | GAACCCTAATTCATATACGGAGC |
| Hox4R | GTTAAATCGGCGGGATGTAG |
| Hox5F | CTACTTCGTCAATTCACTGTCTACA |
| Hox5R | TTCTTTCTTCCATTTCATTCGG |
| AntpF | AGTTTTGGCAGCGCAGAATT |
| AntpR | TTTCTTTTTTCCACTTCATCCG |
| Lox2F | CGTTATAGGGAAACTTGTCCG |
| Lox2R | TATTCGTCTTCGTCTCGTGAG |
| Lox4F | TTATCCAGCGTCGTTTTTC |
| Lox4R | CATGTTCGCCACTATTTCC |
| Lox5F | CACCAGCATCACCAACTCCC |
| Lox5R | ATTTCTATTCGCCTTCGTCTC |
| Post1F | GTATGCCTTTCGTGGGTGG |
| Post1R | TGCTGCTTTGCTGTTTCCG |
| Post2F | TCCTCTCAATCCTATGAACCA |
| Post2R | GACACCGAACTCCGACAATCT |
| EF1aF | CACCACGAGTCTCTCCCTGA |
| EF1aR | GCTGTCACCACAGACCATTCC |
利用已知的软体动物Hox基因, 从厚壳贻贝全基因组数据中筛选出全部含有homeodomain同源保守区的序列, 共101条。通过与其他软体动物的Hox基因序列进行比对, 鉴定出厚壳贻贝中的11条Hox序列(表 3)。按照软体动物Hox基因命名的方式, 将11条厚壳贻贝Hox基因序列命名为Hox1、Hox2、Hox3、Hox4、Hox5、Antp、Lox2、Lox4、Lox5、Post1和Post2。对Hox蛋白理化性质分析发现, CDs的长度范围在573~2 112 bp之间。其中序列长度最长的是Hox2, 编码703个氨基酸, 最短的是Post2, 编码190个氨基酸。对应的蛋白分子质量在21.9~77.9 kDa之间。11个Hox蛋白的GRAVY疏水性值都为负值, 说明Hox对应的蛋白都是亲水蛋白, pI值的范围在5.11~10.22之间, 除了Hox2为偏酸性蛋白, 其他Hox蛋白都是偏碱性蛋白。
| 基因名称 | 氨基酸数 | 亲水性 | 分子质量 | 等电点pI | GenBank登录号 |
| Hox1 | 304 | –0.827 | 33.86 | 8.35 | OM037102 |
| Hox2 | 703 | –0.917 | 77.90 | 5.11 | OM037101 |
| Hox3 | 423 | –1.208 | 47.81 | 7.76 | OM037100 |
| Hox4 | 254 | –1.075 | 29.12 | 9.45 | OM037099 |
| Hox5 | 274 | –1.157 | 31.55 | 9.85 | OM037098 |
| Antp | 225 | –1.243 | 26.85 | 8.93 | OM037097 |
| Lox2 | 234 | –1.036 | 27.26 | 9.74 | OM037105 |
| Lox4 | 271 | –1.214 | 31.12 | 8.16 | OM037106 |
| Lox5 | 295 | –1.052 | 34.09 | 8.33 | OM037107 |
| Post1 | 248 | –0.920 | 28.59 | 9.59 | OM037103 |
| Post2 | 190 | –0.958 | 21.90 | 10.22 | OM037104 |
探究厚壳贻贝中鉴定出的Hox基因的系统发育关系, 利用最大似然法对从厚壳贻贝全基因组数据中筛选出全部含有homeodomain同源保守区的序列的101条蛋白序列进行系统发育分析(图 1)。依据软体动物Hox基因家族分类标准, 将101条序列分属为四类, 同样用红、蓝、绿、紫四种颜色来区分。同时聚类分支的节点自展值都在0.5以上, 说明聚类结果存在一定的可信度。为了进一步探究Hox基因的系统发育关系, 从NCBI数据库中下载了所有已上传的软体动物的Hox基因蛋白序列, 并利用最大似然法对厚壳贻贝Hox基因蛋白序列和其他软体动物Hox基因蛋白序列进行系统发育分析(图 2)。Hox基因家族不同成员分别聚为一支, 表明Hox家族成员在物种间的保守性高于在家族成员间的保守性, 支持了上述序列分析对Hox基因的分类。同时发现, 在Hox基因簇上的位置靠近的基因在聚类树上的关系更相近, 基因簇上位置较远的基因在聚类上关系也更远。比如Post1和Post2聚为一支, Hox4、Antp、Lox5、Lox2和Lox4聚为一个大类, Hox1、Hox2和Hox3各自聚为一类, 此外, Hox5单独聚为一类。然而不同的是, 各个物种的Antp基因虽然在系统进化树中亲缘关系较近, 但并不像其他基因一样聚为一支。此外, 在进化树中, 发现软体动物Hox基因序列并非完全严格按照腹足纲、双壳纲、头足纲等来进行聚类, 存在一定程度的混乱。例如Hox1中的厚壳贻贝并未和双壳纲的其他物种聚为一支, 同样在其他基因, 比如Hox2、Hox4、Hox5中, 又各自聚为一支。这样的结果提示Hox基因家族成员在软体动物中的进化速率是不一样的。通过以上结果, 也能确定鉴定出11个Hox基因均属直系同源基因。
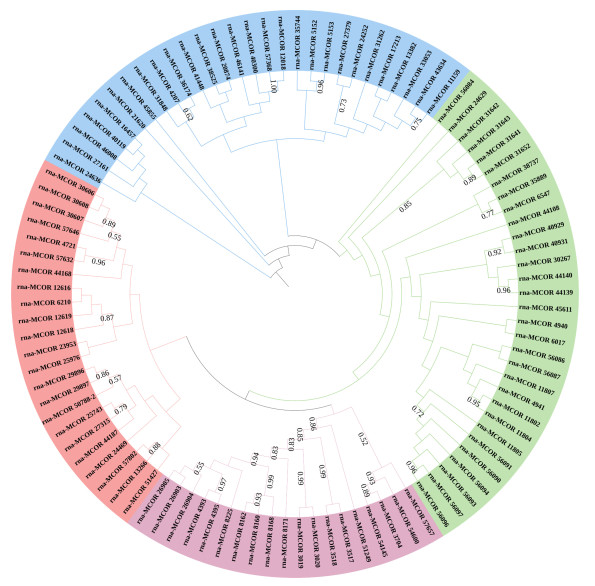 |
| 图 1 厚壳贻贝101条序列的ML系统进化树 Fig. 1 ML phylogenetic tree of M. coruscus based on 101 sequences 注: 不同颜色代表不同的亚家族 |
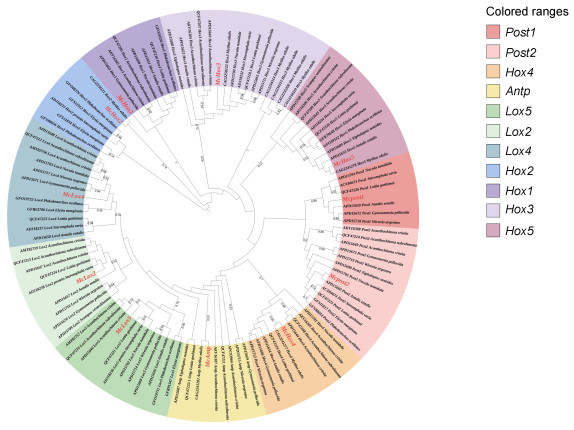 |
| 图 2 基于NCBI上软体动物11条Hox序列的系统发育树 Fig. 2 Phylogenetic tree based on 11 Hox sequences in mollusk in NCBI 注: 不同的颜色代表不同Hox基因的亚家族, 11个厚壳贻贝的Hox基因被标记为红色 |
结果如图 3所示, 11个Hox基因分布在一个染色体片段中, 形成一个基因簇。按5'到3'的顺序, Hox排列顺序为Hox1、Hox2、Hox3、Hox4、Hox5、Lox5、Antp、Lox4, Lox2、Post2和Post1。此外, 观察各个Hox基因之间的距离, 可以看到11个Hox基因可以被分为四簇: Hox1、Hox2和Hox3为一簇(S1), Hox4和Hox5为一簇(S2), Lox5、Antp、Lox4和Lox2为一簇(S3), Post1和Post2为一簇(S4), 并且我们分别用红、橙、黄、紫四个颜色进行区分。
 |
| 图 3 厚壳贻贝11条Hox序列在染色体上的位置 Fig. 3 Chromosomal positions of 11 Hox sequences in M. coruscus 注: 红色: S1; 橙色: S2; 黄色: S3; 紫色: S4 |
将厚壳贻贝11条Hox基因序列的Motif结构和系统发育关系结合起来进行分析(图 4)。Hox4、Hox5、Lox5和Antp在系统发育树中聚为一支, 另一支由Lox2和Lox4组成, 暗示着他们之间的结构相似性或功能上的相关性。在Motif组成上, 在聚为同一簇的Hox基因中看到一定的保守性, 比如Lox2和Lox4的Motif在组成、位置、长度上基本一致。Motif1位于同源结构域, 在所有Hox基因中均存在。然而个别基因仍存在独特的Motif元件。比如, Motif8只存在于Hox1和Hox4; Motif10是Hox3和Antp所特有的; Motif5是Hox2和Post1特有的。
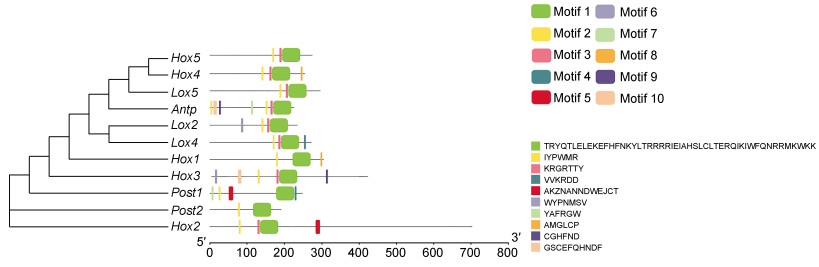 |
| 图 4 厚壳贻贝11条Hox序列的系统发育关系与Motif结构 Fig. 4 Phylogenetic relationship and Motif structures of 11 Hox sequences in M. coruscus |
荧光定量PCR检测的11个Hox基因在厚壳贻六个组织中的表达(图 5, 图 6)。Hox1主要在性腺、外套膜和足三个组织中存在一定量表达, 其中在性腺中的表达水平要显著高于其他组织; 在鳃、闭壳肌和消化腺三个组织中尚未检测到表达或存在微量表达; Hox2在外套膜组织中的表达要显著高于其他组织, 在鳃、性腺等组织中虽然存在一定量的表达, 但都比较低; Hox3主要在鳃、性腺、外套膜和消化腺中存在表达, 在外套膜中的表达要显著高于其他组织, 在足和闭壳肌中不表达或存在微量表达; Hox4在性腺组织中的表达水平相对最高, 其次是鳃和外套膜, 这两个组织的表达水平持平, 而在闭壳肌和消化腺中微量表达, 在足中未检测到表达; Hox5在除了足中的其他五个组织中都存在一定量表达, 其中在鳃组织的表达量最高, 其次是外套膜和性腺; Antp在六个组织中皆检测到一定水平的表达, 其中主要在鳃、外套膜、消化腺中的表达较高, 而在这三个组织中表达量水平最高的为消化腺组织; Lox2在消化腺水平最高, 其次是闭壳肌和外套膜; Lox4在六个组织中都存在一定量表达, 在闭壳肌中表达最高, 其次是消化腺, 在鳃和外套膜中的表达基本持平, 在足中的表达量最低; Lox5只在性腺、外套膜、足三个组织中检测到了表达, 在鳃、闭壳肌、消化腺三个组织中并未检测到表达, 而在表达的三个组织中, 外套膜和足的表达量较高, 在性腺中的表达量较低; Post1在六个组织中都存在表达, 其中在闭壳肌中表达量最高, 其次是在消化腺中存在较高表达量, 在其他四个组织中表达都较低; Post2与Post1的表达模式类似, 在闭壳肌中表达量最高, 其次在消化腺中也有一定表达量。略微不同的是, Post2的整体表达水平比Post1略低一点, 此外Post2在鳃中的表达要稍微高于Post1的表达水平。从组织角度上来看表达量, 可以总结为: 在鳃中Lox2、Lox4和Lox5的表达量远超其他基因的表达, 且这三个基因表达水平持平; 在性腺中, Hox4高量表达, 同时伴随Hox5一定水平的表达; 在外套膜中, Hox2、Hox3、Hox4和Hox5的表达量都很高, 其中Hox4表达水平最高; 在闭壳肌中, Post1、Post2和Lox4都有较高的表达量, 其中Post1最高; 在足中Lox4的表达水平很高; 而在消化腺中, 几乎大部分Hox基因都有参与, 而且都有较高的表达水平。值得注意的是, Post1和Post2的表达水平相似, Lox2和Lox4的表达水平相似, Hox4和Hox5的表达水平相似, Hox2和Hox3的表达水平相似。
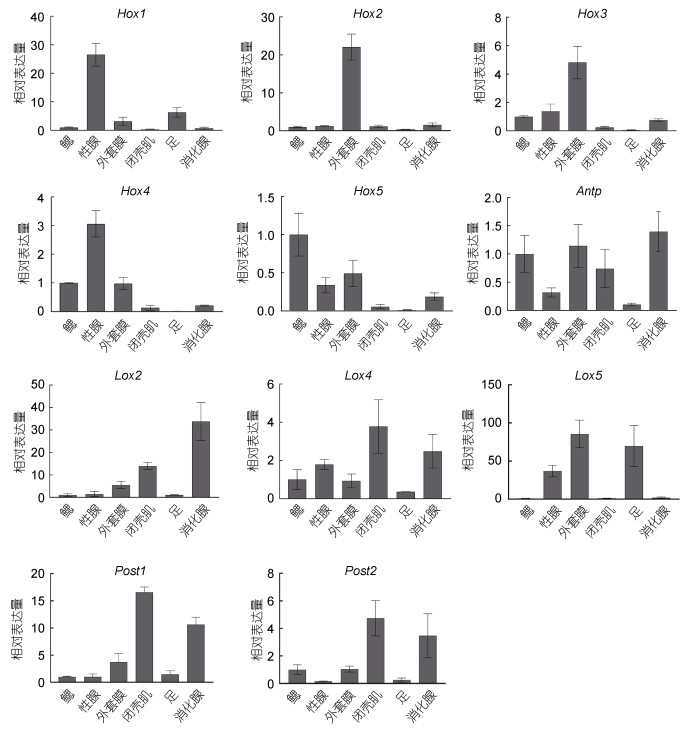 |
| 图 5 厚壳贻贝11条Hox基因在六个成体组织的表达量 Fig. 5 Expressions of 11 Hox genes at six adult tissues in M. coruscus |
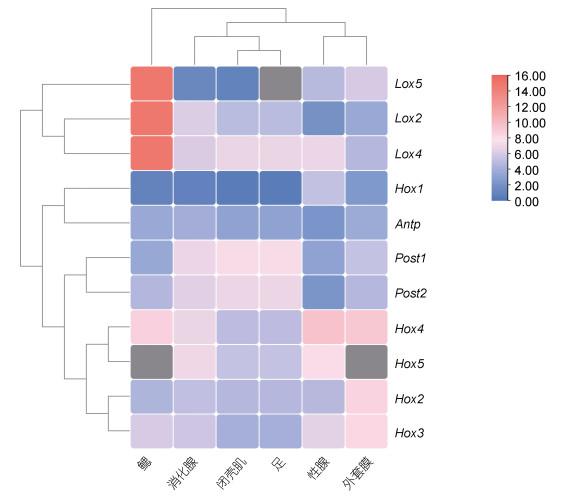 |
| 图 6 厚壳贻贝不同成体组织Hox表达谱 Fig. 6 The Hox genes expression profiles of M. coruscus in different adult tissues |
目前在双壳纲贝类中, 已经有部分物种Hox基因被鉴定出来, 例如长牡蛎, 虾夷扇贝等。王师等(2017)根据其团队研究结果, 认为双壳纲贝类普遍含有一个完整的具有11个Hox基因的基因簇。在贻贝科物种中, 紫贻贝的四个Hox基因通过扩增技术被挖掘(Pérez-Parallé et al, 2005), 然而这并非是在基因组水平上进行挖掘, 挖掘的Hox基因数量有限。Sun等(2017)对深海偏顶蛤(Bathymodiolus platifrons)和菲氏偏顶蛤(Modiolus philippinarum)的基因组信息进行了更加深层次的研究, 而厚壳贻贝基因组信息缺乏。针对这101条序列进行系统发育分析, 得到的进化树将这101条序列大致分为四个分支。分支内的序列表明了各自之间序列以及功能的相似性, 包括亲缘关系。早先Brooke等(1998)对双侧体Hox基因的分子系统发育和基因组组织分析表明, 无脊椎动物的单倍体基因组中有一个Hox簇, 在所有动物门中, Hox簇由四个基因群组成。因此, 本研究中对厚壳贻贝Hox序列系统发育分析得到的四类分支结果并非偶然。从功能上来看, 聚为一类的序列往往在生物体内行使相同的功能或者调控相似的通路, 因此, 聚为四类的厚壳贻贝Hox序列在厚壳贻贝发育过程中可能对应调控着四个不同的区块或者不同的功能。而在用软体动物Hox基因进一步进行系统发育分析时发现其中部分基因聚类为一个大类, 例如Lox2和Lox4, 或Post1和Post2。我们推测这可能与基因来源有关, Lox2和Lox4基因被认为是从一个祖先复制而来的(Balavoine et al, 2002)。它们在头足类、腹足类、双壳类和腕足类中的存在可能表明这种重复发生在Lophotrochozoa和其他原口动物之间的分化之前(Canapa et al, 2005)。此外, 系统发育树结果显示, Antp相比于其他Hox基因虽然亲缘关系相近然而并未聚为一支, 这暗示着, 在不同物种中, Antp的进化速率可能出现了差异, 相比于其他Hox基因, Antp基因序列中的变化要显得更快。
Hox基因主要作为生物体发育过程中体轴形成以及神经器官发育相关的重要调控基因被研究。然而Hox基因不止在发育过程中起作用, 当前研究表明, Hox基因在成体中仍存在重要的功能, 例如, 在各种肿瘤及泌尿系统疾病患者中Hox基因的表达存在异常。Crow等(2006)等通过Northern印迹等方法研究发现, HoxA10基因存在低分化, 非正常的子宫内膜腺癌中的表达明显高于在正常的、增生的、高分化的子宫内膜中的表达。此外, 在急性髓系白血病患者体内, 研究发现Hox基因在白血病细胞内广泛表达, 并且Hox基因通过与某些转录因子联合造成疾病的发生(蒋琼等, 2013)。软体动物在进化史上占据重要的地位, 除了研究Hox在软体动物发育过程中所起到的作用, 对软体动物各个成体组织中Hox功能的研究, 也能为进一步了解Hox基因的功能以及与相关细胞活动或疾病的发生奠定相关理论基础。然而当前Hox基因在软体动物成体组织中的表达模式尚未有太多研究, 因此, 本研究通过荧光定量PCR技术探究Hox基因在厚壳贻贝鳃、性腺、外套膜、闭壳肌、足、消化腺六个组织中的表达模式, 以探究Hox基因在成体组织中可能起到的功能和作用。Hox基因在发育时期表达的特定模式, 决定了相关细胞的命运和分化。虽然在幼体发育过程中的功能十分重要, 但Hox基因在成体中的调控以及表达模式也不可忽视。
我们猜测高表达量的Hox基因在不同组织中可能行使的功能。Hox基因在持续更新的器官系统中一般表达更高, 例如造血干细胞、血管重塑过程中的内皮细胞、子宫内膜细胞、成纤维细胞和骨骼干细胞。鳃作为双壳贝类的主要呼吸器官, 每一鳃片鳃轴的两侧或一侧生有鳃丝, 鳃上生有纤毛。依纤毛的运动使呼吸水流按一定线路通过鳃进行气体交换, 同时包括一些滤食的行为(王芳等, 1998)。我们认为, 厚壳贻贝鳃中高表达的Lox2、Lox4和Lox5可能是与鳃上纤毛相关细胞的生长, 分化以及其他调控有关, 鳃上纤毛细胞的更迭保证了气体交换的效率。然而当前Lox2、Lox4和Lox5主要被认为与神经形成有关, 是否与双壳纲鳃部纤毛细胞的调控有关, 仍需要进一步的探究。Lox4基因不仅在鳃中高表达, 同样的还有足中。贝类的神经系统由脑、足、侧、脏4对神经节和与其联络的神经构成, 足神经节位于足的前部(杨金龙等, 2013)。我们的结果与当前对Lox类基因的结论一致, Lox4在足中的高表达可能与厚壳贻贝足神经的发育调控有关。Hox4在性腺中的高表达, 我们推测, Hox4可能与厚壳贻贝性腺发育有关。然而不同的是, 在凡滨纳对虾中, 精巢和卵巢几乎涵盖了所有的Hox基因, 这可能是与不同物种生殖细胞特性有关(Krumlauf et al, 1993; 孙晓晴, 2015)。贝类的外套膜是一个与生物矿化相关的组织, 通过许多贝壳有机质的分泌和离子的转运, 调控CaCO3晶体沉淀形成贝壳。Irvine等(2000)发现Hox1和Hox4在多毛纲动物的外套膜中表达, 在本文中, Hox2、Hox3和Hox5以Hox4“为首”在外套膜中展现出高水平表达, 表明这四个Hox基因可能在贝壳形成方面具有重要作用。我们推测消化腺中普遍表达的Hox基因可能与Hox的协同调控某种机制相关。早先有相关研究表明, Hox基因在幼体发育时期的版本可以通过表观遗传机制保留在成体内, 其中, Hox基因通过表达或抑制表达, 来维持或决定细胞身份以及所需要的功能, 即成体中Hox基因的区域表达模式通常反映了胚胎的表达模式(Pai et al, 2020)。我们发现, 每个组织中都有一个或多个“负责”的, 可以按表达量区分的Hox亚簇, 例如鳃中的Lox2、Lox4和Lox5, 外套膜中的Hox2、Hox3、Hox4和Hox5, 同时在表达量上, Lox4总是要高于Lox2和Lox5, Hox4总是要高于Hox5, 其他组织也具有相似的规律。因此我们推测, Hox基因在成体组织中也具有一定水平的保留, 这样的保留使得相关器官或神经在发育阶段和成体阶段形成位置上的同一性。
4 结论Hox作为调控生物体轴与发育的一类重要的因子, 其多样化的表达为生物体形态多样性提供了重要的分子基础。因此, 通过本课题组以及NCBI数据库中获取的厚壳贻贝全基因组数据进行挖掘, 并从调控元件组成、染色体位置、系统发育关系以及六个成体组织表达谱上, 对厚壳贻贝Hox进行初步的探究。厚壳贻贝Hox在种内的聚类为4支, 而在整个软体动物水平上聚类为10支。我们推测这可能与双壳纲贝类Hox功能分化有关。而Hox基因在不同成体组织表现出多样性的表达, 在不同纲物种中表现出不同的功能, 在成体特定组织中也具有不同的功能。总之, 本研究为进一步探究厚壳贻贝Hox基因家族功能奠定基础, 也为其他软体动物Hox研究提供参考数据, 但具体影响机制仍需进一步研究。
王师, 杨志辉, 李语丽, 等, 2017. Hox基因及其在软体动物中的研究进展. 中国海洋大学学报, 47(8): 23-30 |
王芳, 董双林, 范瑞青, 等, 1998. 四种滤食性贝类滤食器官鳃的扫描电镜观察. 青岛海洋大学学报, 28(2): 240-244 |
龙治海, 陈虎, 汪斌, 等, 2018. 达氏鲟内参基因β-actin、GAPDH和EF1-α的克隆及表达稳定性. 农业生物技术学报, 26(11): 1846-1855 |
孙晓晴, 2015. 凡纳滨对虾Hox基因结构和表达的初步研究[D]. 青岛: 中国科学院研究生院(海洋研究所): 15.
|
牟政强, 2015. 菲律宾蛤仔不同发育时期转录组及Hox家族表达分析[D]. 大连: 大连海洋大学: 28-32.
|
杨金龙, 李树恒, 刘志伟, 等, 2013. 厚壳贻贝胚胎和早期幼虫神经系统发育的初步研究. 水产学报, 37(4): 512-519 |
陈远, 2013. 鲸类Hox基因的适应性进化及与体轴发育的关系[D]. 南京: 南京师范大学: 7-9.
|
蒋琼, 刘文君, 2013. Hox基因与急性髓系白血病相关性的研究进展. 中国实验血液学杂志, 21(5): 1340-1344 |
BALAVOINE G, DE ROSA R, ADOUTTE A, 2002. Hox clusters and bilaterian phylogeny. Molecular Phylogenetics and Evolution, 24(3): 366-373 DOI:10.1016/S1055-7903(02)00237-3 |
BROOKE N M, GARCIA-FERNÀNDEZ J, HOLLAND P W H, 1998. The ParaHox gene cluster is an evolutionary sister of the Hox gene cluster. Nature, 392(6679): 920-922 DOI:10.1038/31933 |
BÜRGLIN T R, 2011. Homeodomain subtypes and functional diversity [M] // HUGHES T R. A Handbook of Transcription Factors. Dordrecht: Springer: 95-122.
|
CALLAERTS P, LEE P N, HARTMANN B, et al, 2002. Hox genes in the sepiolid squid Euprymna scolopes: implications for the evolution of complex body plans. Proceedings of the National Academy of Sciences of the United States of America, 99(4): 2088-2093 DOI:10.1073/pnas.042683899 |
CANAPA A, BISCOTTI M A, OLMO E, et al, 2005. Isolation of Hox and ParaHox genes in the bivalve Pecten maximus. Gene, 348: 83-88 DOI:10.1016/j.gene.2004.12.039 |
CARPINTERO P, PAZOS A J, ABAD M, et al, 2004. Presence of proboscipedia and caudal gene homologues in a bivalve mollusc. Journal of Biochemistry and Molecular Biology, 37(5): 625-628 |
CROW K D, STADLER P F, LYNCH V J, et al, 2006. The "fish-specific" Hox cluster duplication is coincident with the origin of teleosts. Molecular Biology and Evolution, 23(1): 121-136 DOI:10.1093/molbev/msj020 |
DE ROSA R, GRENIER J K, ANDREEVA T, et al, 1999. Hox genes in brachiopods and priapulids and protostome evolution. Nature, 399(6738): 772-776 DOI:10.1038/21631 |
EKSTROM J A, SUATONI L, COOLEY S R, et al, 2015. Vulnerability and adaptation of US shellfisheries to ocean acidification. Nature Climate Change, 5(3): 207-214 DOI:10.1038/nclimate2508 |
GARCIA-FERNÀNDEZ J, 2005. The genesis and evolution of homeobox gene clusters. Nature Reviews Genetics, 6(12): 881-892 DOI:10.1038/nrg1723 |
HE B, NI Z L, KONG S B, et al, 2018. Homeobox genes for embryo implantation: from mouse to human. Animal Models and Experimental Medicine, 1(1): 14-22 DOI:10.1002/ame2.12002 |
IIJIMA M, AKIBA N, SARASHINA I, et al, 2006. Evolution of Hox genes in molluscs: a comparison among seven morphologically diverse classes. Journal of Molluscan Studies, 72(3): 259-266 DOI:10.1093/mollus/eyl001 |
IRVINE S Q, MARTINDALE M Q, 2000. Expression patterns of anterior Hox genes in the polychaete Chaetopterus: correlation with morphological boundaries. Developmental Biology, 217(2): 333-351 DOI:10.1006/dbio.1999.9541 |
KRUMLAUF R, MARSHALL H, STUDER M, et al, 1993. Hox homeobox genes and regionalisation of the nervous system. Journal of Neurobiology, 24(10): 1328-1340 DOI:10.1002/neu.480241006 |
PAI P, SUKUMAR S, 2020. Hox genes and the NF-κB pathway: a convergence of developmental biology, inflammation and cancer biology. Biochimica et Biophysica Acta (BBA) - Reviews on Cancer, 1874(2): 188450 DOI:10.1016/j.bbcan.2020.188450 |
PEARSON J C, LEMONS D, MCGINNIS W, 2005. Modulating Hox gene functions during animal body patterning. Nature Reviews Genetics, 6(12): 893-904 DOI:10.1038/nrg1726 |
PÉREZ-PARALLÉ M L, CARPINTERO P, PAZOS A J, et al, 2005. The Hox gene cluster in the bivalve mollusc Mytilus galloprovincialis. Biochemical Genetics, 43(7): 417-424 |
SAMADI L, STEINER G, 2009. Involvement of Hox genes in shell morphogenesis in the encapsulated development of a top shell gastropod (Gibbula varia L.). Development Genes and Evolution, 219(9): 523-530 |
SUN J, ZHANG Y, XU T, et al, 2017. Adaptation to deep-sea chemosynthetic environments as revealed by mussel genomes. Nature Ecology & Evolution, 1(5): 0121 |
TAMURA K, PETERSON D, PETERSON N, et al, 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 28(10): 2731-2739 |
WINNEPENNINCKX B, BACKELJAU T, DE WACHTER R, 1996. Investigation of molluscan phylogeny on the basis of 18S rRNA sequences. Molecular Biology and Evolution, 13(10): 1306-1317 |
 2023, Vol. 54
2023, Vol. 54


