
中国海洋湖沼学会主办。
文章信息
- 范孝俊, 王莹, 蔡吕萍, 黄键锋, 刘菲, 李迎澳, 张晓林, 严小军, 廖智. 2023.
- FAN Xiao-Jun, WANG Ying, CAI Lyu-Ping, HUANG Jian-Feng, LIU Fei, LI Ying-Ao, ZHANG Xiao-Lin, YAN Xiao-Jun, LIAO Zhi. 2023.
- 海水酸化对厚壳贻贝(Mytilus coruscus) 外套膜代谢组的急性影响
- EFFECTS OF ACUTE SEAWATER ACIDIFICATION ON METABOLISM OF MANTLE IN MYTILUS CORUSCUS
- 海洋与湖沼, 54(3): 921-933
- Oceanologia et Limnologia Sinica, 54(3): 921-933.
- http://dx.doi.org/10.11693/hyhz20221000276
文章历史
-
收稿日期:2022-10-25
收修改稿日期:2022-12-05
地球目前面临的两大环境问题, 温室效应和海洋酸化, 均与大气中二氧化碳浓度升高有关(Tan et al, 2020b)。大气二氧化碳浓度已从工业革命时期的280 ×10–6增加到目前的近400 ×10–6, 不仅推动了温室效应, 也因此造成海水平均pH的持续降低。Queirós等(2015)预测全球海洋pH将在2300年下降至7.4。此外, 河流入海口的冲淡水也会导致局部海域的pH改变。已有数据表明, 长江口、杭州湾和三门湾pH会出现季节性变化, 且在夏季出现7.75的低值(刘晓辉等, 2017; Zuo et al, 2022)。以上结果表明, 海洋生物面临着长期或者短期波动性的海洋酸化威胁, 因此, 研究海洋生物在酸化条件下其生理生化特征的变化, 将有助于了解海洋生物对酸化的短期胁迫和长期适应过程。
目前已知海洋酸化会造成海洋生物多种生理异常, 包括能量分配紊乱、免疫力下降、新陈代谢同化效率降低、呼吸能提高以及细胞内活性氧增加等(Cao et al, 2018)。此外, 海洋酸化会降低海水中碳酸钙的饱和度, 对以碳酸钙为生物硬组织的部分海洋生物, 如贝类、棘皮动物、珊瑚、藤壶等造成严重的生存威胁(Doney et al, 2009; Ashur et al, 2017; Doo et al, 2019)。例如, 海洋酸化已造成局部海域珊瑚虫的大面积死亡(Eyre et al, 2018); 此外, 海洋酸化对双壳纲软体动物也产生不利影响, 包括幼虫存活率下降(Tan et al, 2020a), 疾病感染率增加(Asplund et al, 2014; Cao et al, 2018), 贝壳生长和发育受阻(Melzner et al, 2009), 足丝强度下降(Zhao et al, 2017)以及贝壳溶解(Chandra et al, 2021)。目前, 对海洋生物在酸化条件下的生理响应, 多以分子组学为研究手段。例如, Goncalves等(2017)利用蛋白质组学和转录组学手段研究了牡蛎(Saccostrea glomerata)鳃组织在酸化条件下的分子响应, 发现包括热休克蛋白, 免疫相关蛋白以及能量代谢相关蛋白及基因表达量等受酸化影响较大。Carroll等(2021)利用蛋白质组学手段研究了南非鲍(Haliotis midae)血细胞在酸化条件下的分子变化, 结果表明细胞骨架、免疫、蛋白质翻译及翻译后修饰、囊泡运输以及能量代谢相关蛋白质含量受酸化影响较大。以上研究结果表明, 采用组学手段研究海洋酸化条件下海洋生物的生理生化响应, 可以了解酸化背景下海洋生物的分子应对策略。
贝类是一类具有重要经济价值的海洋生物, 在当前水产养殖业中所占比重较大。其贝壳是生物矿化的产物, 主要分泌组织为外套膜。由于贝壳的主要成分(碳酸钙晶体)对海洋酸化呈现较强的敏感性, 海洋酸化对贝类, 特别是贝类养殖业已带来巨大影响(Tan et al, 2020a), 因此对贝类生物矿化过程在海洋酸化条件下的相关研究也日益受到人们的重视。以往研究已发现, 海洋酸化会破坏贝类体内的pH稳态, 从而影响外套膜组织中生物矿化关键基因的表达(Wei et al, 2015)。此外, 酸化海水还会导致成年牡蛎的三羧酸循环过程被抑制而导致能量代谢受阻, 或者抑制牡蛎幼虫的糖酵解、脂肪酸氧化等内源性能量供应, 因此导致牡蛎不论是在成年期还是在幼年期, 其生物矿化过程均因能量供应不足而受到抑制(Timmins-Schiffman et al, 2014; Wang et al, 2018; Jiang et al, 2019)。目前, 针对贝类在海洋酸化条件下的生理响应多集中在牡蛎为代表的少数贝类物种(Liu et al, 2020), 目前尚缺乏针对贻贝在酸化条件下, 其生物矿化相关组织的生理响应研究。
厚壳贻贝(Mytilus coruscus)是我国东部海域的主要双壳贝类之一。以往的研究表明, 贻贝属贝类对海洋酸化具有较强的耐受性, 因而导致贻贝属往往成为某些高CO2海域的优势物种(Sunday et al, 2011; Thomsen et al, 2013, 2017; Sui et al, 2017), 但相关机制目前尚不明确。此前研究已发现, 厚壳贻贝体内含有较高浓度的尿素, 且在厚壳贻贝体内注射13C标记尿素后, 其贝壳中13C/12C比值显著上升(王莹等, 2022), 这意味着尿素分子可能在其对抗海洋酸化过程中起到了重要作用。但目前尚无关于贻贝在酸化条件下, 其外套膜组织中代谢物组学分析。代谢组学研究可以直观地展示代谢物组成与变化, 因此可以从代谢物水平了解贝类在海洋酸化条件下的新陈代谢过程变化, 并且从中发现贻贝对海洋酸化的分子响应机制。为此, 以我国东部海域重要的经济贝类厚壳贻贝为研究对象, 对其外套膜组织在急性酸化刺激条件下代谢物的组成和含量变化开展分析, 以探讨贻贝在海洋酸化过程中的生理响应以及贻贝应对海洋酸化的可能分子机制。
1 材料与方法 1.1 厚壳贻贝的采集与样品处理成年厚壳贻贝采集自浙江舟山嵊泗海域, 以洁净海水暂养于恒温水族箱中(温度22 ℃, 盐度25)。对照组贻贝饲养于正常海水(pH 8.1)中, 实验组贻贝饲养于酸化海水(pH 7.3)中。酸化海水参照文献方法(Yarra et al, 2021), 采用稀盐酸进行调配, 每2 h调节一次pH, 使其稳定在7.3。以上两组贻贝在饲养过程中均采用螺旋藻粉作为饲料定时投喂。饲养时间均为48 h。
1.2 外套膜组织样本的采集与代谢物的提取选取厚壳贻贝外套膜组织进行代谢物提取。所有提取试剂使用前均在–20 ℃进行预冷。组织样本中加入内标(L-2-氯苯丙氨酸, 0.06 mg/mL; 甲醇配置)及甲醇-水(体积比为4︰1)溶液, 经研磨(60 Hz, 2 min)后离心(10 000×g, 4 ℃, 10 min), 取上清液冷冻干燥。再以甲醇-水(体积比为1︰4)溶液复溶后, –20 ℃下静置2 h, 经离心(10 000×g, 4 ℃, 10 min)后取上清液, 经0.22 μm滤膜过滤后, 上样超高液相色谱-串联质谱(ACQUITY UPLC I-Class plus, 美国沃特世公司)进行分析, 采用6次重复试验, 质控样品由所有样本等体积混合制备。
1.3 超高液相色谱-串联质谱(UPLC-MS/MS)分析采用HSS T3 (100×2.1 mm, 粒径1.8 μm)反相色谱柱进行代谢物分离, 流动相分别为A液(含0.1%甲酸的水)和B液(含0.1%甲酸的乙腈); 洗脱梯度为12 min内B液比例从5%上升至100%; 流速为0.35 mL/min, 柱温为45 ℃。
色谱柱洗脱组份注入UPLC-MS/MS, 以正负离子模式进行质谱扫描分析。采用全扫描结合全信息串联(MSE)模式进行质谱数据采集; 质荷比(m/z)范围设置为100~1 200; 分别采用低能扫描(Ce, 4 eV)和高能扫描(CE-ramp, 20~45 eV)方式破碎母离子。采用氩气(99.999%)为碰撞诱导离解气体。质谱扫描时间0.2 s; 延迟0.02 s; 毛细管电压2.5 kV; 锥电压40 V; 源温度320 ℃; 脱溶剂气体温度350 ℃; 去溶剂气体流量900 L/h。分析过程中以固定间隔注入质控样品(QC)评估质谱检测的重复性。
原始质谱数据经Progenesis QI v2.3软件(Nonlinear Dynamics, Newcastle, 英国)进行基线过滤、峰识别、积分、保留时间校正、峰对齐和归一化处理。代谢物鉴定基于质谱所得精确分子质量、二级质谱碎片峰以及同位素分布, 分别采用The Human Metabolome Database (HMDB)、Lipid maps (v2.3)和METLIN数据库进行代谢物鉴定。采用单变量分析差异倍数(fold-change)和T检验P-value (n=6)来筛选差异表达代谢物。筛选条件为差异倍数 > 1.20或 < 0.83以及P-value < 0.05, 二者取交集, 获得差异离子。差异代谢物注释以及代谢物通路分析基于KEGG数据库进行。
1.4 贻贝外套膜游离氨基酸组成分析贻贝外套膜组织经液氮研磨后行组织匀浆; 匀浆后的组织样本进行超声破碎(功率150 W, 超声时长3 s, 间隔10 s, 总时长5 min), 再经离心(4 000×g, 4 ℃, 10 min)后, 以截留分子量为3 K的超滤管进行超滤, 滤过液经冷冻干燥后, 以0.02 mol/L稀盐酸进行复溶, 经0.22 μm滤膜过滤后, 上样全自动微量氨基酸分析仪(LA8080A, 日立公司), 参照王春月等人的方法(Wang et al, 2021)进行分析。采用离子交换柱(铝离子交换树脂, 4.6 mm×60.0 mm)进行样品分离; 反应柱为热传导膜不锈钢柱4.6 mm×(42.5 mm+ 42.5 mm); 交换柱温度设置为50 ℃, 反应柱温度设置为135 ℃; 缓冲液流速0.4 mL/min; 茚三酮流速0.35 mL/min。检测波长570 nm, 检测时间150 min。氨基酸标准品为日本日立公司标准氨基酸混合液(B型和AN-II型)。
1.5 外套膜酶活力测定贻贝外套膜组织经液氮研磨、匀浆、离心后取上清液进行总蛋白定量和酶活力测定。其中, 超氧化物歧化酶(superoxide dismutase, SOD), 过氧化氢酶(catalase, CAT), 谷胱甘肽(glutathione, GSH), 5′-核苷酸酶(5′-nucleotidase, 5′-NT), 总抗氧化能力(total antioxidant capacity, T-AOC), 总一氧化氮合酶(total antioxidant capacity, T-NOS)的测定采用试剂盒(南京建成)并按说明书方法进行。总蛋白浓度采用Bicinchoninic Acid (BCA)标准方法测定。
1.6 数据处理和统计分析使用软件Progenesis QI (version 2.2, Waters, UK)和R软件包meta X对质谱数据进行统计分析。单变量分析采用Wilcoxon和T检验来确定实验组和对照组之间的代谢物差异; 多变量统计分析采用柏拉图标准化(Pareto scaling)对数据进行log2转换和比例调整后, 分别进行主成分分析(principal component analysis, PCA)、偏最小二乘-判别分析(partial least squares discrimination analysis, PLS-DA)和正交偏最小二乘方-判别分析(orthogonal partial least squares discrimination analysis, OPLS-DA)。
采用IBM SPSS Statistics 26的单因素方差分析(One-way ANOVA)进行酶活力及游离氨基酸的组间显著性差异分析, 显著差异水平为P < 0.05。
2 结果 2.1 多元统计分析结果对正常海水饲养的对照组贻贝和酸化海水饲喂的实验组贻贝的外套膜组织, 分别进行非靶向UPLC-MS/MS代谢组学分析, 采取6组平行实验。在正离子和负离子检测模式下, 去除相对标准偏差(relative standard deviation, RSD) > 0.3的离子后, 总计获得质谱峰数量为17 366个, 其中正离子模式下为6 779个, 负离子模式下为10 587个; 鉴定代谢物总计7 882种, 其中正离子模式下鉴定3 398种, 负离子模式下鉴定4 484种。所有代谢组学分析的质谱原始数据已上传至Metabolights数据库, 编号为MTBLS5530。
通过PCA分析判断样本之间代谢物的总体分布特征及分析的稳定性; 采用PLS-DA以及OPLS-DA分析评估对照组和实验组之间的统计学差异, 结果见图 1。由图 1可见, 对照组和实验组在数据点的空间分布上存在明显分离(图 1a), 且所有样本处于95%置信区间, 表明酸化海水条件下, 贻贝外套膜组织代谢物与对照组相比具有明显差异。其中, PLS-DA (图 1b)及OPLS-DA (图 1c)分析所得R2X (模型X轴方向累计解释率)和R2Y (模型Y轴方向累计解释率)分别为0.747和0.992; PLS-DA及OPLS-DA分析Q2值(模型的累积预测率)分别为0.936和0.859 (图 1d)。上述数据表明模型预测能力及稳健性良好, 无过度拟合现象, 可用于后续的差异成分分析。
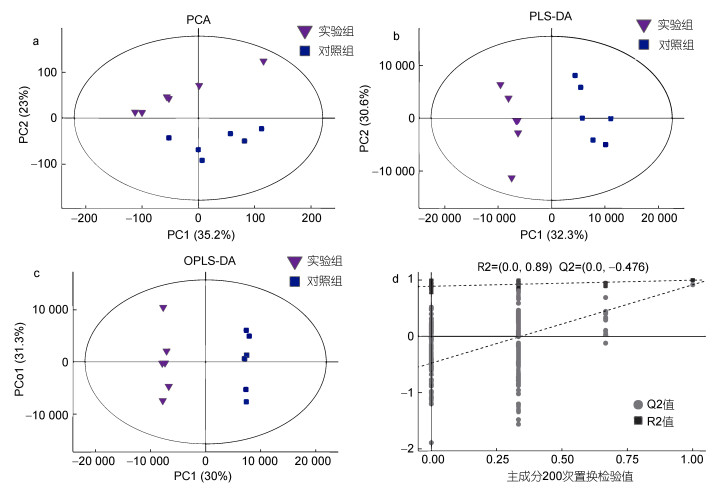 |
| 图 1 经酸化海水处理后, 实验组和对照组厚壳贻贝外套膜组织的代谢物分布PCA、PLS-DA、OPLS-DA得分图和Q2值/R2值分布图 Fig. 1 PCA, PLS-DA, OPLS-DA score plots, and corresponding validation plots (Q2 value and R2 value) of OPLS-DA derived from the control group and the experimental group 注: a. 基于POS和NEG的PCA图; b. 基于POS和NEG的PLS-DA图; c. 基于POS和NEG的OPLS-DA图; d. 基于PLS-DA和OPLS-DA的Q2值和R2值分布图 |
采用单变量分析和T检验手段进行两组间代谢物的统计。进一步采用多维分析和单维分析相结合手段来筛选组间差异代谢物(significant different metabolites, SDM)。首先基于OPLS-DA模型得到变量权重值(variable importance of projection, VIP), 进一步以VIP > 1以及T检验P < 0.05作为SDM筛选阈值。经筛选, 对照组和酸化处理组贻贝外套膜组织中, 共筛选到497种差异代谢物, 其中, 酸化处理组贻贝相比对照组, 其外套膜中有320种SDM上调, 177种下调。在此基础上, 对所有SDM的表达量数据进行火山图分析, 进一步对VIP值最大的50种SDM进行层次聚类(hierarchical clustering)分析, 结果分别见图 2和图 3。上述数据表明, 酸化处理组与对照组相比, 其差异代谢物层次聚类关系良好。
 |
| 图 2 经酸海水化处理后, 实验组和对照组厚壳贻贝外套膜代谢物的分布火山图 Fig. 2 Volcano map of the metabolites of control group and experimental group of M. coruscus mantle after treatment with acidified seawater 注: 火山图基于POS和NEG模式描绘, 每一个点代表一种代谢物, 点的大小代表基于OPLS-DA模型计算的代谢物变量权重值(VIP); 红色代表显著上调代谢物(P < 0.05), 蓝色代表显著下调代谢物(P < 0.05), 灰色代表无显著差异的代谢物(P > 0.05) |
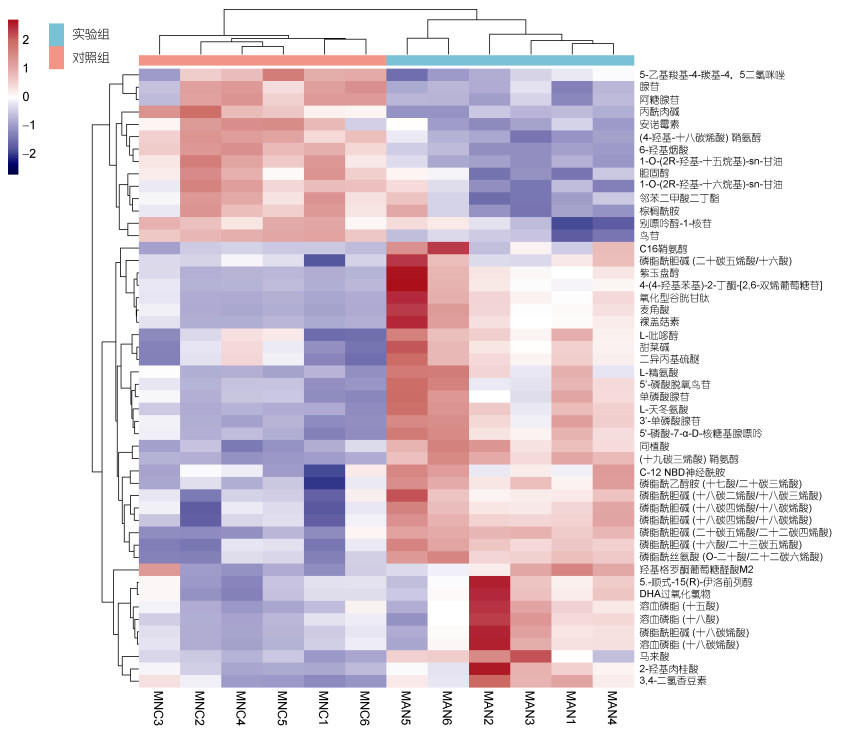 |
| 图 3 厚壳贻贝外套膜经急性酸化处理后, 基于VIP值最大的50种显著差异代谢物的层次聚类分析热图 Fig. 3 Heat-map of hierarchical clustering analysis on the top 50 SDMs of M. coruscus mantel after treatment with acidified seawater 注: MAC代表酸化组, MNC代表对照组; 红色代表上调, 蓝色代表下调, 根据颜色尺度描述代谢物差异水平 |
为进一步了解差异代谢物的生物学功能及其代谢途径变化机制, 利用SDM的KEGG ID进行通路富集分析。应用超几何检验筛选SDM所富集的代谢通路。共有79个SDM被富集到了62条代谢通路, 其中, 显著性富集(P < 0.05)代谢通路为13条, 对显著富集的代谢通路进行气泡图绘制, 结果见图 4。由图 4可见, SDM富集的代谢通路主要包括脂代谢(花生四烯酸代谢、甘油磷脂代谢、鞘脂代谢)、信号转导(mTOR信号通路、FoxO信号通路、神经受体-配体相互作用)、维生素代谢(泛酸和辅酶A生物合成、硫胺素代谢)、核酸代谢(嘌呤代谢、氨酰tRNA的合成)、氨基酸代谢(D-精氨酸和D-鸟氨酸的代谢、精氨酸合成)以及ABC转运体。
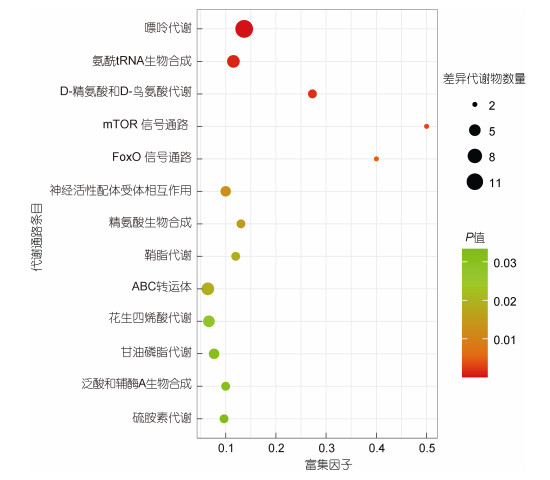 |
| 图 4 厚壳贻贝经酸化海水处理后, 外套膜中差异代谢物的显著通路富集统计气泡图 Fig. 4 The metabolome view map of significant difference metabolic pathways in mantle for M. coruscus after treatment with acidified seawater 注: 基于POS和NEG的显著代谢通路富集气泡图, x轴代表富集因子, y轴代表富集的显著差异代谢通路。富集因子代表差异代谢物和所有注释到此代谢通路的代谢物的比值, 圆点大小代表差异代谢物的数量, 原点越大, 差异代谢物数量越多。圆点的颜色代表着这条代谢通路的P值, 越接近于0, 表示代谢通路越显著 |
利用氨基酸分析仪对实验组和对照组贻贝外套膜组织的游离氨基酸含量进行测定。20种标准氨基酸中, 谷氨酰胺、天冬酰胺、色氨酸和脯氨酸未能检测到。天冬氨酸、甘氨酸和精氨酸是外套膜组织中含量最高的游离氨基酸(表 1)。相比对照组, 酸化组贻贝外套膜中多数游离氨基酸未发生明显变化, 仅谷氨酸、甘氨酸、甲硫氨酸和精氨酸明显上调(P < 0.05) (表 1)。此外, 在其他含氮化合物检测中, 酸化组样品的尿素含量相对于对照组显著升高(P < 0.05) (表 1)。
| 氨基酸名称 | 对照组中浓度/% | 酸化组中浓度/% |
| 牛磺酸 | 1.125±0.763a | 1.224±0.531a |
| 尿素 | 4.76±0.183a | 5.58±0.131b |
| 天冬氨酸 | 1.62±0.112a | 1.85±0.031b |
| 苏氨酸 | 0.13±0.045a | 0.15±0.044a |
| 丝氨酸 | 0.18±0.063a | 0.23±0.047a |
| 谷氨酸 | 0.54±0.013a | 0.69±0.051b |
| 甘氨酸 | 1.04±0.112a | 1.57±0.055b |
| 丙氨酸 | 0.42±0.140a | 0.51±0.065a |
| 缬氨酸 | 0.08±0.032a | 0.1±0.050a |
| 半胱氨酸 | 0.12±0.023a | 0.12±0.022a |
| 甲硫氨酸 | 0.05±0.016a | 0.08±0.049a |
| 异亮氨酸 | 0.05±0.011a | 0.06±0.009a |
| 亮氨酸 | 0.05±0.01a | 0.06±0.015a |
| 酪氨酸 | 0.1±0.022a | 0.1±0.027a |
| 苯丙氨酸 | 0.12±0.003a | 0.11±0.061a |
| 赖氨酸 | 0.12±0.017a | 0.14±0.019a |
| 组氨酸 | 0.11±0.043a | 0.11±0.0005a |
| 精氨酸 | 0.74±0.063a | 0.92±0.037b |
| 注: 不同上标小写字母表示对照组和实验组组间的显著差异(P < 0.05) | ||
进一步对实验组和对照组贻贝外套膜组织开展酶活力测定。结果见表 2。由表 2可见, 与对照组相比, 急性酸化组贻贝外套膜的过氧化氢酶活性及谷胱甘肽的含量明显上调(P < 0.05); 但总抗氧化能力、钠钾泵ATP酶活、总一氧化氮合酶、超氧化物歧化酶活性与对照组相比并无显著差异(P > 0.05)。
| 名称 | 对照组 | 酸化组 |
| 总抗氧化能力 /(U/mg) |
0.121 6±0.009 25a | 0.124 0±0.006 30a |
| 钠钾泵ATP酶活 /(U/mg) |
2.003 6±0.115 02a | 1.965 1±0.375 4a |
| 过氧化氢酶 /(U/mg) |
49.557 9±2.303 61a | 71.894 5±10.052 53b |
| 谷胱甘肽 /(mg/mg) |
0.036 0±0.001 40a | 0.047 7±0.006 21b |
| 总一氧化氮合酶 /(U/mg) |
19.165 2±5.252 35a | 20.323 2±11.662 82a |
| 超氧化物歧化酶 /(U/mg) |
7.677 6±1.057 83a | 8.612 4±2.878 84a |
| 注: 不同上标小写字母表示差异显著(P < 0.05) | ||
目前, 海洋酸化对贝类的影响相关研究已有较多报道, 主要集中在贝类免疫(Wang et al, 2016; Zhao et al, 2018)、生物矿化(Fitzer et al, 2014; Chandra Rajan et al, 2021; Wang et al, 2022)、足丝黏附(Zhao et al, 2017; Zhao et al, 2020)等生理过程。相关研究已揭示海洋酸化对贝类的能量代谢(Jiang et al, 2019; Pousse et al, 2020)、细胞骨架(Wei et al, 2015)、活性氧水平(Cao et al, 2018)等产生不利影响。目前对贝类在海洋酸化条件下的生理和分子响应研究多集中在以牡蛎、蛤类等为代表的贝类物种, 且实验条件设置多以长期酸化为主(Tan et al, 2020b)。但考虑到贝类对海洋酸化具有某种长期适应性(Thomsen et al, 2013, 2017; Fitzer et al, 2019; Tan et al, 2020b), 因此, 对短期急性酸化条件下的贝类开展其生理响应研究将有助于更为直观地展示贝类在应对酸化过程中的分子机制。
通过对厚壳贻贝设置48 h急性酸化, 并对其外套膜组织开展代谢物组学分析, 我们首先注意到, 厚壳贻贝外套膜中的代谢物组成在急性酸化条件下发生明显改变。相比对照组, 外套膜在急性酸化条件下的差异代谢物主要集中在嘌呤及嘌呤衍生物、脂类及其衍生物、氨基酸及其衍生物、维生素和甜菜碱等化合物。上述差异代谢物主要富集于嘌呤代谢、能量代谢、精氨酸合成代谢、脂代谢等通路。其中, 嘌呤代谢通路中, ATP含量在酸化组贻贝外套膜中相比对照组呈现明显下调, 而ADP和AMP则呈现明显上调(P < 0.05) (表 3); 此外, 我们也注意到NAD (nicotinamide adenine dinucleotide, 氧化型烟酰胺腺嘌呤二核苷酸)的含量在酸化组贻贝外套膜中相比对照组也呈现明显上调(P < 0.05), 推测与NADH (nicotinamide adenine dinucleotide reduced, 还原型烟酰胺腺嘌呤二核苷酸)的快速消耗有关。上述结果均意味着急性酸化导致了贻贝外套膜组织的能量消耗速率的增加。这一结果与此前的研究结果类似, 即海洋酸化会增加贻贝的能量消耗, 也因此导致贻贝在海洋酸化条件下表现出摄食速率的增加(Thomsen et al, 2013; Ventura et al, 2016)。游离氨基酸分析结果表明, 酸化组贻贝外套膜中谷氨酸含量明显上调(P < 0.05)。谷氨酸是三羧酸循环反应的关键氨基酸之一, 通常在细胞内被谷氨酸脱氢酶催化生成α-酮戊二酸, 并伴随着NAD转化为NADH (Boland et al, 1977)。结合酸化组贻贝外套膜中NAD含量的上升, 我们推测谷氨酸脱氢的步骤可能受酸化的影响而收到抑制, 因此导致了谷氨酸和NAD含量的上升。此前研究中人们已注意到, 海洋酸化会通常伴随着海水中溶解氧浓度的下降(Gobler et al, 2016); 因此, 部分海洋生物在酸化海水中, 其耗氧量也会下降(Carey et al, 2016)。结合以上文献报道和本研究结果, 我们推测, 急性酸化对贻贝的三羧酸循环产生了某种抑制作用。这对贝类而言是一种极为不利的影响, 一方面贝类在酸化条件下需要消耗更多的能量来应对酸化胁迫; 另一方面, 三酸酸循环的部分抑制导致了能量供应不足。
| 代谢通路名称 | 通路编号 | (差异倍数)差异代谢物 | P值 | 变量权重值 |
| 神经活性配体受体相互作用 | myi04080 | (1.978)L-天冬氨酸 | 0.000 3 | 6.75 |
| (0.360)棕榈酰乙醇酰胺 | < 0.000 1 | 1.25 | ||
| (3.181)白三烯C4 | 0.016 7 | 2.79 | ||
| 嘌呤代谢 | myi00230 | (0.625)腺嘌呤 | 0.015 7 | 1.85 |
| (1.627)3’-单磷酸腺苷 | 0.000 3 | 15.60 | ||
| (0.150)腺嘌呤脱氧核苷酸 | 0.000 8 | 1.07 | ||
| (0.338)脱氧鸟苷 | < 0.000 1 | 1.65 | ||
| (1.460)脱氧鸟苷二磷酸 | 0.001 3 | 1.43 | ||
| (0.464)鸟嘌呤 | < 0.000 1 | 1.63 | ||
| (0.451)鸟苷 | < 0.000 1 | 4.84 | ||
| (1.290)磷酸鸟苷 | 0.000 4 | 2.80 | ||
| (0.519)次黄苷 | 0.032 0 | 3.33 | ||
| (1.598)腺苷二磷酸 | 0.000 8 | 2.32 | ||
| (1.651)单磷酸腺苷 | 0.004 0 | 7.82 | ||
| (0.445)腺苷 | 0.001 0 | 13.31 | ||
| (2.068)腺苷3, 5-二磷酸 | 0.002 1 | 1.07 | ||
| 氨酰tRNA生物合成 | myi00970 | (1.404)L-赖氨酸 | 0.008 6 | 2.17 |
| (1.423) L-脯氨酸 | 0.007 8 | 1.26 | ||
| (1.599)L精氨酸 | 0.003 7 | 7.82 | ||
| (1.978) L-天冬氨酸 | 0.000 3 | 6.75 | ||
| (1.570) L-酪氨酸 | 0.028 4 | 1.55 | ||
| (1.339) L-异亮氨酸 | 0.002 2 | 4.59 | ||
| D-精氨酸和D-鸟氨酸代谢 | myi00472 | (1.562) D-精氨酸 | 0.008 5 | 1.44 |
| (1.507) D-鸟氨酸 | 0.007 3 | 1.62 | ||
| 花生四烯酸代谢 | myi00590 | (0.697)异前列腺素 | 0.001 4 | 1.73 |
| (1.367) 8-异丙前列腺素F2a | 0.034 2 | 1.41 | ||
| (4.156)20-羟基白三烯E4 | 0.001 6 | 1.79 | ||
| 花生四烯酸代谢 | myi00590 | (4.578)白三烯E4 | 0.002 1 | 1.08 |
| (10.762)磷脂酰胆碱(二十碳三烯酸/二十二碳四烯酸) | < 0.000 1 | 1.65 | ||
| (2.980磷脂酰胆碱(十四酸/二十碳四烯酸) | 0.000 3 | 2.17 | ||
| (6.786)磷脂酰胆碱(二十碳五烯酸/二十二碳四烯酸) | < 0.000 1 | 7.87 | ||
| (6.850)二十二碳五烯酸/十四碳烯酸) | < 0.000 1 | 2.99 | ||
| (0.610)磷脂酰胆碱(十八碳烯酸/十八碳烯酸) | 0.000 8 | 1.52 | ||
| 硫胺素代谢 | myi00730 | (1.570) L-酪氨酸 | 0.028 3 | 1.55 |
| (4.908烟酰胺腺嘌呤二核苷酸 | 0.006 5 | 1.52 | ||
| (0.310) 5′-磷酸吡哆醛 | 0.003 5 | 1.01 | ||
| 泛酸和辅酶A生物合成 | myi00770 | (1.978)L-天冬氨酸 | 0.000 3 | 6.75 |
| (2.068)腺苷3, 5-二磷酸 | 0.002 1 | 1.07 | ||
| (1.621)精胺 | 0.020 6 | 1.81 | ||
| 精氨酸生物合成 | myi00220 | (1.599) L-精氨酸 | 0.003 7 | 7.82 |
| (1.978) L-天冬氨酸 | 0.000 3 | 6.75 | ||
| (1.432) N-α-乙酰-L-瓜氨酸 | 0.031 1 | 1.26 | ||
| 鞘脂代谢 | myi00600 | (1.595)神经酰胺 | 0.007 7 | 1.34 |
| (2.381)二氢鞘氨醇 | 0.020 1 | 3.48 | ||
| (3.565)鞘氨醇 | < 0.000 1 | 1.37 | ||
| 甘油磷脂代谢 | myi00564 | (10.762)磷脂酰胆碱(二十碳三烯酸/二十二碳四烯酸) | < 0.000 1 | 1.65 |
| (2.980)磷脂酰胆碱(十四碳酸/二十碳四烯酸) | 0.000 3 | 2.17 | ||
| (6.786)磷脂酰胆碱(二十碳五烯酸/二十二碳四烯酸) | < 0.000 1 | 7.87 | ||
| (6.850)磷脂酰胆碱(二十二碳五烯酸/十四碳烯酸) | < 0.000 1 | 2.99 | ||
| (0.610)磷脂酰胆碱(十八碳烯酸/十八碳烯酸) | 0.000 8 | 1.52 | ||
| (2.242)溶血磷脂(十五酸) | 0.041 9 | 6.48 | ||
| (1.940)溶血磷脂(十八碳烯酸) | 0.044 3 | 5.04 | ||
| (1.576)溶血磷脂(二十碳四烯酸) | 0.008 2 | 1.03 | ||
| (1.764)溶血磷脂(二十碳二烯酸) | 0.027 6 | 2.62 | ||
| (1.998)溶血磷脂(二十二碳六烯酸) | 0.036 3 | 4.33 | ||
| (2.194)磷脂酰胆碱(二十碳四烯酸) | 0.049 7 | 2.25 | ||
| (1.702)溶血磷脂(二十碳四烯酸) | 0.010 0 | 1.38 | ||
| (2.938)磷脂酰乙醇胺(二十二碳二烯酸/二十碳烯酸) | 0.005 4 | 1.46 | ||
| (0.498)磷脂酸(十六酸/十八碳烯酸) | 0.019 2 | 1.11 | ||
| 注: 差异倍数为Exp组/Ctrl组比值, P值由T检验方法获得, 变量权重值基于OPLS-DA模型计算获得 | ||||
海洋酸化对贻贝外套膜的氨基酸代谢也产生明显影响, 包括天冬氨酸、精氨酸、脯氨酸、酪氨酸等含量的显著上调(P < 0.05) (表 3)。氨基酸分析仪的分析结果也证实了精氨酸和天冬氨酸含量的上升(表 1), 该结果也与此前对紫贻贝(Mytilus edulis)在酸化条件下的代谢组学研究结果一致(Ellis et al, 2014)。从SDMs富集的通路来看, 厚壳贻贝外套膜中的精氨酸合成代谢通路在酸化条件下被激活。精氨酸合成通路在贝类中已被证实与一氧化氮的合成有关, 并在贝类免疫中发挥重要作用(王晓安等, 2003); 但本研究中, 一氧化氮合酶的酶活在酸化条件下并未发生明显变化(P > 0.05) (表 2), 这表明精氨酸合成途径的激活可能与其他功能有关。此前研究中已发现, 精氨酸可作为尿素的合成前体, 而尿素可进一步分解为碳酸根和氨离子并参与了部分物种, 如微生物的生物矿化过程(de Muynck et al, 2010; Krajewska, 2018)。氨基酸分析仪的结果也表明, 酸化条件下, 厚壳贻贝外套膜组织中尿素含量有明显上调(P < 0.05) (表 1)。尿素循环在厚壳贻贝中的存在及其代谢过程已在前期研究中被证实(王莹等, 2022), 而精氨酸和天冬氨酸均为尿素循环的关键中间代谢物。其中, 天冬氨酸是合成精氨琥珀酸的前体分子, 而精氨琥珀酸可进一步转化为精氨酸, 并通过精氨酸酶的裂解产生尿素(王莹等, 2022)。上述结果表明, 酸化条件下, 贻贝外套膜的精氨酸合成途径的激活可能有助于产生更多的精氨酸, 并作为前体经裂解后产生尿素, 以帮助贝壳在酸化条件下提供足够的碳酸根离子参与生物矿化。
急性酸化对厚壳贻贝外套膜的脂类代谢也产生明显影响, 表现为花生四烯酸, 鞘脂和甘油磷脂代谢途径的激活, 富集于上述脂类代谢途径的代谢物多数呈上调趋势, 且多为不饱和脂肪酸(表 3)。脂肪酸首先是一种重要的能量分子, 因此, 推测厚壳贻贝外套膜组织在酸化条件下, 脂肪酸类代谢物的含量上升可能有助于满足贻贝在酸化条件下对于能量的需求。此外, 已有研究发现, 海洋酸化会导致虾卤虾(Artemia sinica)体内多不饱和脂肪酸含量明显上升, 特别是二十二碳五烯酸(eicosapentaenoic acid)和二十二碳六烯酸(Docosahexaenoic acid) (Gao et al, 2018); 本研究中也得到类似结果, 两种脂肪酸在酸化胁迫组贻贝外套膜组织中, 相比对照组分别上调6.85倍和1.98倍。多不饱和脂肪酸被认为参与了免疫防御(Calder, 2017); 因此, 酸化条件下, 厚壳贻贝外套膜组织中多不饱和脂肪酸含量的上升可能与其免疫调节有关。但具体机制尚需进一步研究。此外, 磷脂酰胆碱(phosphatidylcholine, PC)含量在酸化组贻贝外套膜组织中显著上调, 且多为含不饱和脂肪酸链PC (表 3)。PC与维持细胞膜的流动性有密切联系(Zheng et al, 2017), 其脂肪酸链不饱和程度的增加意味着在酸化刺激下外套膜组织细胞膜流动性的提高, 此外, 在本研究中, 酸化条件下贻贝外套膜组织中的胆固醇含量出现显著下调。作为细胞膜的重要成分, 胆固醇在相变温度以上, 其含量和细胞流动性成反比(Zhang et al, 2020), 因此, 降低也意味着细胞膜的流动性增强。也意味着细胞膜流动性的加强。已知贻贝在低温条件下, 可通过增加膜脂不饱和度来保持膜的流动性以对抗低温对膜的不利影响(Camus et al, 2000); 而在针对海绵的研究中, 也发现温度和酸化胁迫会导致多不饱和脂肪酸含量上升(Bennett et al, 2018)。上述研究表明, 细胞膜中多不饱和脂肪酸的增加有助于维持生物膜在不利条件下的流动性和稳定性。该结论在针对地中海贻贝的研究中已获得初步证实, 表现为贻贝在酸化条件下, 其溶酶体膜稳定性并未受到影响(Sezer et al, 2020), 其背后的机制可能就在于多不饱和脂肪酸含量的上升和胆固醇的下降。
除上述代谢物之外, 我们还观察到如甜菜碱、NMDA (N-methyl-d-aspartic acid, N-甲基-D-天冬氨酸)等代谢物含量显著上调, 以及阿糖腺苷、6-氢鞘氨醇等代谢物的显著下调。甜菜碱在贻贝中的存在很早已被证实(Wright et al, 1992), 且被认为是一种重要的细胞渗透压保护剂和甲基供体(Zhao et al, 2018)。甜菜碱的合成前体为甘氨酸, 在此前研究中, 已发现甘氨酸-甜菜碱合成途径在贻贝面临环境胁迫时被激活, 继而对贻贝组织的渗透压调节产生重要影响(De Vooys et al, 2002; Liang et al, 2020)。在本研究中, 我们注意到甜菜碱及甘氨酸含量在酸化组贻贝外套膜中均明显上调(P < 0.05), 推测急性酸化导致海水中质子浓度的上升, 从而对贻贝组织细胞的渗透压产生影响。因此, 甜菜碱和甘氨酸含量的上升可能有助于贻贝外套膜组织通过渗透压调节对抗酸化影响, 使贻贝在酸化海水中减少质子渗透, 从而保持细胞内环境pH的相对稳定。NMDA是一种由D-天冬氨酸经甲基化后的产物, 在生物体内可作为NMDA信号通路的激活剂, 通过激活NMDA信号通路而加强细胞外钙离子向细胞内的转运, 以此介导多种生理过程(Korde et al, 2016)。酸化组贻贝外套膜中NMDA含量的上升意味着该信号通路及其相关联的钙离子转运的激活。此前的研究已表明, NMDA信号通路在牡蛎(Crassostrea gigas)幼虫中起到调控变态发育, 通过增加细胞内钙离子浓度而有助于幼虫在变态发育(Vogeler et al, 2021)。考虑到甜菜碱是一种甲基供体(Zhao et al, 2018), 而NMDA的合成需要对D-天冬氨酸进行甲基化, 我们推测NMDA和甜菜碱含量在酸化组贻贝外套膜中的含量上升, 可能有助于激活NMDA信号通路, 从而加强钙离子的转运, 并以此参与贝壳在酸化条件下的生物矿化过程。
下调代谢物中, 阿糖腺苷被认为具有抗病毒活性, 可以通过抑制病毒DNA的合成来达成抗病毒的效果(Seley-Radtke et al, 2018)。此外, 6-氢鞘氨醇是一种鞘脂衍生物, 具有广谱的抗菌活性, 被认为是人类皮肤表面抗菌屏障的重要组成部分(Wertz et al, 1990)。上述两种代谢物在贻贝急性酸化条件下出现下调, 表明贻贝的免疫功能可能受酸化影响而有所减弱。此前已有研究报道在海洋酸化会条件下, 贝类对致病菌的易感性增加(Asplund et al, 2014; Hernroth et al, 2016); 而地中海贻贝(M. galloprovincalis)在酸化条件下, 其鳃组织的抑菌活性出现明显下降(Hernroth et al, 2016)。由此可见, 海水酸化对贻贝可能具有免疫抑制作用, 其背后的机制可能在于部分免疫效应分子的含量下调。
此外, 酸化组贻贝外套膜中, 还原型谷胱甘肽含量呈现明显上调(P < 0.05)并伴随着CAT活性的上升(P < 0.05) (表 2)。表明厚壳贻贝外套膜组织在急性酸化条件下, 其抗氧化能力有所增强。考虑到此前已有研究报道贝类在海洋酸化条件下, 其活性氧水平呈上升趋势(Wang et al, 2016; Sun et al, 2017), 因此, 贻贝外套膜组织在急性酸化条件下, 其抗氧化能力的增强将有助于应对升高的活性氧水平。但值得注意的是, 在海水酸化对贝类的影响研究中, 不同贝类物种以及不同组织可能导致研究结果存在差异。例如, 在海水酸化条件下, 贻贝性腺组织的CAT和SOD酶活力被抑制, 但GSH含量上升(Xu et al, 2021); 在太平洋牡蛎消化腺中, 海水酸化导致GSH含量, 以及CAT和SOD酶活均下调(Wang et al, 2016); 而在贻贝鳃组织中, 多重胁迫(包括海水酸化、低氧和高温)导致其组织内GSH含量先上调后下调, SOD和CAT酶活则受到抑制(Khan et al, 2021)。上述文献报道结果与本研究结果之间的差异, 推测由于实验条件设置, 以及物种或组织差异所导致。
4 结论综上所述, 急性海洋酸化在短期内会对贻贝外套膜产生明显的代谢物组成和含量变化, 差异代谢物富集的代谢通路主要包括脂代谢、信号转导、维生素代谢、核酸代谢、氨基酸代谢以及ABC转运体。结合外套膜游离氨基酸组成分析以及抗氧化活性分析, 推测厚壳贻贝通过激活尿素合成、增强细胞膜流动性以及加强渗透压调节和钙离子转运等方式来应对海洋酸化的威胁。
王莹, 范孝俊, 刘菲, 等, 2022. 厚壳贻贝鸟氨酸-尿素循环中的关键代谢物及基因分析. 中国生物化学与分子生物学报, 38(5): 638-647 |
王晓安, 蒋小满, 郑哲民, 2003. 软体动物的一氧化氮及其合酶的研究进展. 动物学杂志, 38(6): 97-103 |
刘晓辉, 孙丹青, 黄备, 等, 2017. 东海沿岸海域表层海水酸化趋势及影响因素研究. 海洋与湖沼, 48(2): 398-405 |
ASHUR M M, JOHNSTON N K, DIXSON D L, 2017. Impacts of ocean acidification on sensory function in marine organisms. Integrative and Comparative Biology, 57(1): 63-80 DOI:10.1093/icb/icx010 |
ASPLUND M E, BADEN S P, RUSS S, et al, 2014. Ocean acidification and host-pathogen interactions: blue mussels, Mytilus edulis, encountering Vibrio tubiashii. Environmental Microbiology, 16(4): 1029-1039 DOI:10.1111/1462-2920.12307 |
BENNETT H, BELL J J, DAVY S K, et al, 2018. Elucidating the sponge stress response; Lipids and fatty acids can facilitate survival under future climate scenarios. Global Change Biology, 24(7): 3130-3144 DOI:10.1111/gcb.14116 |
BOLAND M J, BENNY A G, 1977. Enzymes of nitrogen metabolism in legume nodules. Purification and properties of NADH-dependent glutamate synthase from Lupin nodules. European Journal of Biochemistry, 79(2): 355-362 DOI:10.1111/j.1432-1033.1977.tb11816.x |
CALDER P C, 2017. Omega-3 fatty acids and inflammatory processes: from molecules to man. Biochemical Society Transactions, 45(5): 1105-1115 DOI:10.1042/BST20160474 |
CAMUS L, GRØSVIK B E, BØRSETH J F, et al, 2000. Stability of lysosomal and cell membranes in haemocytes of the common mussel (Mytilus edulis): effect of low temperatures. Marine Environmental Research, 50(1/2/3/4/5): 325-329 |
CAO R W, WANG Q, YANG D L, et al, 2018. CO2-induced ocean acidification impairs the immune function of the Pacific oyster against Vibrio splendidus challenge: an integrated study from a cellular and proteomic perspective. Science of the Total Environment, 625: 1574-1583 DOI:10.1016/j.scitotenv.2018.01.056 |
CAREY N, DUPONT S, SIGWART J D, 2016. Sea hare Aplysia punctata (Mollusca: Gastropoda) can maintain shell calcification under extreme ocean acidification. The Biological Bulletin, 231(2): 142-151 DOI:10.1086/690094 |
CARROLL S L, COYNE V E, 2021. A proteomic analysis of the effect of ocean acidification on the haemocyte proteome of the South African abalone Haliotis midae. Fish & Shellfish Immunology, 117: 274-290 |
CHANDRA RAJAN K, MENG Y, YU Z N, et al, 2021. Oyster biomineralization under ocean acidification: from genes to shell. Global Change Biology, 27(16): 3779-3797 DOI:10.1111/gcb.15675 |
DE MUYNCK W, VERBEKEN K, DE BELIE N, et al, 2010. Influence of urea and calcium dosage on the effectiveness of bacterially induced carbonate precipitation on limestone. Ecological Engineering, 36(2): 99-111 DOI:10.1016/j.ecoleng.2009.03.025 |
DE VOOYS C G N, GEENEVASEN J A J, 2002. Biosynthesis and role in osmoregulation of glycine-betaine in the Mediterranean mussel Mytilus galloprovincialis LMK.. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 132(2): 409-414 DOI:10.1016/S1096-4959(02)00052-0 |
DONEY S C, FABRY V J, FEELY R A, et al, 2009. Ocean acidification: the other CO2 problem. Annual Review of Marine Science, 1: 169-192 DOI:10.1146/annurev.marine.010908.163834 |
DOO S S, EDMUNDS P J, CARPENTER R C, 2019. Ocean acidification effects on in situ coral reef metabolism. Scientific Reports, 9(1): 12067 DOI:10.1038/s41598-019-48407-7 |
ELLIS R P, SPICER J I, BYRNE J J, et al, 2014. 1H NMR metabolomics reveals contrasting response by male and female mussels exposed to reduced seawater pH, increased temperature, and a pathogen. Environmental Science & Technology, 48(12): 7044-7052 |
EYRE B D, CYRONAK T, DRUPP P, et al, 2018. Coral reefs will transition to net dissolving before end of century. Science, 359(6378): 908-911 DOI:10.1126/science.aao1118 |
FITZER S C, MCGILL R A R, TORRES GABARDA S, et al, 2019. Selectively bred oysters can alter their biomineralization pathways, promoting resilience to environmental acidification. Global Change Biology, 25(12): 4105-4115 DOI:10.1111/gcb.14818 |
FITZER S C, PHOENIX V R, CUSACK M, et al, 2014. Ocean acidification impacts mussel control on biomineralisation. Scientific Reports, 4: 6218 DOI:10.1038/srep06218 |
GAO Y, ZHENG S C, ZHENG C Q, et al, 2018. The immunerelated fatty acids are responsive to CO2 driven seawater acidification in a crustacean brine shrimp Artemia sinica. Developmental & Comparative Immunology, 81: 342-347 |
GOBLER C J, BAUMANN H, 2016. Hypoxia and acidification in ocean ecosystems: coupled dynamics and effects on marine life. Biology Letters, 12(5): 20150976 DOI:10.1098/rsbl.2015.0976 |
GONCALVES P, THOMPSON E L, RAFTOS D A, 2017. Contrasting impacts of ocean acidification and warming on the molecular responses of CO2-resilient oysters. BMC Genomics, 18(1): 431 DOI:10.1186/s12864-017-3818-z |
HERNROTH B, BADEN S, TASSIDIS H, et al, 2016. Impact of ocean acidification on antimicrobial activity in gills of the blue mussel (Mytilus edulis). Fish & Shellfish Immunology, 55: 452-459 |
JIANG Z J, WANG X Q, RASTRICK S P S, et al, 2019. Metabolic responses to elevated pCO2 in the gills of the Pacific oyster (Crassostrea gigas) using a GC-TOF-MS-based metabolomics approach. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 29: 330-338 DOI:10.1016/j.cbd.2019.01.003 |
KHAN F U, CHEN H, GU H X, et al, 2021. Antioxidant responses of the mussel Mytilus coruscus co-exposed to ocean acidification, hypoxia and warming. Marine Pollution Bulletin, 162: 111869 DOI:10.1016/j.marpolbul.2020.111869 |
KORDE A S, MARAGOS W F, 2016. Direct exposure to N-methyl-D-aspartate alters mitochondrial function. Neuroscience Letters, 623: 47-51 DOI:10.1016/j.neulet.2016.04.054 |
KRAJEWSKA B, 2018. Urease-aided calcium carbonate mineralization for engineering applications: a review. Journal of Advanced Research, 13: 59-67 DOI:10.1016/j.jare.2017.10.009 |
LIANG R Y, SHAO X Q, SHI Y J, et al, 2020. Antioxidant defenses and metabolic responses of blue mussels (Mytilus edulis) exposed to various concentrations of erythromycin. Science of the Total Environment, 698: 134221 DOI:10.1016/j.scitotenv.2019.134221 |
LIU Z Q, ZHANG Y K, ZHOU Z, et al, 2020. Metabolomic and transcriptomic profiling reveals the alteration of energy metabolism in oyster larvae during initial shell formation and under experimental ocean acidification. Scientific Reports, 10(1): 6111 DOI:10.1038/s41598-020-62963-3 |
MELZNER F, GUTOWSKA M A, LANGENBUCH M, et al, 2009. Physiological basis for high CO2 tolerance in marine ectothermic animals: pre-adaptation through lifestyle and ontogeny?. Biogeosciences, 6(10): 2313-2331 DOI:10.5194/bg-6-2313-2009 |
POUSSE E, POACH M E, REDMAN D H, et al, 2020. Energetic response of Atlantic surfclam Spisula solidissima to ocean acidification. Marine Pollution Bulletin, 161: 111740 DOI:10.1016/j.marpolbul.2020.111740 |
QUEIRÓS A M, FERNANDES J A, FAULWETTER S, et al, 2015. Scaling up experimental ocean acidification and warming research: from individuals to the ecosystem. Global Change Biology, 21(1): 130-143 DOI:10.1111/gcb.12675 |
SELEY-RADTKE K L, YATES M K, 2018. The evolution of nucleoside analogue antivirals: a review for chemists and non-chemists. Part 1: early structural modifications to the nucleoside scaffold. Antiviral Research, 154: 66-86 DOI:10.1016/j.antiviral.2018.04.004 |
SEZER N, KILIÇ Ö, SIKDOKUR E, et al, 2020. Impacts of elevated pCO2 on Mediterranean mussel (Mytilus galloprovincialis): metal bioaccumulation, physiological and cellular parameters. Marine Environmental Research, 160: 104987 DOI:10.1016/j.marenvres.2020.104987 |
SUI Y, HU M H, SHANG Y Y, et al, 2017. Antioxidant response of the hard shelled mussel Mytilus coruscus exposed to reduced pH and oxygen concentration. Ecotoxicology and Environmental Safety, 137: 94-102 DOI:10.1016/j.ecoenv.2016.11.023 |
SUN T L, TANG X X, JIANG Y S, et al, 2017. Seawater acidification induced immune function changes of haemocytes in Mytilus edulis: a comparative study of CO2 and HCl enrichment. Scientific Reports, 7: 41488 DOI:10.1038/srep41488 |
SUNDAY J M, CRIM R N, HARLEY C D G, et al, 2011. Quantifying rates of evolutionary adaptation in response to ocean acidification. PLoS One, 6(8): e22881 DOI:10.1371/journal.pone.0022881 |
TAN K, LIU H X, YE T, et al, 2020a. Growth, survival and lipid composition of Crassostrea gigas, C. angulata and their reciprocal hybrids cultured in southern China. Aquaculture, 516: 734524 DOI:10.1016/j.aquaculture.2019.734524 |
TAN K, ZHENG H P, 2020b. Ocean acidification and adaptive bivalve farming. Science of the Total Environment, 701: 134794 DOI:10.1016/j.scitotenv.2019.134794 |
THOMSEN J, CASTIES I, PANSCH C, et al, 2013. Food availability outweighs ocean acidification effects in juvenile Mytilus edulis: laboratory and field experiments. Global Change Biology, 19(4): 1017-1027 DOI:10.1111/gcb.12109 |
THOMSEN J, STAPP L S, HAYNERT K, et al, 2017. Naturally acidified habitat selects for ocean acidification-tolerant mussels. Science Advances, 3(4): e1602411 DOI:10.1126/sciadv.1602411 |
TIMMINS-SCHIFFMAN E, COFFEY W D, HUA W, et al, 2014. Shotgun proteomics reveals physiological response to ocean acidification in Crassostrea gigas. BMC Genomics, 15(1): 951 DOI:10.1186/1471-2164-15-951 |
VENTURA A, SCHULZ S, DUPONT S, 2016. Maintained larval growth in mussel larvae exposed to acidified under-saturated seawater. Scientific Reports, 6: 23728 DOI:10.1038/srep23728 |
VOGELER S, CARBONI S, LI X X, et al, 2021. Cloning and characterisation of NMDA receptors in the Pacific oyster, Crassostrea gigas (Thunberg, 1793) in relation to metamorphosis and catecholamine synthesis. Developmental Biology, 469: 144-159 DOI:10.1016/j.ydbio.2020.10.008 |
WANG Q, CAO R W, NING X X, et al, 2016. Effects of ocean acidification on immune responses of the Pacific oyster Crassostrea gigas. Fish & Shellfish Immunology, 49: 24-33 |
WANG M H, JEONG C B, LEE Y H, et al, 2018. Effects of ocean acidification on copepods. Aquatic Toxicology, 196: 17-24 DOI:10.1016/j.aquatox.2018.01.004 |
WANG X, LI P, CAO X Q, et al, 2022. Effects of ocean acidification and tralopyril on bivalve biomineralization and carbon cycling: a study of the Pacific Oyster (Crassostrea gigas). Environmental Pollution, 313: 120161 DOI:10.1016/j.envpol.2022.120161 |
WANG C Y, YAN X J, HE J Y, et al, 2021. Responses to β-alanine and carnosine supplementation of mussel Mytilus coruscus as revealed by UPLC–MS/MS based untargeted metabolomics. Aquaculture Reports, 20: 100730 DOI:10.1016/j.aqrep.2021.100730 |
WEI L, WANG Q, NING X X, et al, 2015. Combined metabolome and proteome analysis of the mantle tissue from Pacific oyster Crassostrea gigas exposed to elevated pCO2. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 13: 16-23 DOI:10.1016/j.cbd.2014.12.001 |
WERTZ P W, DOWNING D T, 1990. Free sphingosine in human epidermis. Journal of Investigative Dermatology, 94(2): 159-161 DOI:10.1111/1523-1747.ep12874122 |
WRIGHT S H, WUNZ T M, SILVA A L, 1992. Betaine transport in the gill of a marine mussel, Mytilus californianus. The American Journal of Physiology, 263(2): R226-R232 |
XU G G, KONG H, CHANG X Q, et al, 2021. Gonadal antioxidant responses to seawater acidification and hypoxia in the marine mussel Mytilus coruscus. Environmental Science and Pollution Research, 28(38): 53847-53856 DOI:10.1007/s11356-021-14584-0 |
YARRA T, RAMESH K, BLAXTER M, et al, 2021. Transcriptomic analysis of shell repair and biomineralization in the blue mussel, Mytilus edulis. BMC Genomics, 22(1): 437 DOI:10.1186/s12864-021-07751-7 |
ZHANG Y, LI Q C, DONG M D, et al, 2020. Effect of cholesterol on the fluidity of supported lipid bilayers. Colloids and Surfaces B: Biointerfaces, 196: 111353 DOI:10.1016/j.colsurfb.2020.111353 |
ZHAO X G, GUO C, HAN Y, et al, 2017. Ocean acidification decreases mussel byssal attachment strength and induces molecular byssal responses. Marine Ecology Progress Series, 565: 67-77 DOI:10.3354/meps11992 |
ZHAO G F, HE F, WU C L, et al, 2018. Betaine in inflammation: mechanistic aspects and applications. Frontiers in Immunology, 9: 1070 DOI:10.3389/fimmu.2018.01070 |
ZHAO L Q, LIANG J, LIANG J P, et al, 2020. Experimental study of transgenerational effects, pH and predation risk on byssus production in a swiftly spreading invasive fouling Asian mussel, Musculista senhousia (Benson). Environmental Pollution, 260: 114111 DOI:10.1016/j.envpol.2020.114111 |
ZHENG L, LIN Y B, LU S, et al, 2017. Biogenesis, transport and remodeling of lysophospholipids in Gram-negative bacteria. Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids, 1862(11): 1404-1413 DOI:10.1016/j.bbalip.2016.11.015 |
ZUO Y Q, ZENG R Y, TIAN C M, et al, 2022. The importance of conditionally rare taxa for the assembly and interaction of fungal communities in mangrove sediments. Applied Microbiology and Biotechnology, 106(9/10): 3787-3798 DOI:10.1007/s00253-022-11949-4?utm_source=xmol&utm_content=meta |
 2023, Vol. 54
2023, Vol. 54


