
中国海洋湖沼学会主办。
文章信息
- 张静静, 王亚冰, 王倩, 韩多彩, 彭士明. 2023.
- ZHANG Jing-Jing, WANG Ya-Bing, WANG Qian, HAN Duo-Cai, PENG Shi-Ming. 2023.
- 不同抗流能力大黄鱼(Larimichthys crocea)肌肉转录组学差异分析
- TRANSCRIPTOME ANALYSIS OF MUSCLE TISSUE OF LARIMICHTHYS CROCEA WITH DIFFERENT FLOW RESISTANCE
- 海洋与湖沼, 54(5): 1507-1516
- Oceanologia et Limnologia Sinica, 54(5): 1507-1516.
- http://dx.doi.org/10.11693/hyhz20230200034
文章历史
-
收稿日期:2023-02-16
收修改稿日期:2023-05-21
2. 中国农业科学院研究生院 北京 100081
2. Graduate School of Chinese Academy of Agricultural Sciences, Beijing 100081, China
大黄鱼是我国养殖规模最大的海水鱼类, 被列为我国八大优势出口养殖水产品之一(农业农村部渔业渔政管理局等, 2021; 韩承义等, 2022)。传统的大黄鱼养殖主要集中在近海海域, 近海网箱养殖过程中, 除陆源输入外, 排泄物及残饵以及养殖区自身污染物的大量排放可引起局部海域氮磷营养盐、有机质含量过高, 造成水质恶化。随着大黄鱼养殖规模的扩大, 病害频发、品质退化、成活率低等问题日益突出, 严重影响了大黄鱼的产业效益(Cao et al, 2015; Ma et al, 2020; 徐鹏等, 2022)。因此, 推动大黄鱼深远海养殖产业发展是实现大黄鱼养殖提质增效、产业转型升级的有效途径, 也是践行大食物观, 向江河湖海要食物, 推进我国深蓝渔业发展的重要战略举措(徐皓等, 2021)。深远海养殖海域营养丰富、水体交换率高、污染物含量少, 配合现代化养殖装备及先进的养殖技术, 可高效获得高品质的养殖大黄鱼(邹国华等, 2021)。然而, 深远海养殖水域浪高流急、海况恶劣, 且易受台风大浪的侵袭, 这对于大黄鱼的养殖是一个巨大的挑战, 要求养殖大黄鱼需要具备较强的抗流能力。目前, 通过国审的4个大黄鱼新品种其选育的性状主要聚焦在生长、条形及耐低温等方面, 这些品种无法应对复杂的海域环境, 特别是较快的水流条件。深远海养殖鱼类新品种现已被列入《2022农业农村产业发展重大技术需求》。因此, 为满足深远海养殖大黄鱼种业发展需求, 需培育抗流能力强适宜深远海养殖的大黄鱼新品种, 以推动我国大黄鱼深远海养殖产业的高质量发展。
鱼类性状的遗传解析可为新品种创制及其育种技术开发提供重要的理论基础。目前已有的针对大黄鱼性状遗传基础解析的研究主要聚焦在疾病(Zhao et al, 2021; Bai et al, 2022; Zhang et al, 2022)、低氧(Mu et al, 2020)、低温(曾霖等, 2022)、生长(Zhou et al, 2019)、性别控制(Luo et al, 2019)等方面, 针对大黄鱼抗流性状的遗传基础解析尚未见有相关报道。本研究基于转录组技术(RNA-seq)对不同抗流能力大黄鱼肌肉组织进行转录组分析, 以期从转录组学的层面解析大黄鱼抗流性状的分子基础, 研究结果可为培育适宜深远海养殖大黄鱼抗流新品种奠定理论基础。
1 材料与方法 1.1 实验鱼抗流分组及样本采集2021年9月从福建省福鼎市沙埕湾主养区收集1 000余尾大黄鱼, 购买于同一家养殖户, 这批鱼来自同一批受精卵(来源于宁德市富发水产有限公司), 养殖区位于沙埕湾后港村的养殖渔排, 体重(274.58± 14.09) g, 体长(23.9±0.96) cm, 为1龄大黄鱼, 其养殖周期、养殖模式及所处水域的水文特征均为一致。大黄鱼暂养于2口室内水泥池(直径6 m, 水深1.5 m, 500尾/池)中, 进行一周的。暂养条件为: 水温(27.0± 1.0) ℃, 盐度25~26, 溶氧量DO > 6 mg/L。每天早晚两次投喂商业饲料至饱食状态, 日换水量1/2。
在进行抗流筛选实验前大黄鱼禁食24 h, 通过抗流实验装置(图 1)进行大黄鱼抗流能力强弱的分筛, 抗流装置内水深0.5 m, 每次取50条鱼放入水槽中(E区), 用充气泵保持足够的溶氧水平, 水流控制采用无极变速, 以实验鱼体长(约20 cm/s)的流速作为初始流速, 使实验鱼适应20~30 min, 随后在1 min内使流速增加到1.0 m/s, 观察并记录大黄鱼的抗流时间, 将抗流时间 > 30 min的大黄鱼归为HM组, 抗流时间 < 5 min且以抵触隔网30 s作为筛选条件确定为SM组。用于进行样品采集及统计48 h累计死亡率的实验鱼其具体操作流程如下: 在分筛实验前, 根据大黄鱼的游动能力, 将所有大黄鱼初步分为了游动能力强组和游动能力弱组。操作的目的除了提高分筛的效率外, 主要是考虑到抗流装置每次分筛只能一次性放置50尾鱼, 实验鱼被筛选出来的先后顺序是不同的。为了准确统计抗流组和非抗流组的48 h累计死亡率, 避免相同实验组内实验鱼在时间上的不同步性, 在初步分组的基础上, 分别针对游动能力强组和游动能力弱组各进行3批次的抗流分筛。针对游动能力强组的3批次抗流分筛, 分别筛选到35、38、39尾HM组实验鱼, 每次分筛结束后将筛选到的HM组实验鱼放置于单独的一个水泥池中(直径4 m, 水深1 m), 然后分别统计3个水泥池中实验鱼的48 h累计死亡率。针对游动能力弱组的3批次抗流分筛, 分别筛选到37、40、36尾SM组实验鱼, 采用上述相同的方法统计48 h累计死亡率。48 h后, 分别从HM组、SM组随机收集18尾大黄鱼, 以20 mg/L丁香酚溶液进行麻醉, 统一收集背鳍中间下方1 cm左右的肌肉组织0.2 g, 每6尾大黄鱼肌肉组织等量混合放置于2 mL的无菌冻存管中, 每个处理组共收集3管, 经液氮速冻后置于–80 ℃冰箱保存。
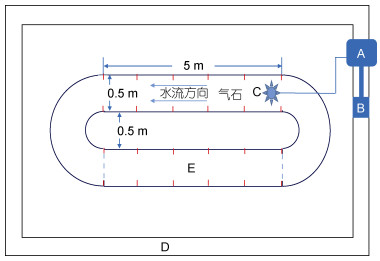 |
| 图 1 大黄鱼抗流实验装置示意图 Fig. 1 The schematic diagram of anti-flow experiment of large yellow croaker 注: A: 电机; B: 变频器; C: 螺旋桨; D: 水泥池; E: 跑道式水槽, 红色短线表示插槽, 用于放置隔离网片, 网片放置位置如蓝色虚线所示 |
根据Trizol试剂盒(Invitrogen, 美国)提取每管肌肉组织的总RNA, 并分别使用Nanodrop®分光光度计、Agilent 2100生物分析仪进行RNA的纯度、浓度及完整性检测。用带有Oligo(dT)的磁珠富集mRNA, 以mRNA为模板, 合成cDNA, 利用AM Pure XP beads纯化cDNA, 纯化的双链cDNA再进行末端修复、加poly(A)尾并连接测序接头, 然后用AMPure XP beads进行片段大小选择; 最后通过PCR富集得到cDNA文库。检测合格的cDNA文库在Illumina HiseqTM 2500平台上测序。
1.2.2 转录组分析利用Fast QC软件对测序得到的原始数据进行质量评估, 去除原始数据中含有带接头和低质量的片段, 保证获得的数据具有品质较高的有效测序数据(clean reads)。利HISAT2软件, 使用BWT算法将clean reads与大黄鱼参考基组(ftp://ftp. ncbi.nlm.nih.gov/genomes/all/GCF/000/972/845/GCF_000972845.2_L_crocea_2.0)进行比对。使用DESeq2软件采用FPKM计算方法将clean data标准化, 以差异倍数|log2 fold change|≥1且P < 0.05为标准筛选差异表达基因(DEGs)。对组装的序列进行遗传相似性比较, 以进行蛋白质功能注释, 将单基因与GO、COG (clusters of orthologous groups)、KOG (eukaryotic orthologous groups)、KEGG、Nr (non-redundant)、Swiss-Pro和Pfam数据库进行比对并注释。根据注释结果, 分别对DEGs的功能和生物学途径进行分类。将DEGs与GO数据库和KEGG数据库进行比对, 从而获得GO功能注释和KEGG通路富集分析。
1.2.3 DEGs RT-qPCR验证随机选取DEGs中的8个基因, 以β-actin为内参基因, 进行RT-qPCR检验, 特异性引物使用软件NCBI在线设计(表 1)。使用Quant Studio 6 Flex进行RT-qPCR, 反应体系按Ultra SYBR Mixture (康为, 中国)试剂盒说明书进行。反应体系为: 2×Ultra SYBR Mixture 12.5 μL; Forward primer (10 μmol/L) 0.5 μL; Reverse primer (10 μmol/L) 0.5 μL; Template cDNA 1 μL; ddH2O 10.5 μL, 总体积25 μL。采用两步法PCR扩增标准程序, 循环参数为反应程序: 95 ℃预变性10 min, 95 ℃变性15 s, 60 ℃退火延伸1 min, 共40个循环。对所得数据进行溶解曲线分析, 用2–∆ ∆ Ct法计算基因的相对表达量。
| 基因名称 | 基因ID | 正向引物(5'~3') | 反向引物(5'~3') | 产物长度/bp |
| β-actin | 104932075 | CTACGAGGGTTATGCCCTGCC | TGAAGGAGTAACCGCGCTCTGT | 107 |
| amp3a | 104930104 | CGGGTTGGACACGGTTACAGAAC | GACGACGGGATGCTTGGTGAAG | 144 |
| jund | 104930914 | TGTCTCCCATCGACATTGACTC | ATGGGTGAGGACTTTCTGCTTC | 221 |
| myo7a | 113746571 | TAATGCTGCTGCTGCTGCTCTG | CTCCAACTCACACTGACACTCCTC | 101 |
| igf-1 | 104926290 | GACGAGTGCTGCTTCCAAAG | TGCGGTACTAACCTTGGGTG | 138 |
| mmp2 | 104926945 | AGGAAACCCATGTGGCAGAAGAAAG | GGCGTGTGAATGAGAGTGGAGTAAC | 142 |
| hid1b | 104928156 | GCTCATCGTGATCTTCCACAAAG | GTACACGAGGTTGGAGTTACCAT | 274 |
| myo1eb | 104937078 | ACAGCATCAACAGGAACTTTGTG | TTTCTCGACCAATCAGGTACAGG | 189 |
| ubqln1 | 104933122 | AACATCCACAACAACTTTCTGCC | GATCCTTTCACACCTCTGACGAT | 178 |
累计死亡率数据(
利用RNA-seq对HM组、SM组进行测序分析, 对测得的原始数据进一步质控后得到21 522 509~ 24 869 415条有效测序量, 与大黄鱼参考基因组比对后得到41 412 309~47 651 205条匹配的序列, 比对率为95.27%~96.37%, 拼接共获得33 191个单基因(unigene)。本试验中GC含量区间为51.51%~52.15%, Q30 > 92.9% (表 2), 表明测序数据质量可用于对后续数据分析。
| 注释数据库 | 注释 数目 |
注释率/% | 300 bp≤长度 < 1 000 bp | 长度 ≥1 000 bp |
| COG Annotation | 7 818 | 32.24 | 2 057 | 5 707 |
| GO Annotation | 18 494 | 76.26 | 5 373 | 12 895 |
| KEGG Annotation | 24 177 | 99.69 | 7 860 | 15 871 |
| KOG Annotation | 16 663 | 68.71 | 4 593 | 11 861 |
| Pfam Annotation | 21 967 | 90.58 | 6 818 | 14 831 |
| Swissprot Annotation | 19 315 | 79.64 | 5 618 | 13 454 |
| Nr Annotation | 24 244 | 99.97 | 7 883 | 15 911 |
| All Annotation | 24 252 | — | 7 885 | 15 916 |
将unigene与常用数据库进行比对并注释, 发现共有24 252条unigene被注释, 其中有18 494个unigene在GO中注释(76.26%), 长度大于1 000 bp有12 895个unigene; 在KEGG注释到24 177个unigene (99.69%), 长度大于1 000 bp有15 871个单基因(表 3)。
| 样本 | 读数 | 总碱基数 | 有效测序量 | 比对数目 | 比对率/% | GC含量/% | Q30/% |
| HM-1 | 24 869 415 | 7 460 824 500 | 24 869 415 | 47 651 205 | 95.80 | 51.63 | 93.21 |
| HM-2 | 21 522 509 | 6 456 752 700 | 21 522 509 | 41 412 309 | 96.21 | 51.71 | 93.68 |
| HM-3 | 22 598 779 | 6 779 633 700 | 22 598 779 | 43 558 252 | 96.37 | 51.61 | 93.70 |
| SM-1 | 22 322 361 | 6 696 708 300 | 22 322 361 | 42 868 913 | 96.02 | 52.15 | 93.85 |
| SM-2 | 23 618 051 | 7 085 415 300 | 23 618 051 | 45 001 087 | 95.27 | 51.03 | 92.90 |
| SM-3 | 23 915 186 | 7 174 555 800 | 23 915 186 | 45 611 243 | 95.36 | 51.51 | 93.11 |
将HM组与SM组肌肉组织DEGs进行对比分析。将基因表达量倍数设置为二倍以上, P < 0.05的基因作为DEGs。由HM组与SM大黄鱼肌肉组织的RNA测序和基因表达谱分析可知: DEGs的总数一共有1 806, 其中有1 090个基因显著上调(P < 0.05; 图 2, 红色), 716个基因显著下调(P < 0.05; 图 2, 绿色)。聚类分析热图显示, HM与SM组间差异基因表达模式相差较大, HM组个体间表达模式相似, 但是SM组个体间基因表达模式存在明显差异(图 3)。
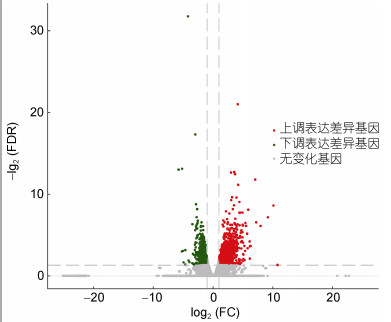 |
| 图 2 差异表达基因火山图 Fig. 2 Volcano diagram of differentially expressed genes |
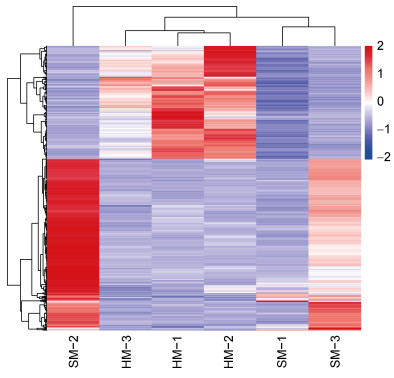 |
| 图 3 组间差异表达基因分层聚类热图 Fig. 3 Heatmap of differentially expressed genes between groups 注: 图中红色代表表达量上调基因, 蓝色代表表达量下调基因 |
为进一步探究HM组与SM组DEGs的功能, 对DEGs进行GO功能注释。DEGs在细胞组分(cellular component, CC)、分子功能(molecular function, MF)和生物学过程(biological process, BP)中均有富集。在CC中显著富集在肌钙蛋白复合物、蛋白酶体辅助复合物、肌球蛋白、线粒体内膜等条目; 在MF中显著富集在MAP激酶酪氨酸/丝氨酸/苏氨酸磷酸酶活性条目; 在BP中显著富集在Rho蛋白信号转导的调节条目(图 4)。
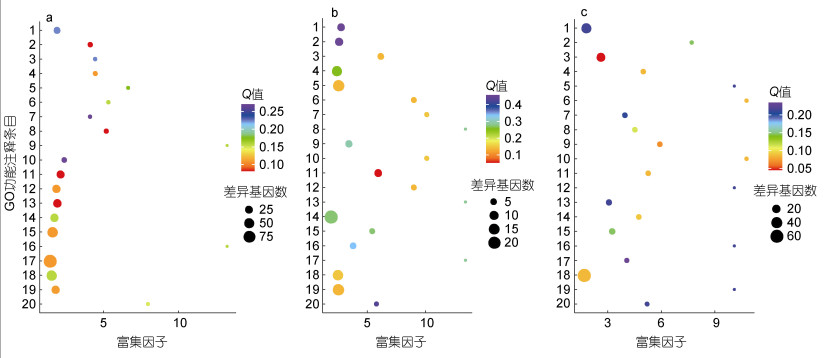 |
| 图 4 转录组差异表达基因的GO分析 Fig. 4 Gene ontology (GO) item analyses of differentially expressed genes in transcriptome 注: a. 细胞组分: 1. Z盘; 2. 肌钙蛋白复合物; 3. 肌浆网; 4. 肌节; 5. 蛋白酶体调节颗粒, 盖子亚复合物; 6. 蛋白酶体核心复合物, α-亚基复合物; 7. 蛋白酶体复合物; 8. 蛋白酶体辅助复合物; 9. 钾离子通道复合物; 10. 核染色质; 11. 肌球蛋白; 12. 肌原纤维; 13. 线粒体内膜; 14. 黏着斑; 15. 细胞外基质; 16. 以DNA为模板的RNA聚合酶Ⅰ复合物; 17. 细胞质; 18. 细胞骨架; 19. 三聚体胶原; 20. 乳糜微粒。b. 分子功能: 1. 泛素连接酶结合; 2. 泛素结合; 3. 鞘氨醇-1-磷酸受体活性; 4. RNA聚合酶Ⅱ近端启动子序列特异性DNA; 5. 鸟苷酸交换因子活性; 6. 蛋白酪氨酸/苏氨酸磷酸酶活性; 7. 孕酮受体结合; 8. NADPH结合; 9. 肌动蛋白结合; 10. 微管正端结合; 11. MAP激酶酪氨酸/丝氨酸/苏氨酸磷酸酶活性; 12. MAP激酶丝氨酸/苏氨酸磷酸酶活性; 13. 细胞内ATP门控氯离子通道活性; 14. 细胞外基质结构成分; 15. DNA结合5' - 3' RNA聚合酶活性; 16. 细胞色素C氧化酶活性; 17. 氯离子跨膜转运体活性; 18. ATPase活性; 19. 肌动蛋白结合; 20. 6-磷酸果糖-2-激酶的活性。c. 生物过程: 1. RNA聚合酶Ⅱ对转录的调节; 2. 横纹肌收缩的调节; 3. Rho蛋白信号转导的调节; 4. 活性氧代谢过程; 5. 血管平滑肌细胞的正调控; 6. 成纤维细胞增殖的正调控; 7. B细胞增殖的正调控; 8. 酪氨酸肽基去磷酸化; 9. 苏氨酸肽基去磷酸化; 10. Ras蛋白信号转导的负调控; 11. ERK1和ERK2级联的负调控; 12. 软骨凝结的负调控; 13. mRNA顺式剪切; 14. MAPK活性失活; 5. 趋化性; 16. 细胞对胰岛素样生长因子刺激的反应; 17. 细胞对激素刺激的反应; 18. 细胞粘附; 19. 骨小梁形成; 20. 血管成熟 |
本研究中共有24 177个基因在KEGG数据库中匹配, 并将富集程度最显著的20条通路进行散点图绘制。HM组与SM组相比, 上调的DEGs富集到的信号通路主要与能量代谢和信号转导相关, 包括心肌收缩、氧化磷酸化信号通路、黏着斑、ECM-受体相互作用、AGE-RAGE、MAPK、心肌细胞中的肾上腺素能等信号通路, 而下调的DEGs富集的信号通路主要为真核生物核糖体的发生、蛋白酶体、内质网中的蛋白加工、RNA转运以及泛素介导的蛋白水解等(图 5)。
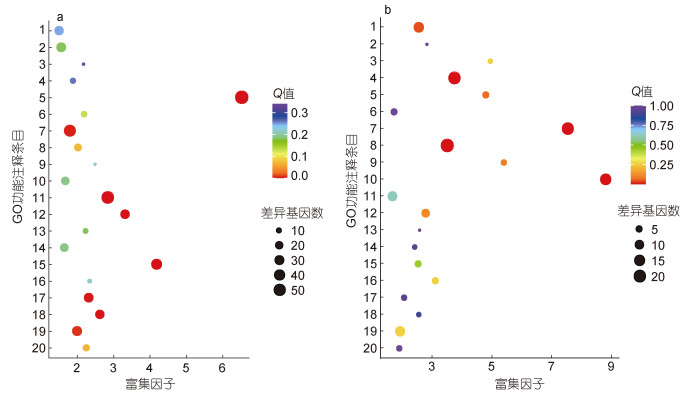 |
| 图 5 肌肉组织差异表达基因KEGG功能富集分布 Fig. 5 KEGG enrichment analysis of differentially expressed genes in muscle tissue 注: a. 上调基因KEGG Pathway富集分布: 1. 紧密连接; 2. 肌动蛋白细胞骨架的调控; 3. 丙酮酸代谢; 4. p53信号通路; 5. 氧化磷酸化; 6. 线粒体-吞噬; 7. MAPK信号通路; 8. GnRH信号通路; 9. 乙醛酸和羧酸代谢; 10. 乙醛酸和二羧酸代谢; 11. 黏着斑; 12. ECM-受体相互作用; 13. Cys-Met代谢; 14. 细胞黏附分子(CAMs); 15. 心肌收缩; 16. 精氨酸和脯氨酸代谢; 17. 磷脂信号通路; 18. AGE−RAGE信号通路; 19. 心肌细胞中的肾上腺素能信号通路; 20. 脂肪细胞因子信号通路。b. 下调基因KEGG Pathway富集分布: 1. 泛素介导的蛋白水解; 2. 萜类主干生物合成; 3. 硒化物代谢; 4. RNA转运; 5. RNA聚合酶; 6. RNA降解; 7. 真核生物核糖体发生; 8. 内质网中的蛋白加工; 9. 蛋白质外排; 10. 蛋白酶体; 11. mTOR通路; 12. 线粒体吞噬–动物; 13. 错配修复; 14. 同源重组; 15. 谷胱甘肽代谢; 16. 果糖和甘露糖代谢; 17. 细胞铁死亡; 18. 自噬-其他; 19. 自噬-动物; 20. 氨基酸和核苷酸代谢 |
随机选取转录组数据中8个DEGs做RT-qPCR验证。RT-qPCR所得到的基因表达情况与转录组获得的基因表达趋势一致, 说明转录组结果可靠(图 6)。
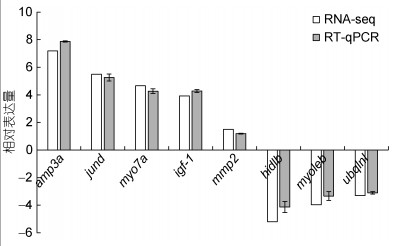 |
| 图 6 RNA-Seq与RT-qPCR的基因表达比较分析 Fig. 6 Comparative analysis of differentially expressed genes of RNA-Seq and RT-qPCR 注: amp3a: 腺苷单磷酸脱氨酶3a; jund: jund原癌基因; myo7a: 肌球蛋白-7型; igf-1: 胰岛素样生长因; mmp2: 基质金属蛋白酶; hid1b: hid1域包含b; myo1eb: 肌球蛋白I Eb; ubqln1: 泛素1 |
抗流分组后, HM组和SM组大黄鱼均出现死亡情况, 但SM组的死亡率在各个时间点均极显著高于HM组(P < 0.01)。其中SM组在抗流分组后12 h内就出现死亡现象, 48 h内累计死亡率达到了18.00%, 而HM组在抗流试验24 h内才出现死亡情况, 48 h内累计死亡率为5.33% (表 4)。
| 项目 | 时间/h | |||
| 0 | 12 | 24 | 48 | |
| HM组累计死亡率/% | 0 | 0 | 2.00±0.00 | 5.33±1.15 |
| SM组累计死亡率/% | 0 | 11.67±1.53* | 14.67±2.31* | 18.00±2.00* |
| 注: *表示同列数据间存在显著差异(P < 0.05) | ||||
本研究采用RNA-Seq技术对比揭示了不同抗流能力大黄鱼群体间肌肉组织的基因表达差异, 为进一步挖掘定位大黄鱼抗流性状关键控制基因奠定了重要基础。通过DEGs聚类分析的结果发现, 抗流弱的SM组DEGs并没有全部聚类在一起, 个体间差异较为明显, 其原因可能与SM组个体间的抗流能力存在较大差异有关。在本实验条件下, SM组大黄鱼其抗流时间的跨度从0~5 min, 换言之, 有的个体抗流的时间可以维持将近5 min, 但有的个体其抗流的时间可能不足1 min, 甚至个别个体的抗流时间仅有20~30 s, 由此导致了SM组个体间的抗流能力差异较大; 而抗流能力强的HM组中, 所有个体的抗流时间均在30 min以上, 意味着所有个体均具有较强的抗流能力, 个体间的抗流能力差异不大, 所以HM组DEGs聚类分析的结果显示其一致性较好。众所周知, 鱼类表型性状受遗传与环境的双重控制(Jonsson et al, 2019; Raffini et al, 2020; Downie et al, 2021), 本研究中大黄鱼的来源相同, 因此可以断定不同大黄鱼个体间抗流能力的差异主要受遗传因素的影响, SM组个体间的抗流能力存在较大差异进而导致了DEGs的表达模式未能呈现出较好的一致性。
3.2 DEGs的GO富集分析肌球蛋白、肌钙蛋白复合物是构成肌肉结构的关键组成部分, 肌节是肌肉收缩的基本单位, 参与各种类型的细胞运动和细胞骨架的维持(Drazic et al, 2018; Ochiai et al, 2020)。本研究发现, 不同抗流能力大黄
鱼群体间的DEGs主要富集在肌球蛋白、肌钙蛋白复合物、肌节和Rho蛋白信号转导的调控等与肌肉收缩功能有关的条目, 这表明了大黄鱼肌肉组织中肌球蛋白、肌钙蛋白复合物与肌节等重要组分的生理功能与其抗流能力的强弱之间存在密切的关联。这一点在斑马鱼(Danio rerio)和金头鲷(Sparus aurata)的研究中也得到了证实, 其游泳能力与骨骼肌肌球蛋白和肌钙蛋白基因的表达密切相关(Van Der Meulen et al, 2006; Palstra et al, 2020)。当然, 鱼类肌肉组织中重要组分的生理功能与其相关基因的表达不仅与遗传因素相关, 与环境因素同样密切相关。Moya等(2019)指出持续性的游泳会使金头鲷(Sparus aurata)幼鱼肌纤维特征发生明显变化, 使虹鳟(Oncorhynchus mykiss)肌肉收缩发育相关的多个基因表达发生改变(Palstra et al, 2013), 此外, Totland等(1987)研究表明较强的水流条件会使大西洋鲑鱼(Salmo salar)白肌中单位面积的纤维数量减少, 单个肌肉纤维的平均面积增加。本研究中, 实验所用大黄鱼的养殖环境因素是一致的, 因此可以确定, 遗传因素的差异影响了大黄鱼个体间肌肉组织中重要组分的生理功能进而决定了其抗流能力的强弱。
3.3 DEGs的KEGG通路分析心肌收缩对鱼类维持正常的生理活动具有重要作用(Singh et al, 2016; Kubly et al, 2019; Nabeebaccus, 2022), 周盈颖(2001)发现心肌收缩受心肌细胞中肾上腺素能信号通路的调控。本研究发现, 在抗流能力强的大黄鱼肌肉组织中心肌收缩相关基因的表达显著上调, 表明大黄鱼心肌收缩功能与其抗流能力间存在正相关关系。心输出量和血氧含量是鱼类游泳能力的重要指标, 持续游泳中鱼类通过心输出量和血氧含量的改变影响能量代谢和氧气利用, 来维持细胞稳态。Castro等(2013)发现大西洋鲑鱼的游泳训练可以通过激活特定基因来诱导心脏的肥大和增生参与对心脏生长的调节。反过来, 心脏生长可以潜在地增加心输出量, 促进氧气和营养物质的输送。Li等(2023)利用临界游泳速度对筛选得到的快速和慢速游泳的大黄鱼幼鱼进行转录组分析发现差异基因主要富集在氧化磷酸化、心肌收缩等途径, 说明游泳过程中大黄鱼通过增加心输出量影响能量代谢和氧气利用, 来维持细胞稳态。同样地, 具有较强游泳能力的蓝鳍金枪鱼(Thunnus orientalis), 强大的心肌收缩能力是其在快速游泳状态下保持心室充盈、维持心输出量的重要生理基础(Ciezarek et al, 2020)。除此之外, 较强的水流条件也会引起鱼类新陈代谢的显著变化, 进而引起机体能量需求的增加(袁喜等, 2012; Schreck et al, 2016; Yu et al, 2022; 柴若愚等, 2023)。研究已证实, 氧化磷酸化信号通路不仅与ATP的生成途径有关, 还是能量代谢的重要枢纽(Lin et al, 2020; Su et al, 2020)。本研究中, HM组大黄鱼肌肉组织中参与氧化磷酸化信号通路的基因表达显著上调, 这些基因编码的产物已证实在呼吸链电子传递过程中发挥重要作用(Neel et al, 2020), 推测抗流组个体中氧化磷酸化产生的ATP是其具有较强抗流能力的能量基础。黏着斑是细胞外基质和细胞内骨架之间联系的桥梁, 对细胞运动有重要作用(Moulder et al, 2010; Li et al, 2019; Mackay et al, 2021), 而ECM-受体相互作用信号通路可参与调控细胞生长分化(Kanchanawong et al, 2023)。本研究结果显示, 抗流能力强的大黄鱼肌肉组织中黏着斑与ECM-受体相互作用信号通路的相关基因同样显著上调。
3.4 抗流分组后累计死亡率分析应激是影响鱼类生命活动的重要影响因素之一(Nitz et al, 2020)。于丽娟等(2014)发现不同水流速度下的中华倒刺鲃肌肉(Spinibarbus sinensis)自由基代谢会发生改变, 而自由基代谢紊乱会引起氧化/还原系统的失衡, 进而导致鱼体的应激损伤。本研究中SM组大黄鱼肌肉组织中泛素介导的蛋白水解、蛋白酶体、内质网中的蛋白加工等与氧化应激相关的信号通路基因明显上调, 表明水流刺激显著诱发了SM组大黄鱼的应激反应。娄宇栋等(2019)在对美国红鱼(Sciaenops ocellatus)肝脏转录组分析中同样发现, 高流速下美国红鱼会触发细胞内的胁迫相关基因PKA、Wnt的上调, 这表明过快的水流会引起鱼类的氧化应激反应。内质网作为外界环境的压力感受器(Yoo et al, 2017), 可以启动机体通过自噬或细胞凋亡来清除损伤的细胞。本研究内质网中蛋白加工信号通路被显著富集, 而其在介导错误折叠的蛋白进入胞质中发挥重要作用(Iurlaro et al, 2016), 这也进一步表明了水流的刺激导致了SM组大黄鱼机体内产生了应激损伤, 此是导致SM组累计死亡率明显高于HM组的主要原因之一。
4 结论本研究基于不同抗流能力大黄鱼群体间肌肉组织DEGs的GO功能与KEGG通路富集分析, 推测抗流能力强的大黄鱼可能是通过改变肌肉收缩的方式及能量代谢来提高机体应对水流的能力。结合死亡率等表型数据的分析, 进一步印证了较强的抗流能力可显著提高大黄鱼在强水流刺激下的成活率, 研究结果为深入挖掘抗流相关基因提供了基础信息, 并为培育适宜深远海养殖的大黄鱼抗流新品种奠定了理论基础。
于丽娟, 李秀明, 易建华, 等, 2014. 不同水流速度对中华倒刺鲃幼鱼自由基代谢的影响[J]. 中国水产科学, 21(1): 101-107. |
农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会, 2021. 中国渔业统计年鉴2021[M]. 北京: 中国农业出版社, 18-26.
|
邹国华, 宋炜, 谢正丽, 2021. 大黄鱼深远海大型围栏养殖技术[J]. 中国水产: 57-60. |
周盈颖, 2001. 心肌细胞β1和β2肾上腺素能信号传导系统的区别[J]. 山西医科大学学报, 32(S1): 31-34. |
娄宇栋, 冯建, 何娇娇, 等, 2019. 流速胁迫对美国红鱼的转录特性研究[J]. 浙江海洋大学学报(自然科学版), 38(1): 13-22, 29. |
袁喜, 涂志英, 韩京成, 等, 2012. 流速对细鳞裂腹鱼游泳行为及能量消耗影响的研究[J]. 水生生物学报, 36(2): 270-275. |
柴若愚, 尹恒, 霍润明, 等, 2023. 水流速度对黑鲷和美国红鱼续航游泳能力及生理代谢的影响[J]. 水生生物学报, 47(5): 723-731. DOI:10.7541/2023.2022.0186 |
徐皓, 刘晃, 徐琰斐, 2021. 我国深远海养殖发展现状与展望[J]. 中国水产, (6): 36-39. |
徐鹏, 柯巧珍, 苏永全, 等, 2022. 大黄鱼种质资源保护与利用现状及建议[J]. 水产学报, 46(4): 674-682. |
韩承义, 吴雄飞, 许斌福, 等, 2022. 2020年中国大黄鱼产业现状分析及发展建议[J]. 渔业研究, 44(4): 395-406. |
曾令清, 付成, 付世建, 2018. 鲫幼鱼游泳运动能力的个体变异与表型关联[J]. 水生生物学报, 42(3): 561-570. |
曾霖, 王永红, 宋炜, 等, 2022. 基于转录组解析铜驯化对低温胁迫下大黄鱼氧化损伤的影响[J]. 中国水产科学, 29(10): 1425-1436. |
BAI Y L, WANG M, ZHAO J, et al, 2022. Comparative transcriptome analysis reveals immunoregulation mechanism of lncRNA-mRNA in gill and skin of large yellow croaker (Larimichthys crocea) in response to Cryptocaryon irritans infection[J]. BMC Genomics, 23(1): 206. DOI:10.1186/s12864-022-08431-w |
CAO L, NAYLOR R, HENRIKSSON P, et al, 2015. China's aquaculture and the world's wild fisheries[J]. Science, 347(6218): 133-135. DOI:10.1126/science.1260149 |
CASTRO V, GRISDALE-HELLAND B, HELLAND S J, et al, 2013. Cardiac molecular-acclimation mechanisms in response to swimming-induced exercise in Atlantic salmon[J]. PLoS One, 8(1): e55056. DOI:10.1371/journal.pone.0055056 |
CIEZAREK A, GARDNER L, SAVOLAINEN V, et al, 2020. Skeletal muscle and cardiac transcriptomics of a regionally endothermic fish, the Pacific bluefin tuna, Thunnus orientalis[J]. BMC Genomics, 21(1): 642. DOI:10.1186/s12864-020-07058-z |
DOWNIE A T, LEIS J M, COWMAN P F, et al, 2021. The influence of habitat association on swimming performance in marine teleost fish larvae[J]. Fish and Fisheries, 22(6): 1187-1212. DOI:10.1111/faf.12580 |
DRAZIC A, AKSNES H, MARIE M, et al, 2018. NAA80 is actin's N-terminal acetyltransferase and regulates cytoskeleton assembly and cell motility[J]. Proceedings of the National Academy of Sciences of the United States of America, 115(17): 4399-4404. |
IURLARO R, MUÑOZ-PINEDO C, 2016. Cell death induced by endoplasmic reticulum stress[J]. The FEBS Journal, 283(14): 2640-2652. |
JONSSON B, JONSSON N, 2019. Phenotypic plasticity and epigenetics of fish: embryo temperature affects later-developing lift-history traits[J]. Aquatic Biology, 28: 21-32. |
KANCHANAWONG P, CALDERWOOD D A, 2023. Organization, dynamics and mechanoregulation of integrin-mediated cell-ECM adhesions[J]. Nature Reviews Molecular Cell Biology, 24(2): 142-161. |
KUBLY K L, STECYK J A W, 2019. Contractile performance of the Alaska blackfish (Dallia pectoralis) ventricle: Assessment of the effects of temperature, pacing frequency, the role of the sarcoplasmic reticulum in contraction and adrenergic stimulation[J]. Comparative Biochemistry and Physiology: Part A: Molecular & Integrative Physiology, 238: 110564. |
LI S P, LIU X, LIN T T, et al, 2023. Muscle fiber plasticity, stress physiology, and muscle transcriptome determine the inter-individual difference of swimming performance in the large yellow croaker (Larimichthys crocea)[J]. Aquaculture, 567: 739247. |
LI G M, SONG X B, LI R, et al, 2019. Zyxin-involved actin regulation is essential in the maintenance of vinculin focal adhesion and chondrocyte differentiation status[J]. Cell Proliferation, 52(1): e12532. |
LIN G M, AUDIRA G, JUNIARDI S, et al, 2020. Metatranscriptomic analysis reveals co-expression pattern of mitochondrial oxidative phosphorylation (OXPHOS) genes among different species of bony fishes in muscle tissue[J]. Journal of King Saud University-Science, 32(7): 3084-3090. |
LUO S Y, GAO X M, DING J, et al, 2019. Transcriptome sequencing reveals the traits of spermatogenesis and testicular development in large yellow croaker (Larimichthys crocea)[J]. Genes, 10(12): 958. |
MA R, MENG Y Q, ZHANG W B, et al, 2020. Comparative study on the organoleptic quality of wild and farmed large yellow croaker Larimichthys crocea[J]. Journal of Oceanology and Limnology, 38(1): 260-274. |
MACKAY L, LEHMAN E, KHADRA A, 2021. Deciphering the dynamics of lamellipodium in a fish keratocytes model[J]. Journal of Theoretical Biology, 512: 110534. |
MOULDER G L, CREMONA G H, DUERR J, et al, 2010. α-actinin is required for the proper assembly of Z-disk/focal-adhesion-like Structures and for efficient locomotion in Caenorhabditis elegans[J]. Journal of Molecular Biology, 403(4): 516-528. |
MOYA A, TORRELLA J R, FERNÁNDEZ-BORRÀS J, et al, 2019. Sustained swimming enhances white muscle capillarisation and growth by hyperplasia in gilthead sea bream (Sparus aurata) fingerlings[J]. Aquaculture, 501: 397-403. |
MU Y N, LI W R, WU B, et al, 2020. Transcriptome analysis reveals new insights into immune response to hypoxia challenge of large yellow croaker (Larimichthys crocea)[J]. Fish & Shellfish Immunology, 98: 738-747. |
NABEEBACCUS A, 2022. Biochemistry and physiology of cardiac muscle[J]. Medicine, 50(6): 327-332. |
NEEL E M, SCHARLAU M, MILLETT F, 2020. Regulation of electron transfer from cytochrome C to cytochrome C oxidase by phosphorylation of CC THR-28[J]. Biophysical Journal, 118(3): 608a-609a. |
NITZ L F, PELLEGRIN L, MALTEZ L C, et al, 2020. Temperature and hypoxia on oxidative stress responses in pacu Piaractus mesopotamicus[J]. Journal of Thermal Biology, 92: 102682. |
OCHIAI Y, OZAWA H, 2020. Biochemical and physicochemical characteristics of the major muscle proteins from fish and shellfish[J]. Fisheries Science, 86(5): 729-740. |
PALSTRA A P, BELTRAN S, BURGERHOUT E, et al, 2013. Deep RNA Sequencing of the skeletal muscle transcriptome in swimming fish[J]. PLoS One, 8(1): e53171. |
PALSTRA A P, ROQUE A, KRUIJT L, et al, 2020. Physiological effects of water flow induced swimming exercise in seabream Sparus aurata[J]. Frontiers in Physiology, 11: 610049. |
RAFFINI F, SCHNEIDER R F, FRANCHINI P, et al, 2020. Diving into divergence: differentiation in swimming performances, physiology and gene expression between locally-adapted sympatric cichlid fishes[J]. Molecular Ecology, 29(7): 1219-1234. |
SCHRECK C B, TORT L, FARRELL A P, et al, 2016. Biology of stress in fish[M]. New York: Academic Press, 251-294.
|
SINGH A R, SIVADAS A, SABHARWAL A, et al, 2016. Chamber specific gene expression landscape of the zebrafish heart[J]. PLoS One, 11(1): e0147823. |
SU F, ZHOU F F, ZHANG T, et al, 2020. Quantitative proteomics identified 3 oxidative phosphorylation genes with clinical prognostic significance in gastric cancer[J]. Journal of Cellular and Molecular Medicine, 24(18): 10842-10854. |
TOTLAND G K, KRYVI H, JØDESTØL K A, et al, 1987. Growth and composition of the swimming muscle of adult Atlantic salmon (Salmo salar L.) during long-term sustained swimming[J]. Aquaculture, 66(3/4): 299-313. |
VAN DER MEULEN T, SCHIPPER H, VAN DEN BOOGAART J G M, et al, 2006. Endurance exercise differentially stimulates heart and axial muscle development in zebrafish (Danio rerio)[J]. American Journal of Physiology-Regulatory, Integrative and Comparative Physiology, 291(4): R1040-R1048. |
YOO Y S, HAN H G, JEON Y J, 2017. Unfolded protein response of the endoplasmic reticulum in tumor progression and immunogenicity[J]. Oxidative Medicine and Cellular Longevity, 2017: 2969271. |
YU X M, SOUSA V F M F, OLIVEIRA B M, et al, 2022. Induced sustained swimming modifies the external morphology, increasing the oxygen-carrying capacity and plasma lactate levels of juvenile gilthead seabream (Sparus aurata) without changing fish performance or skeletal muscle characteristics[J]. Aquaculture, 560: 738503. |
ZHANG Y B, SHEN W L, DING J, et al, 2022. Comparative transcriptome analysis of head kidney of Aeromonas hydrophila-infected hypoxia-tolerant and normal large yellow croaker[J]. Marine Biotechnology, 24(6): 1039-1054. |
ZHAO J, ZHOU T, BAI H Q, et al, 2021. Genome-wide association analysis reveals the genetic architecture of parasite (Cryptocaryon irritans) resistance in large yellow croaker (Larimichthys crocea)[J]. Marine Biotechnology, 23(2): 242-254. |
ZHOU Z X, HAN K H, WU Y D, et al, 2019. Genome-wide association study of growth and body-shape-related traits in large yellow croaker (Larimichthys crocea) using ddRAD sequencing[J]. Marine Biotechnology, 21(5): 655-670. |
 2023, Vol. 54
2023, Vol. 54


