
中国海洋湖沼学会主办。
文章信息
- 姜之信, 李慷, 巴旭冰, 曾祥标, 刘利平. 2023.
- JIANG Zhi-Xin, LI Kang, BA Xu-Bing, ZENG Xiang-Biao, LIU Li-Ping. 2023.
- 中国近海洄游日本鳗鲡(Anguilla japonica)玻璃鳗肠道微生物菌群分析
- ANALYSIS OF INTESTINAL MICROFLORA OF JAPANESE EEL (ANGUILLA JAPONICA) IN MIGRATION ALONG CHINA OFFSHORE
- 海洋与湖沼, 54(6): 1728-1736
- Oceanologia et Limnologia Sinica, 54(6): 1728-1736.
- http://dx.doi.org/10.11693/hyhz20230300065
文章历史
- 收修改稿日期:2023-03-18
2. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室 上海 201306;
3. 上海海洋大学 上海水产养殖工程技术研究中心 上海 201306
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China;
3. Shanghai Engineering Research Center of Aquaculture, Shanghai Ocean University, Shanghai 201306, China
日本鳗鲡(Anguilla japonica)是东亚重要的经济鱼类之一, 在我国、日本以及韩国广泛养殖(刘利平等, 2017)。生活史复杂, 按照生长阶段可分为: 受精卵、柳叶鳗、玻璃鳗、线鳗、黄鳗、银鳗等多个阶段(Manabe et al, 2011; Wahlberg et al, 2014)。自然界中捕捞到柳叶鳗的概率极低, 且多为偶然性获得, 截至目前, 养殖成鳗所需的苗种(即玻璃鳗)完全依赖于天然捕捞, 鳗苗的大量捕捞导致日本鳗鲡资源锐减(Tsukamoto et al, 2009), 在2014年国际自然保护联盟(IUCN)发表的“红色名录”中, 日本鳗鲡被认定为濒危物种。虽然Tanaka研究团队于2003年通过人工促熟获得少量变态成功的玻璃鳗(Tanaka et al, 2003), 但幼鱼成活率低、饵料成本高, 仍无法达到工厂化养殖的要求, 其重要因素在于开口饵料问题没有得到有效的解决。研究表明, 自然界中的初孵仔鱼可能以海洋颗粒有机物POM为食, 比如海洋雪(Miller et al, 2013), 通常认为海洋雪是深海中由有机物所组成的碎屑, 其成分包括: 已死或将死的动植物(浮游生物)、原生生物(如硅藻)、细菌、粪便颗粒、泥沙和尘土等(Alldredge et al, 1988)。由于自然界中的柳叶鳗获得概率极低, 目前仍无足够的样品数量支持系统性的研究, 因此对其食物来源仍有争议。
鱼类肠道菌群可通过分泌消化酶等方式参与营养物质的代谢吸收, 而不同食性的鱼类肠道菌群中存在不同的标志性微生物, 因此可通过研究野生鳗鲡幼鱼的优势肠道菌群及其功能, 推测其摄食习惯, 有助于确定其早期食物组成和促进开口饵料的研发。近年来, 高通量测序技术在医学(Zhang et al, 2021)、水环境(Yang et al, 2022)以及家禽(Zhao et al, 2018)等领域得到了广泛的应用, 是目前研究生境内微生物最常用的技术手段, 越来越多的学者将高通量测序技术应用于水产动物肠道微生物研究, Hsu等(2018)对成年日本鳗鲡、花鳗鲡(A. marmorata)以及太平洋双色鳗鲡(A. bicolor pacifica)的肠道进行高通量测序, 发现鳗鲡肠道优势菌属为鲸杆菌属(Cetobacterium)、梭菌属(Clostridium)、希瓦氏菌属(Shewanella)、不动杆菌属(Acinetobacter)和拟杆菌属(Bacteroides)等。张金花等(2017)研究发现, 斑马鱼肠道优势菌门为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、梭菌属和蓝细菌(Cyanobacteria)。
本研究通过对采集的野生玻璃鳗肠道微生物进行高通量测序, 分析不同洄游采样点玻璃鳗肠道菌群结构特点, 明确肠道菌群多样性变化及其肠道优势菌, 分析玻璃鳗肠道消化功能及为可能的食物组成提供科学依据。
1 材料与方法 1.1 玻璃鳗样本采集根据玻璃鳗由南至北的洄游习性, 于2021年3~4月依次在浙江省台州市(T)、浙江省舟山市(D)、江苏省盐城市(Y)用定置网捕集野生玻璃鳗样品(表 1), 每个采样点随机选取60条体表无伤、活力良好的玻璃鳗进行体质量、体长的测量, 随后在无菌环境中进行解剖, 将已去除内容物的肠道保存在1.5 mL离心管中放于–80 ℃冰箱, 用于后续试验分析。
| 项目 | 台州市(T) | 舟山市(D) | 盐城市(Y) |
| 经度 | 121.691 293°E | 122.121 534°E | 121.102 222°E |
| 纬度 | 28.828 928°N | 30.605 263°N | 32.852 038°N |
每个采样点随机选取6条肠道样品, 每2条肠道样品进行混样, 用于DNA提取和测序。样品DNA提取参照海洋动物组织基因组DNA提取试剂盒(TIANGEN)说明书进行, 所提DNA浓度和纯度经检测(Nanodrop 2000)合格后送至广东美格基因科技有限公司进行高通量测序。
1.3 高通量测序基因扩增及测序过程如下: 根据16S rRNA基因序列特点和Illumina MiSeq平台测序要求, 针对V3+V4区域设计特异引物338F: 5′-ACTCCTACG GGAGGCAGCA-3′, 806R: 5′-GGACTACHVGGGTW TCTAAT-3′进行PCR扩增(引物片段大小470 bp, 带有barcode)。PCR反应体系(50 μL)为2x Premix Taq 25 μL, 正反引物(10 mmol/L)各1 μL, 样品DNA (20 ng/μL) 50 ng, 补充Nuclease-free water至50 μL。PCR反应条件: 94 ℃ 5 min; 94 ℃ 30 s, 52 ℃ 30 s, 72 ℃ 30 s, 30个循环; 72 ℃ 10 min; 4 ℃保存直至使用。使用E.Z.N.A.® Gel Extraction Kit凝胶回收试剂盒回收PCR混合产物, T.E缓冲液洗脱回收目标DNA片段。建库按照NEBNext® ULTRATM DNA Library Prep Kit for Illumina®试剂盒标准流程进行建库操作, 完成后进行上机测序。
1.4 数据分析实验所得的形态学数据采用EXCEL 2019和SPSS 24.0软件进行处理和单因素方差分析, 差异显著者进行LSD多重比较, 设显著性水平为P < 0.05。
对原始数据(Raw reads)进行双端去杂、拼接和去嵌合体等质控处理, 获得优质的有效序列(Valid tags), 以相似度97%的阈值为基础来进行可操作分类单元归类, 对OTUs代表序列进行物种注释, 并与Silva数据库序列信息比对, 获得生物注释。利用序列最少的样本对OTUs丰度进行归一化处理, 进行Alpha多样性分析, 最后通过PICRUSt 2预测三个采样点玻璃鳗肠道菌群功能, 根据COG数据库进行功能推断, 以上生物信息分析通过广州美格生物云在线平台(http://cloud.magigene.com/yomics)进行。
2 结果与分析 2.1 玻璃鳗的形态学指标随着采样点纬度的增加, 玻璃鳗的体长和体质量呈逐渐增长趋势, 但相邻采样点间体长、体质量差异不显著(图 1, P > 0.05)。盐城玻璃鳗样品平均体长[(56.49±0.13) mm]大于舟山与台州的样品[(55.48± 0.29) mm, (54.98±0.05) mm]; 玻璃鳗平均体质量的结果相似, 由大至小分别为盐城样品[(0.12±0.01) g]、舟山样品[(0.11±0.01) g]和台州样品[(0.10±0.08) g]。
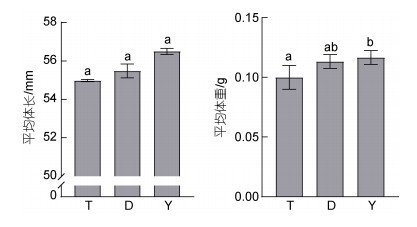 |
| 图 1 洄游路径上不同采样点(T, D, Y)玻璃鳗样品的平均体长和平均体重(n=60) Fig. 1 Average body weight and body length of glass eel from different sampling sites (T, D, Y) along the migration route (n=60) 注: 字母T、D、Y代表采样点台州、舟山、盐城; 图中不同字母表示差异显著(P < 0.05) |
本研究基于16S rRNA高通量测序技术对日本鳗鲡玻璃鳗肠道微生物V3+V4区进行分析, 9组样品总计获得122 241条有效数据(表 1), 根据97%的相似度进行OTU分类, 获得327个OTUs, 台州、舟山和盐城分别得到112、152、171个OTUs, 其中三个采样点共有的OTUs数目为33个, 共涉及19门33纲85目133科184属。
稀释性曲线可以直观地反映高通量测序数据的准确性以及菌群的多样性, 本实验对所有样本多种α多样性指数进行了分析, 具体结果如图 2所示, Shannon指数曲线逐渐趋于平坦, 虽然有的分组数据量较少, 但都已经过转折点进入平台期, 继续增加数据量, 只会出现少量新物种, 说明测序数据量合理, 实验结果可信。覆盖率指数曲线显示所有样本的覆盖率均在95%以上, 表明测序数据具有较好的覆盖性。
| 采样点 | 原始数据 | 有效数据 | OTU数目 | 香农指数 | 覆盖率 |
| T | 43 089 | 42 600 | 112 | 3.499 0 | 0.988 5 |
| D | 42 761 | 42 000 | 152 | 4.494 2 | 0.970 8 |
| Y | 38 424 | 37 641 | 171 | 4.734 6 | 0.993 2 |
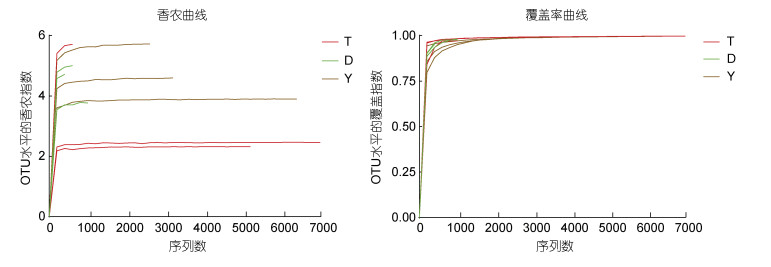 |
| 图 2 洄游路径上不同采样点(T, D, Y)玻璃鳗肠道微生物的OTU α多样性指数 Fig. 2 OTU-level α diversity index curves of intestinal microorganisms of glass eel from different sampling sites (T, D, Y) along the migration route 注: 字母T、D、Y代表采样点台州、舟山、盐城 |
根据物种注释结果在门水平对物种相对丰度进行统计分析, 由图 3可知, 相对丰度居前5的物种包括变形菌门、放线菌门、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)以及绿弯菌门(Chloroflexi)。三个采样点均以变形菌门和放线菌门为优势菌门, 其中变形菌门、放线菌门、拟杆菌门、厚壁菌门的总丰度占细菌总量的95%以上, 舟山采样点的厚壁菌门以及盐城采样点的拟杆菌门丰度较其他两个采样点高, 但彼此间差异不显著(P > 0.05)。
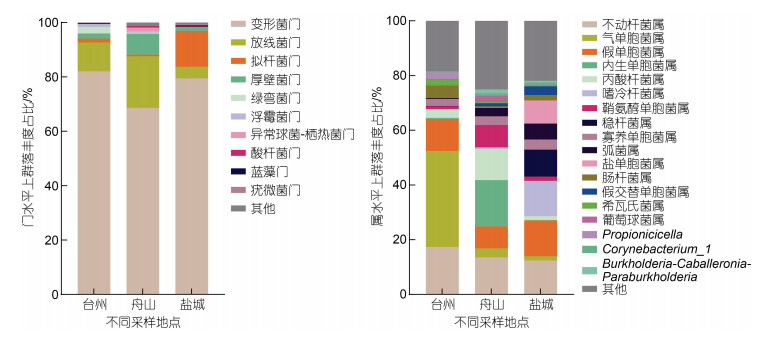 |
| 图 3 洄游路径上不同采样点(台州、舟山、盐城)玻璃鳗肠道细菌主要类群的组成 Fig. 3 Community composition of major bacterial taxa of intestinal microorganisms of glass eel from different sampling sites (Taizhou, Zhoushan, Yancheng) along the migration route 注: 相对占比低于0.01%的菌群以及数据库中没有找到对应于该序列分类信息的菌群均归类于其他 |
在属水平上对每个样品的菌群结构及分布进行统计分析, 样品组丰度前5的菌属包括: 变形杆菌门下的不动杆菌属、气单胞菌属(Aeromonas)、假单胞菌属(Pseudomonas)、内生单胞属(Endozoicomonas), 放线菌门下的丙酸杆菌属(Cutibacterium)等菌属。台州微生物以不动杆菌(17.33%)、气单胞菌(35.09%)、假单胞菌(11.64%)、肠杆菌属(Enterobacter, 4.48%)、丙酸杆菌(2.97%)的丰度较高, 为优势菌属; 舟山以不动杆菌(13.52%)、内生单胞属(16.91%)、丙酸杆菌(11.39%)、假单胞菌属(8.06%)、鞘氨醇单胞菌属(Sphingomonas, 7.88%)的丰度较高, 为优势菌属; 盐城以不动杆菌(12.36%)、假单胞菌(13.03%)、嗜冷杆菌属(Psychrobacter, 12.91%)、稳杆菌属(Empedobacter, 9.88%, 属于拟杆菌门)、盐单胞菌属(Halomonas, 8.48%)的丰度较高, 为优势菌属。三个采样点共享以不动杆菌属和假单胞菌属为优势菌群的微生物, 属于玻璃鳗的固定优势菌群。
2.4 肠道共有及特有微生物菌群分析通过对不同洄游采样点玻璃鳗肠道微生物不同分类等级进行分析, 得出各个采样点中玻璃鳗肠道共有和特有的肠道菌群的种类和数量, 便于我们比较不同洄游采样点的相似性和差异性。如图 4所示, 台州(T)、舟山(D)、盐城(Y)玻璃鳗肠道微生物菌群样品中共有的OTUs数量为33种, 台州特有的OTU数目为61种, 舟山特有的OTUs数目为81种, 盐城特有的OTUs数目为110种。
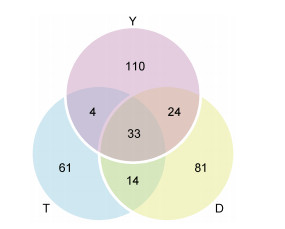 |
| 图 4 洄游路径上不同采样点(T, D, Y)玻璃鳗肠道内共有和独特的OTU数目 Fig. 4 The number of shared and unique OTU in the intestinal microorganisms of glass eel from different sampling sites (T, D, Y) along the migration route 注: 字母T、D、Y代表采样采样点台州、舟山、盐城 |
不同洄游采样点间共享的属水平的微生物菌群包括不动杆菌、假单胞菌、丙酸杆菌、气单胞菌等菌群(图 5)。不动杆菌属以及假单胞菌属在三个采样点玻璃鳗肠道中均占有较高的丰度, 气单胞菌属仅在台州采样点占有较高的丰度(台州、舟山、盐城占比分别为35.09%、3.27%、1.58%), 而舟山采样点内生单胞菌属以及丙酸杆菌属丰度较其余两地高, 盐城采样点也有高于其余两地的菌群, 如嗜冷杆菌属、稳杆菌属(Empedobacter)等。因此, 玻璃鳗在洄游过程中共享一个核心菌群, 但不同采样点间菌群的占比丰度略有差异。
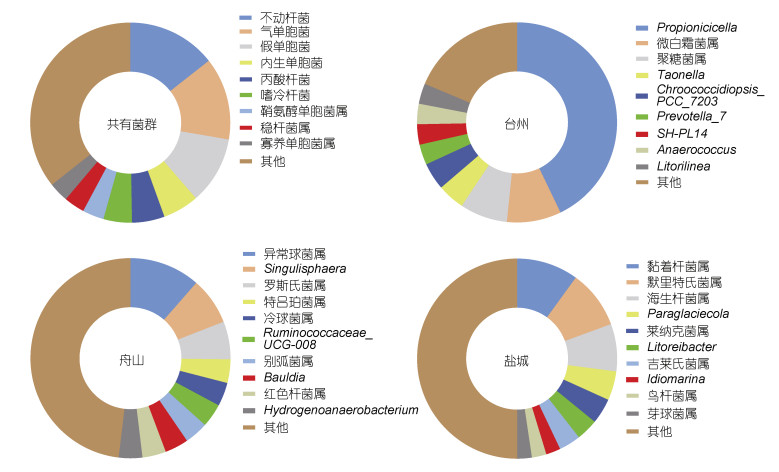 |
| 图 5 洄游路径上不同采样点(T, D, Y)玻璃鳗肠道内共有和独特的微生物菌群 Fig. 5 Shared and unique microflora in the intestine of glass eel from different sampling sites (T, D, Y) along the migration route 注: 字母T、D、Y代表采样采样点台州、舟山、盐城 |
同时, 不同洄游采样点玻璃鳗肠道中具有多种特有的肠道菌群。如图 5所示, 台州特有的肠道菌群为产丙酸细胞菌属(Propionicicella)、微白霜菌属(Micropruina)、聚糖菌属(Defluviicoccus)等; 舟山丰度较高的肠道菌群为异常球菌属(Deinococcus)、Singulisphaera、罗斯氏菌属(Rothia)等; 盐城以默里特氏菌属(Moritella)、Tenacibaculum、海生杆菌属(Marivita)等特有菌属的丰度较高。同样, 三个采样点玻璃鳗肠道中有多种菌群仅在T和D采样点肠道中存在, 如放线菌门下的放线菌属(Actinomyces)和厚壁菌门下的链球菌属(Streptococcus); 而乳杆菌(Lactobacillus)、盐单胞菌属等仅在D和Y采样点肠道中发现, 微杆菌属(Microbacterium)、假苍白杆菌(Pseudochrobactrum)等多个菌群在T和Y中占据较低的丰度, 在D采样点并未检测到。不同采样点玻璃鳗肠道中特有的微生物菌群种类多样, 但其菌群丰度非常低, 占比均未超过1%。
2.5 肠道菌群功能预测本研究利用PICRUSt2对三个采样点的玻璃鳗肠道微生物进行功能预测分析。在一级功能层面上, 各采样点玻璃鳗肠道菌群的主要功能包括代谢(Metabolism)、细胞加工(Cellular processes and signaling)、信息存储和处理(Information storage and processing)和缺失的功能描述(Poorly characterized)等; 在二级功能层面上, 对相对丰度大于等于0.01%的肠道菌群进行功能注释和聚类, 共获得24个功能类别(图 6)。结果显示, 在代谢类别中, 三个采样点玻璃鳗肠道均以氨基酸运输和代谢(Amino acid transport and metabolism)功能占比最高, 而碳水化合物的转运和代谢(Carbohydrate transport and metabolism)、脂质的转运和代谢(Lipid transport and metabolism)功能分别位于第八位和第十一位。
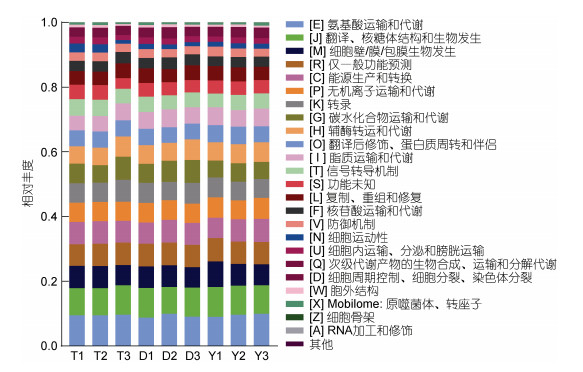 |
| 图 6 不同洄游采样点(T, D, Y)玻璃鳗肠道微生物功能预测图 Fig. 6 Prediction of intestinal microbial function of glass eel from different sampling sites (T, D, Y) along the migration route 注: 字母T、D、Y代表采样采样点台州、舟山、盐城 |
本研究结果显示, 洄游路径上不同采样点玻璃鳗肠道菌群主要由变形菌门、放线菌门、厚壁菌门和拟杆菌门组成。变形菌门是细菌中最大的一门, 也是本试验玻璃鳗肠道丰度最高的菌群, 占比68.7%~ 82.0%, 与大菱鲆(邢孟欣, 2013)和牙鲆仔稚鱼(刘增新, 2017)等鱼类肠道菌群研究结果相似。通过测序发现, 玻璃鳗肠道中的变形菌门包括不动杆菌、气单胞菌以及内生单胞菌属等, 其中三个采样点占比最高的肠道菌属为不动杆菌属(12.4%~17.3%)。Iida等(1984)通过对未进食的日本鳗鲡鳗苗进行饵料投喂实验, 发现随着饵料由颤蚓(Tubifex sp.)逐渐转变为配方饲料时, 不动杆菌属和气单胞菌属占比上升, 可见不动杆菌可能为日本鳗鲡的优势固有菌群, 也是银鲫(Carassius auratusgibelio)、青鱼(Mylopharyngodon piceus)、鳙(Parabramis pekinensis)等淡水鱼中的优势菌属, 可产生抑制鳗弧菌的物质, 有效预防病原菌感染(Spanggaard et al, 2001; 张涵等, 2013), 具有产生胞外蛋白酶的能力(邓智明, 2018)。胞外蛋白酶可直接消化、促进吸收蛋白质, 增强鱼类机体对营养物质的摄取(Ray et al, 2012; 邓智明, 2018), 玻璃鳗肠道中不动杆菌的存在为其降解蛋白质提供可能性。部分假单胞菌属可作为肠道益生菌改善水产动物的营养消化、生长以及提升动物的抗逆能力(Pérez et al, 2010)。研究人员发现, 草鱼和鲫鱼肠道内的假单胞菌可分泌胞外消化酶, 如蛋白酶、脂肪酶、淀粉酶和纤维素酶, 促进了两种鱼对饵料的消化吸收(冯雪等, 2008)。军曹鱼体内也分离出具有较强的产蛋白酶能力的假单胞菌(王瑞旋等, 2008), 由此可见, 假单胞菌的一些种类对营养物质尤其是蛋白质具有分解作用。舟山样品中, 菌群丰度较高的内生单胞菌属(占比16.9%)也同样具有分解蛋白质的功能, 其通常存在于宿主组织内的细胞聚集体中, 参与宿主相关的蛋白质和碳水化合物运输和循环(Neave et al, 2016)。气单胞菌是一类主要存在于鱼类肠道中的纤维素降解菌(Talwar et al, 2018), 尤其是草鱼等草食性鱼类的核心菌群(Wu et al, 2012), 在台州的肠道样品中, 气单胞菌属的丰度占比为35.1%, 显著超过其他两个采样点的菌群丰度, 可能是玻璃鳗在台州摄取较多的浮游植物或受到水环境中微生物的影响导致的这一结果(孟晓林等, 2019)。在虹鳟肠道中分离出可分泌高效蛋白酶的气单胞菌(Koca et al, 2015), 表明部分种类的气单胞菌也具有消化蛋白质的功能。通过对肠道菌群进行功能预测发现, 氨基酸的转运和代谢占据最高的丰度占比(8.7%~10.0%), 证明玻璃鳗在洄游途中可能摄取以蛋白质为主的饵料作为其能量来源。
厚壁菌门含有诸多有益菌, 如乳杆菌和链球菌等, 有助于机体对碳水化合物的利用(Nuriel-Ohayon et al, 2016; 田娟等, 2022)。本研究在玻璃鳗肠道中检测到乳杆菌和链球菌, 但三个采样点略有差异, 其中台州采样点未检测到乳杆菌属, 盐城采样点未发现链球菌属, 而舟山采样点两者均有发现, 但丰度均较低, 占比不到1%。厚壁菌门的明串珠菌属(Leuconostoc)和放线菌门的放线菌属也属于纤维素降解菌(Talwar et al, 2018; 冯雪等, 2018)。有研究表明, 放线菌门中的红球菌属(Rhodococcus)能够分泌大量的活性物质, 可利用有机化合物作为能源和碳源, 同时可以降解藻类物质(李小彩, 2007; 邱孜博等, 2016)。本实验玻璃鳗肠道中以上几种细菌丰度较低, 推测可能是玻璃鳗在洄游过程中对纤维素类饵料的需求较蛋白质类饵料的数量少, 而肠道菌群功能预测结果中, 氨基酸转运与代谢的占比高于碳水化合物和脂质的占比也验证了此观点。受采样点环境的影响, 本实验不同采样点样品有些特异菌属, 此类现象在水生动物中比较常见, 比如罗非鱼养殖, 通过对两种不同养殖环境下的罗非鱼肠道微生物进行比较分析, 发现稻田组罗非鱼肠道菌群丰度较高的为鲸杆菌属、幽门螺旋菌属(Paeniclostrdium)和罗姆布茨菌属(Romboutsia), 而池塘组罗非鱼丰度较高的菌群为鲸杆菌属、肠弧菌属(Enterovibrio)和邻单胞菌属(Plesiomonas) (张婧怡等, 2020)。不同生长环境也会影响鱼类肠道菌群的物种组成及丰度, 因此推测是三个采样点海水环境差异, 导致玻璃鳗肠道中含有较多相对丰度占比很低的特有菌群。
玻璃鳗肠道菌群中除消化功能的细菌外, 优势菌属中也包含部分致病菌属, 如常见的气单胞菌属和不动杆菌属(Thanh et al, 2021; 孙莉, 2015)。气单胞菌属中已报道的维氏气单胞菌(Aeromonas veronii) (杨求华, 2012)和嗜水气单胞菌(Aeromonas hydrophila) (张伟妮, 2010), 以及不动杆菌属中的鲍曼不动杆菌(Acinetobacter baumannii) (孙云等, 2020)便是鳗鲡养殖中常见的致病菌。2019年海南海口某养殖场暴发的美洲鳗细菌性疾病, 死亡率高达80%, 便是由鲍曼不动杆菌引起的(孙云等, 2020)。2010年厦门某日本鳗鲡养殖场出现大面积发病情况, 是维氏气单胞菌引发(杨求华等, 2012)。推测本实验中肠道所含致病菌也可能来源于水体环境。
综上所述, 三个采样点的玻璃鳗共享以不动杆菌和假单胞菌为主的核心菌群, 其在洄游过程中可能主要摄取以蛋白质含量较高的饵料, 对碳水化合物以及脂质成分需求量相对较少。海洋中的浮游植物和浮游动物均含有蛋白质(杨宇峰等, 2000), 且浮游动物蛋白质含量远远高于浮游植物, 因此玻璃鳗在洄游过程中可能更偏向于以浮游动物为摄食对象, 该研究可为今后日本鳗鲡仔稚鱼饵料的研发提供参考。
4 结论本研究表明, 三个采样点玻璃鳗肠道菌群共享不动杆菌属和假单胞菌属等核心菌群, 受采样点环境的影响, 不同采样点玻璃鳗肠道中均含有丰度较高的特殊菌群, 如台州市的气单胞菌、舟山市的内生单胞属和丙酸杆菌以及盐城市的嗜冷杆菌属和稳杆菌属等, 不动杆菌属、假单胞菌属、气单胞菌属以及内生单胞菌属在其他鱼类肠道中被证实有着消化蛋白质的功能, 推测玻璃鳗在洄游过程中摄取较多的蛋白质氨基酸作为其主要的能量来源。
王瑞旋, 冯娟, 2008. 军曹鱼肠道细菌及其产酶能力的研究[J]. 海洋环境科学, 27(4): 309-312. |
邓智明, 2018. 野生与养殖圆口铜鱼肠道微生物研究[D]. 南京: 南京农业大学.
|
田娟, 谢宁宁, 肖文富, 等, 2022. 灭活植物乳杆菌及其代谢物对草鱼生长性能和肠道健康的影响[J]. 水产学报, 46(10): 1980-1991. |
冯雪, 吴志新, 祝东梅, 等, 2008. 草鱼和银鲫肠道产消化酶细菌的研究[J]. 淡水渔业, 38(3): 51-57. |
邢孟欣, 2013. 养殖大菱鲆肠道微生物多样性及功能分析[D]. 青岛: 中国科学院研究生院(海洋研究所).
|
刘利平, 刘登攀, 蒲金成, 等, 2017. 日本鳗鲡仔鱼的开口饵料和行为特征[J]. 水产学报, 41(5): 703-710. |
刘增新, 2017. 牙鲆(Paralichthys olivaceus)仔稚幼鱼肠道菌群结构变化及定植规律研究[D]. 上海: 上海海洋大学.
|
孙云, 何明旺, 张盼盼, 等, 2020. 美洲鳗鲡致病性鲍曼不动杆菌的分离、鉴定及致病性分析[J]. 水产学报, 44(9): 1561-1571. |
孙莉, 2015. 鲍曼不动杆菌血流感染的流行特征和危险因素分析[D]. 杭州: 浙江大学.
|
李小彩, 2007. 一株红球菌的溶藻特性及应用研究[D]. 济南: 山东大学.
|
杨宇峰, 黄祥飞, 2000. 浮游动物生态学研究进展[J]. 湖泊科学, 12(1): 81-89. |
杨求华, 郭松林, 关瑞章, 等, 2012. 鳗鲡病原性维氏气单胞菌的分离与鉴定[J]. 生物技术通报, (7): 134-139. |
邱孜博, 汪荣, 张杨, 等, 2016. 红球菌及其生物降解作用研究进展[J]. 食品科学, 37(7): 254-258. |
张伟妮, 朱志华, 陈智伟, 等, 2010. 日本鳗鲡腐皮病病原菌的分离及鉴定[J]. 淡水渔业, 40(2): 41-46. |
张金花, 桂芳, 司婧, 等, 2017. 斑马鱼肠道微生物的研究进展[J]. 中国实验动物学报, 25(6): 637-642. |
张涵, 周涛, 王岩, 2013. 综合养殖池塘中三角帆蚌和鱼类肠道细菌的组成[J]. 水生生物学报, 37(5): 824-835. |
张婧怡, 肖俊, 梁军能, 等, 2020. 不同养殖环境下罗非鱼肠道微生物的比较分析[J]. 广西科学院学报, 36(2): 164-170. |
孟晓林, 李文均, 聂国兴, 2019. 鱼类肠道菌群影响因子研究进展[J]. 水产学报, 43(1): 143-155. |
ALLDREDGE A L, SILVER M W, 1988. Characteristics, dynamics and significance of marine snow[J]. Progress in Oceanography, 20(1): 41-82. |
HSU H Y, CHANG F C, WANG Y B, et al, 2018. Revealing the compositions of the intestinal microbiota of three Anguillid eel species using 16S rDNA sequencing[J]. Aquaculture Research, 49(7): 2404-2415. |
IIDA T, YAMAMOTO A, WAKABAYASHI H, 1984. Changes in intestinal flora of the juvenile eel, Anguilla japonica, after beginning to feed[J]. Fish Pathology, 19(3): 201-204. |
KOCA S B, YIGIT N Ö, DIDINEN B I, et al, 2015. Effects of enzyme-producing probiotic bacteria isolated from the gastrointestinal tract of trout on the growth performance, survival, and digestive enzyme activity of rainbow trout fry (Oncorhynchus mykiss)[J]. Israeli Journal of Aquaculture- Bamidgeh, 67: 1-9. |
MANABE R, AOYAMA J, WATANABE K, et al, 2011. First observations of the oceanic migration of Japanese eel, from pop-up archival transmitting tags[J]. Marine Ecology Progress Series, 437: 229-240. |
MILLER M J, CHIKARAISHI Y, OGAWA N O, et al, 2013. A low trophic position of Japanese eel larvae indicates feeding on marine snow[J]. Biology Letters, 9(1): 20120826. |
NEAVE M J, APPRILL A, FERRIER-PAGÈS C, et al, 2016. Diversity and function of prevalent symbiotic marine bacteria in the genus Endozoicomonas[J]. Applied Microbiology and Biotechnology, 100(19): 8315-8324. |
NURIEL-OHAYON M, NEUMAN H, KOREN O, 2016. Microbial changes during pregnancy, birth, and infancy[J]. Frontiers in Microbiology, 7: 1031. |
PÉREZ T, BALCÁZAR J L, RUIZ-ZARZUELA I, et al, 2010. Host-microbiota interactions within the fish intestinal ecosystem[J]. Mucosal Immunology, 3(4): 355-360. |
RAY A K, GHOSH K, RINGØ E, 2012. Enzyme-producing bacteria isolated from fish gut: a review[J]. Aquaculture Nutrition, 18(5): 465-492. |
SPANGGAARD B, HUBER I, NIELSEN J, et al, 2001. The probiotic potential against vibriosis of the indigenous microflora of rainbow trout[J]. Environmental Microbiology, 3(12): 755-765. |
TALWAR C, NAGAR S, LAL R, et al, 2018. Fish gut microbiome: current approaches and future perspectives[J]. Indian Journal of Microbiology, 58(4): 397-414. |
TANAKA H, KAGAWA H, OHTA H, et al, 2003. The first production of glass eel in captivity: fish reproductive physiology facilitates great progress in aquaculture[J]. Fish Physiology and Biochemistry, 28(1/2/3/4): 493-497. |
THANH N C, RAJESWARI V D, NARAYANAN M, et al, 2021. Evaluation of probiotic susceptibility of virulent Aeromonas sp. by a study on gut histology of Cyprinus carpio[J]. Process Biochemistry, 111: 154-159. |
TSUKAMOTO K, AOYAMA J, MILLER M J, 2009. Present status of the Japanese eel: resources and recent research [M] // CASSELMAN J M, CAIRNS D. Eels at the Edge: American Fisheries Society, Symposium. Bethesda: American Fisheries Society: 21-35.
|
WAHLBERG M, WESTERBERG H, AARESTRUP K, et al, 2014. Evidence of marine mammal predation of the European eel (Anguilla anguilla L.) on its marine migration[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 86: 32-38. |
WU SG, WANG G T, ANGERT E R, et al, 2012. Composition, diversity, and origin of the bacterial community in grass carp intestine[J]. PLoS One, 7(2): e30440. |
YANG Z B, WANG J, SHANG C C, et al, 2022. Spatial and temporal changes in bacterial community structure in adjacent waters of Dagu River estuary of Jiaozhou Bay (China) revealed by high-throughput sequencing[J]. Regional Studies in Marine Science, 52: 102302. |
ZHANG Y Y, MA L Y, 2021. Application of high-throughput sequencing technology in HIV drug resistance detection[J]. Biosafety and Health, 3(5): 276-280. |
ZHAO L L, YIN H C, LU T F, et al, 2018. Application of high-throughput sequencing for microbial diversity detection in feces of specific-pathogen-free ducks[J]. Poultry Science, 97(7): 2278-2286. |
 2023, Vol. 54
2023, Vol. 54


