中国海洋湖沼学会主办。
文章信息
- 汪慧娟, 何志巧, 石戈, 周素明, 张晓林, 申望. 2023.
- WANG Hui-Juan, HE Zhi-Qiao, SHI Ge, ZHOU Su-Ming, ZHANG Xiao-Lin, SHEN Wang. 2023.
- 大黄鱼(Larimichthys crocea) Tristetraprolin基因的克隆与功能分析
- MOLECULAR CHARACTERIZATION AND FUNCTIONAL ANALYSIS OF TRISTETRAPROLIN FROM LARGE YELLOW CROAKER LARIMICHTHYS CROCEA
- 海洋与湖沼, 54(6): 1786-1796
- Oceanologia et Limnologia Sinica, 54(6): 1786-1796.
- http://dx.doi.org/10.11693/hyhz20230500103
文章历史
-
收稿日期:2023-05-08
收修改稿日期:2023-07-29
2. 宁波大学海洋学院 浙江宁波 315211
2. School of Marine Sciences, Ningbo University, Ningbo 315211, China
炎症反应是病原感染、组织损伤等胁迫作用诱导的机体适应性反应。受控的适度炎症反应清除入侵病原或修复组织损伤, 而失控的高炎症反应则导致组织器官损伤, 诱发炎症性疾病甚至死亡(Medzhitov, 2008)。因此, 炎症因子表达在转录水平和转录后水平都受到严格调控(Yoshinaga et al, 2019)。RNA结合蛋白(RNA binding protein, RBP)介导的在转录后水平调节炎症因子mRNA加工、定位、降解和翻译, 是炎症因子表达调控的关键机制(Kafasla et al, 2014; Uchida et al, 2019)。
Tristetraprolin (TTP, 别名ZFP36、TIS11、GOS24、NUP475)是ZFP36家族RBP的原型蛋白, 广泛存在于真核生物中(Makita et al, 2021)。哺乳动物TTP都有3个保守功能结构域: N-端的核输出信号(nuclear export signal, NES)、中央串联的2个CCCH型锌指结构域(tandem zinc-finger domain, TZF)和C-端的NOT1-结合结构域(NOT1-binding domain, NOT1-BD) (Akira et al, 2021)。TZF结构域识别、结合靶mRNA 3′-非翻译区(untranslating region, UTR)的AU-富集元件(AU-rich element, ARE), N端和C端结构域募集CCR4-NOT1脱腺嘌呤复合体(CCR4-NOT1 deadenylase complex)、脱帽复合体(decapping complex, Dcp)、核酸外切体(exosomes)、5′~3′外切核糖核酸酶1 (5′~3′ exoribonuclease 1, XRN1)等促进靶mRNA降解, 调节靶基因表达(Akira et al, 2021)。TTP靶RNA组研究显示TTP的靶mRNA主要是炎症和免疫相关基因mRNA, 包括经典炎症因子TNF-α、IL-1β、IL-6的mRNA (Kratochvill et al, 2011; Mukherjee et al, 2014)。与之相对应, TTP参与调控多种炎症和免疫相关疾病的发生和发展(Zhang et al, 2021)。例如, 基因敲除TTP, TTP-KO小鼠表现出严重的炎症型疾病综合征, 包括恶液质、关节炎、皮肤炎、结膜炎、白血病等, TNF-α中和抗体处理可显著减轻症状(Taylor et al, 1996; Carballo et al, 2001)。反之, 敲除TTP mRNA 3′-UTR的AREs顺式元件过表达TTP, 下调LPS诱导的巨噬细胞和小鼠炎症因子表达, 减轻风湿性关节炎、内毒素血症、牛皮癣、多发性硬皮病等免疫相关疾病小鼠模型的临床症状(Patial et al, 2016b)。因此, TTP被认为是炎症性疾病的潜在治疗靶点(Mahmoud et al, 2019; Li et al, 2022)。感染性养殖病害一直是制约水产养殖业发展的瓶颈问题。例如, 在亚洲鱼类养殖业总经济损失中, 仅弧菌病所造成的经济损失就约占50% (Xu et al, 2022)。高炎症反应也是鱼类感染性病害的病理基础(Dai et al, 2023), 但迄今还未见鱼类TTP功能的研究报道, 特别是TTP对鱼类炎症因子表达的调节作用。
大黄鱼(Larimichthys crocea)是我国养殖规模最大的海水鱼(农业农村部渔业渔政管理局等, 2022)。随着养殖规模和养殖密度的增大, 大黄鱼感染性病害防治压力也越来越大(王凡等, 2019)。而抗生素等传统感染性病害防治方法由于环境安全和食品安全等问题, 已被禁用或严格限制使用(胡洋等, 2022), 开发抗生素替代物等大黄鱼感染性病害防治新方法成为保障产业健康发展的迫切需求。因此, 本研究以大黄鱼TTP (命名为LcTTP)为研究对象, 采用RACE (rapid amplification of cDNA end)技术克隆LcTTP基因全长cDNA序列并进行生物信息学分析, 研究LcTTP的结构特征和系统发生; 采用实时荧光定量技术(Real-time Quantitative PCR, RT-qPCR)检测LcTTP基因的组织表达模式; 并通过在大黄鱼头肾细胞系LYC-hK中过表达LcTTP, 研究LcTTP对LPS诱导的TNF-α mRNA表达水平的调节作用及其机制。研究结果可为进一步探讨LcTTP对大黄鱼抗感染炎症反应的调控作用以及评估其应用价值提供参考。
1 材料与方法 1.1 实验材料健康大黄鱼[平均体重(142.7±12.2) g]购买自浙江省舟山市半岛水产养殖有限公司, 在实验室循环海水养殖系统中使用过滤海水饲养一周适应环境后取样, 水温24~25 ℃。
为检测LcTTP基因的组织表达特异性, 5条健康大黄鱼浸泡于0.02% MS-222 (国药, 中国)麻醉后, 取肝、肠、胃、肌肉、心、脾、肾、鳃和脑等9个组织的样本放置于1.5 mL EP管中, 加入RNALaterTM动物组织RNA稳定保存液(碧云天, 上海), –80 ℃冻存备用。
1.2 全长cDNA克隆从大黄鱼NCBI基因组数据库(登录号: 12197)中检索到一个TTP基因同源物(基因登录号: 104928501)及其预测的mRNA序列(登录号: XM_010742797.3) (Ao et al, 2015)。以预测的大黄鱼TTP mRNA序列为参考, 使用NCBI在线引物设计工具Primer-Blast设计大黄鱼TTP mRNA 5′-RACE、3′-RACE和开放阅读框(opening reading frame, ORF) PCR引物(表 1) (Ye et al, 2012)。Trizol法提取总RNA, 按TaKaRa RNAiso Plus (TaKaRa, 日本)说明书操作。5′-RACE和3′-RACE使用TaKaRa SMARTer® RACE 5′/3′ Kit (TaKaRa, 日本), 操作按说明书进行。3′-RACE引物组合: 第一轮PCR引物LcTTPF1/3′-RACE UPM long primer, 第二轮巢式PCR引物LcTTPF2/3′-RACE UPM short primer; 5′-RACE引物组合: 第一轮PCR引物LcTTPR2/5′-RACE UPM long primer, 第二轮巢式PCR引物LcTTPR1/5′-RACE UPM short primer。ORF PCR扩增使用TaKaRa Ex Taq® DNA Polymerase (TaKaRa, 日本), 模板为5′/3′-RACE cDNA, 引物为ELcTTPF/ELcTTPR, 扩增程序: 95 ℃ 2 min; 95 ℃ 15 s, 56 ℃ 30 s, 72 ℃ 1 min, 35次循环。PCR产物经电泳检测、琼脂糖胶回收后连接simple pMD-19T vector (TaKaRa, 日本), 转化E.coli DH5α (TaKaRa, 日本), 挑阳性单克隆送上海生工测序, 拼接5′-RACE、3′-RACE和ORF序列获得LcTTP全长cDNA序列。
| 引物名称 | 序列(5′→3′) | 用途 |
| LcTTPF1 | TCCGAAATGCTTGACGAC | 3′-RACE |
| LcTTPR1 | AGGGAGTCACCAAACACTGG | 5′-RACE |
| LcTTPF2 | CTAACATGGGGTTCAGCCGT | 3′-RACE, RT-qPCR |
| LcTTPR2 | TGGCTCCAGGATGAGAGGAA | 5′-RACE, RT-qPCR |
| ELcTTPF | TGTACAAGTCCGAAATGCTTGACGA | 表达 |
| ELcTTPR | GCGGCCGCTTAGTCAGACAGGGACAG | 表达 |
| LcACTF | CCTTCACCACCACAGCCGAG | RT-qPCR |
| LcACTR | ATTCCGCAAGATTCCATACCGA | RT-qPCR |
| LcTNFαF | AACGCCTCACACCTCTCA | RT-qPCR |
| LcTNFαR | GTCCACAGTTTGTCTCCTCTATTC | RT-qPCR |
| LcIL1βF | ATCTGGCAAGGATCAGCTCA | RT-qPCR |
| LcIL1βR | ACCAGTTGTTGTAGGGGACG | RT-qPCR |
使用ExPASy在线工具包Compute pI/MW (https://web.expasy.org/compute_pi/)在线预测理论分子量和等电点; 使用NCBI在线工具包CD-Search识别保守结构域(Marchler-Bauer et al, 2017); 使用SignalP 6.0预测信号肽(Teufel et al, 2022); 使用Euk-mPLoc 2.0预测LcTTP的亚细胞定位(Chou et al, 2010); 使用NetNES 1.1工具包预测核输出信号(NES) (Fu et al, 2011); 使用BLASTP程序在NCBI非冗余(nr)数据库中搜索同源序列(Johnson et al, 2008); 多序列比对使用ClustalW1.83软件包(Larkin et al, 2007); 使用MEGA version 7软件包的邻接(Neighbor-Joining, NJ)法构建系统发育树(Kumar et al, 2016); 本研究中多序列比对和系统发育树分析使用的序列见表 2。
| GenBank登录号 | 拉丁名 | 中文名 | 与LcTTP的序列一致性/% |
| XP_010741099.1 | Larimichthys crocea | 大黄鱼 | 100.00 |
| XP_036937641.1 | Acanthopagrus latus | 黄鳍鲷 | 91.46 |
| XP_035535819.1 | Morone saxatilis | 条纹鲈鱼 | 91.28 |
| XP_045930373.1 | Micropterus dolomieu | 小口黑鲈 | 86.76 |
| XP_047449844.1 | Mugil cephalus | 鲻鱼 | 81.36 |
| NP_001106542.1 | Xenopus tropicalis | 热带爪蟾 | 45.27 |
| XP_040262428.1 | Bufo bufo | 中华大蟾蜍 | 42.76 |
| XP_032083892.1 | Thamnophis elegans | 吊带蛇 | 45.85 |
| XP_026546747.1 | Notechis scutatus | 虎蛇 | 44.68 |
| XP_038238166.1 | Dermochelys coriacea | 棱皮龟 | 43.77 |
| XP_037740475.1 | Chelonia mydas | 绿海龟 | 43.63 |
| XP_029769063.1 | Terrapene carolina triunguis | 三指箱龟 | 43.61 |
| XP_039181066.1 | Crotalus tigris | 虎斑响尾蛇 | 43.54 |
| XP_015676155.1 | Protobothrops mucrosquamatus | 原矛头蝮 | 42.48 |
| XP_026578640.1 | Pseudonaja textilis | 东部拟眼镜蛇 | 43.47 |
| XP_048358068.1 | Sphaerodactylus townsendi | 圣胡安角球趾壁虎 | 43.17 |
| NP_003398.3 | Homo sapiens | 人 | 40.69 |
| NP_004917.2 | Homo sapiens | 人 | 38.08 |
| NP_008818.3 | Homo sapiens | 人 | 36.27 |
| NP_035886.1 | Mus musculus | 小鼠 | 39.58 |
| NP_031590.1 | Mus musculus | 小鼠 | 38.08 |
| NP_001001806.1 | Mus musculus | 小鼠 | 36.09 |
| NP_001009549.1 | Mus musculus | 小鼠 | 32.75 |
Trizol法提取总RNA, 按TaKaRa RNAiso Plus (TaKaRa, 日本)说明书操作。取1 μg总RNA按TaKaRa PrimeScriptTM RT reagent Kit with gDNA Eraser试剂盒(TaKaRa, 日本)使用说明合成cDNA第一链。20 μL RT-qPCR反应体系按BeyoFastTM SYBR Green qPCR Mix (碧云天, 中国)试剂盒说明书配制, 即SYBR Green qPCR Mix 10 μL、上下游引物混合物2.0 μL、cDNA 2.0 μL、超纯水6 μL, 引物序列见表 1; 反应程序: 95 ℃ 2 min; 95 ℃ 15 s, 60 ℃ 30 s, 40次循环, 反应在ABI QuantStudio 1实时检测系统(Applied Biosystems, 美国)上运行; 扩增完成后, 采用熔解曲线分析验证扩增特异性。以β-actin为内参基因, 采用2–ΔΔCt法分析目的基因的相对表达水平; 在mRNA稳定性分析中, 以放线菌素D处理0 h TNF-α mRNA表达水平为参照, 采用2–ΔCt法分析其他时间点TNF-α mRNA的相对表达水平(Livak et al, 2001)。
1.5 构建pEGFP-N1-LcTTP重组载体(1.2)中测序验证正确LcTTP ORF单克隆扩大培养后提取pMD-19T-LcTTP重组质粒, pMD-19T-LcTTP重组质粒和pEGFP-N1表达载体质粒(Clontech, 美国)分别用限制性内切酶NotI和BsrGI (TaKaRa, 日本)双酶切, 酶切片段胶回收纯化后使用T4 DNA连接酶(TaKaRa, 日本)连接, 连接产物转化E.coli DH5α挑单克隆送上海生工测序, 测序验证正确的pEGFP-N1-LcTTP单克隆扩大培养后使用Endo-free Plasmid Mini Kit Ⅱ (Omega, 美国)提取无内毒质粒。重组表达载体pEGFP-N1-LcTTP表达产物为GFP-LcTTP融合蛋白。
1.6 细胞培养和转染大黄鱼头肾细胞系LYC-hK按之前报道培养(Zhou et al, 2023)。简而言之, 使用含15%胎牛血清(FBS; 四季青, 中国)、100 U/mL青霉素/链霉素(P/S)的DMEM/HAM’S F-12完全培养基(Hyclone, 美国)置于26 ℃、5% CO2的二氧化碳培养箱中培养。
细胞转染按GP-transfect-Mate转染试剂(吉玛, 中国)说明书操作。简而言之: 6孔板按2×105细胞/孔接种LYC-hK细胞, 24 h后3 μg质粒DNA与6 μL GP-transfect-Mate试剂混合于400 μL无FBS的DME/F-12培养基静置20 min后转染LYC-hK细胞, 转染6 h换完全培养基继续培养48 h, 荧光显微镜(Leica, 德国)观察转染效率后进行后续实验。
1.7 数据分析所有数据均采用GraphPad Prism 6.0软件(GraphPad software Inc., 美国)进行分析, 并以均数±均数标准误差(SEM)表示。组间统计学差异采用非配对双尾学生t检验, P值小于0.05为有统计学意义。
2 结果 2.1 LcTTP全长cDNA序列分析将5′-RACE、3′-RACE和ORF扩增序列拼接得到的LcTTP全长cDNA序列共1 508 bp, 其中ORF 1 248 bp, 5′-UTR 77 bp, 3′-UTR 183 bp, polyA结构上游63 bp处有一加尾信号AATAA (图 1)。
 |
| 图 1 LcTTP cDNA核酸序列和推导的氨基酸序列 Fig. 1 The nucleotide sequence and deduced amino acid sequence of LcTTP 注: 下划线示核输出信号(NES); 阴影区示串联重复锌指结构域(TZF); 星号示CCCH型锌指结构域的保守氨基酸残基位点; 粗体示起始密码子和终止密码子; 方框表示加尾信号 |
推导的LcTTP蛋白序列包含415个氨基酸残基, 理论分子量(MW) 44 897.78 Da, 预测等电点(pI) 8.37。SignalP 6.0分析LcTTP未在N端检测到信号肽, Euk-mPLoc 2.0亚细胞定位分析预测LcTTP定位在细胞核, NetNES 1.1分析显示在LcTTP的N端有一NES序列(T10KNFMNLALDDGLL23; 图 1)。CD-Search分析显示ZFP36家族特征结构域TZF也保守存在于LcTTP中央区域。多序列比对显示LcTTP氨基酸序列与黄鳍鲷(XP_036937641.1)、条纹鲈鱼(XP_035535819.1)、人(NP_003398.3)和小鼠(NP_035886.1) TTP氨基酸序列的同源性分别为91.46%、91.28%、40.69%和39.58%; 且哺乳动物TTP保守的重要功能结构域: 中央TZF结构域和C端NOT1-BD结构域也在LcTTP中保守存在(图 2)。
 |
| 图 2 脊椎动物代表物种TTP多序列比对 Fig. 2 The multiple alignment of the predicted LcTTP with other vertebrate species 注: 黑色阴影示相同氨基酸位点; 灰色示相似氨基酸残基位点; 上划线以人TTP为参考, 示保守结构域; NES: 核输出序列; TZF: 串联锌指结构域; CTD: C端结构域; 星号示CCCH型锌指结构域保守氨基酸位点 |
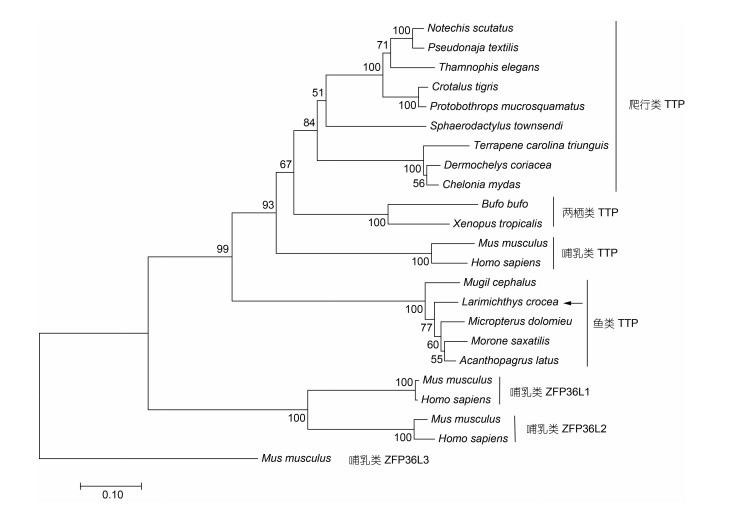
系统发育分析显示: 脊椎动物TTP聚为一支(置信度99%), 与哺乳动物ZFP36家族的其他成员ZFP36L1、ZFP36L2和ZFP36L3分开。在脊椎动物中, TTP又聚类为哺乳动物、爬行动物、两栖动物和鱼类4个进化支, LcTTP与其他硬骨鱼TTP聚为一支(图 3)。以上结果提示本研究克隆得到的LcTTP是大黄鱼ZFP36家族成员TTP。
基因的组织表达特异性与其生理功能密切相关。为检测LcTTP基因的组织表达特异性, 本研究采集5条健康大黄鱼的脑、胃、鳃、肝、脾、肾、头肾、肌肉、心脏等9个组织, RT-qPCR检测不同组织中LcTTP mRNA的表达水平。结果显示9个组织均检测到LcTTP mRNA表达, 但不同组织表达量差异较大(图 4)。肌肉组织LcTTP mRNA表达量最高(53.63±27.43), 其次为胃(16.99±14.38)和鳃(10.18±4.27), 而脑(1.10±0.23)、肝(2.58±1.02)、肾(2.98±1.25)、脾(3.01±1.03)、头肾(4.42±1.26)、心脏(4.89±1.93)等组织LcTTP mRNA表达量均较低。
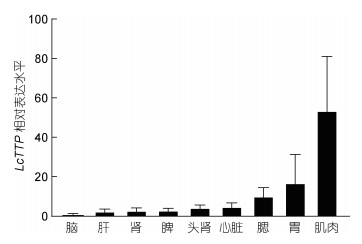 |
| 图 4 LcTTP基因的组织表达谱(n=5) Fig. 4 Tissue-specific expression patterns of LcTTP (n=5) |
LPS是诱导炎症因子表达的经典病原相关分子模式(pathogen associated molecular pattern, PAMP)分子。哺乳动物研究显示LPS通过诱导巨噬细胞、淋巴细胞、嗜中性粒细胞等免疫细胞TTP表达, 促进TNF-α、IL-1β等炎症因子mRNA降解, 下调LPS诱导的炎症因子表达(Mahtani et al, 2001; Fairhurst et al, 2003; Tchen et al, 2004)。因此, 本研究首先调查LPS是否诱导LYC-hK细胞炎症因子和LcTTP基因表达。研究结果显示(图 5): LPS (1 μg/mL)处理LYC-hK细胞3 h显著上调TNF-α mRNA表达量(1.48倍; P < 0.01), 6 h时显著下调(0.77倍; P < 0.05); 而IL-1β mRNA表达量在LPS处理2 h时显著上调(1.98倍; P < 0.01), 6 h时显著下调(0.69倍; P < 0.01), 之后逐渐上升, 24 h时又显著上调(1.64倍; P < 0.05); LcTTP mRNA表达量在LPS处理3 h时显著上调(1.57倍; P < 0.001), 其他时间点均出现小幅下调(1 h 0.80倍、2 h 0.83倍、6 h 0.75倍、12 h 0.78倍、24 h 0.81倍)。以上结果提示LYC-hK细胞中LcTTP可能参与调控LPS诱导的炎症因子表达。
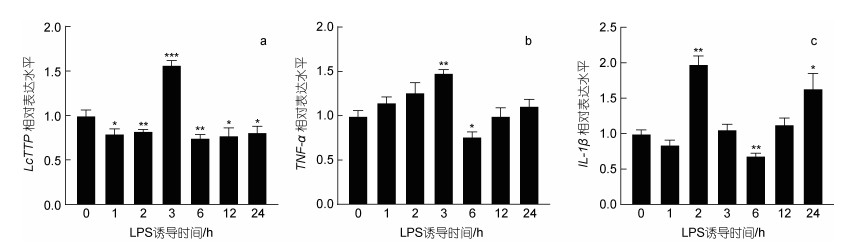 |
| 图 5 LPS诱导LYC-hK细胞LcTTP和炎症因子表达 Fig. 5 The expression of LcTTP and inflammatory factor induced by LPS in LYC-hK cells 注: a. LcTTP; b. TNF-α; c. IL-1β |
炎症因子TNF-α mRNA是哺乳动物中最早鉴定研究的TTP靶mRNA, TTP通过结合TNF-α mRNA 3′-UTR的ARE顺式元件促进TNF-α mRNA降解, 抑制TNF-α表达(Taylor et al, 1996; Carballo et al, 1997)。因此, 本研究在大黄鱼LYC-hK细胞中过表达LcTTP, 调查LcTTP是否调节LPS诱导的TNF-α表达及其作用机制。研究结果显示(图 6): 与空载体pEGFP-N1转染对照组相比, pEGFP-N1-LcTTP转染的过表达组在LPS (1 μg/mL)处理0.5 h (3.48 vs 4.80; P < 0.01)、1 h (3.44 vs 11.27; P < 0.001)、1.5 h (3.50 vs 4.52; P < 0.05)、3 h (2.24 vs 3.12; P < 0.01)、6 h (1.34 vs 2.36; P < 0.01)均显著上调TNF-α mRNA表达量, 峰值在LPS处理1 h时。表明过表达LcTTP在LPS诱导早期大幅上调TNF-α mRNA表达量, LPS处理1 h后TNF-α mRNA表达量快速下调到接近对照组水平, 提示大黄鱼TNF-α mRNA可能是LcTTP的靶mRNA。
 |
| 图 6 过表达LcTTP调节LYC-hK细胞LPS诱导的TNF-α基因表达 Fig. 6 The overexpression of LcTTP regulates LPS-induced TNF-α expression in LYC-hK cells |
促进靶mRNA降解是哺乳动物TTP调节炎症因子表达的主要机制之一(Bataclan et al, 2021)。本研究在LPS (1 μg/mL)处理LYC-hK细胞1 h后, 以放线菌D (5 mg/mL)抑制转录, RT-qPCR分析转录抑制后pEGFP-N1转染对照组和pEGFP-N1-LcTTP转染过表达组TNF-α mRNA的降解速率, 调查LcTTP对靶mRNA稳定性的影响。研究结果如图 7所示, 过表达LcTTP显著上调TNF-α mRNA的降解速率, pEGFP-N1转染对照组和pEGFP-N1-LcTTP转染过表达组TNF-α mRNA的半衰期(t1/2)分别为115 min和40 min, 表明LcTTP可通过促进TNF-α mRNA降解调节TNF-α表达。
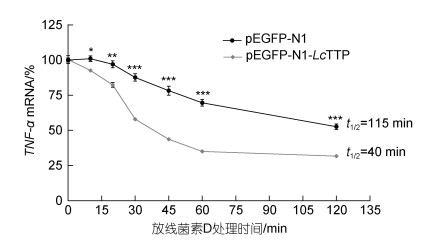 |
| 图 7 LcTTP促进TNF-α mRNA降解 Fig. 7 LcTTP promotes TNF-α mRNA decay |
哺乳动物研究显示TTP可通过促进细胞因子mRNA降解调节免疫反应(Makita et al, 2021), 是炎症和免疫相关疾病的潜在治疗靶点(Patial et al, 2016a)。显然, 借鉴哺乳动物研究成果开展鱼类TTP的功能研究, 可为鱼类感染性养殖病害的防治提供新线索。本研究采用RACE技术克隆了大黄鱼LcTTP的全长cDNA序列, 推导的氨基酸序列分析显示哺乳动物TTP重要功能结构域在LcTTP中也保守存在, 系统发生分析显示LcTTP与其他脊椎动物TTP聚为一支且与哺乳动物ZFP36家族的其他成员ZFP36L1、ZFP36L2和ZFP36L3等分离; 组织表达特异性分析显示LcTTP基因在检测的9个大黄鱼组织中广泛表达, 但不同组织表达水平差异较大; 在大黄鱼头肾细胞系LYC-hK中, LPS处理诱导炎症因子表达时也调节LcTTP基因的表达, 提示LYC-hK细胞中LcTTP可能参与调控LPS诱导的炎症因子表达; LYC-hK细胞过表达LcTTP证实LcTTP可通过促进TNF-α mRNA降解调节LPS诱导的TNF-α表达。这些研究结果提示脊椎动物中TTP的结构和功能可能非常保守, 大黄鱼LcTTP也可通过促进炎症因子mRNA降解调节炎症因子表达, 可能是大黄鱼炎症反应的关键调控蛋白, 在大黄鱼感染性病害防治策略开发中有潜在应用价值。
ZFP36蛋白家族是以中央TZF结构域为结构特征的一类RNA结合蛋白, 广泛存在于从单细胞真核生物、无脊椎动物到哺乳动物的整个真核生物界(Blackshear et al, 2014)。哺乳动物ZFP36蛋白家族通常有3个成员: TTP、ZFP36L1和ZFP36L2, 小鼠ZFP36蛋白家族还有一个在胎盘和胚胎外组织特异表达的ZFP36L3 (Blackshear et al, 2005)。鱼类和两栖类包含哺乳动物ZFP36家族的直系同源物和一个独特的C3H-4蛋白, 该蛋白有一个典型的TZF结构域和2个退化的锌指结构域(De et al, 1999)。TTP是ZFP36家族首先报道的原型蛋白, 通常有3个保守结构域: N-端的NES信号序列、中央TZF结构域和C-端的NOT1-BD (Ciais et al, 2013)。多序列比对显示哺乳动物TTP的3个功能结构域在LcTTP中也保守存在, 但LocNES工具包分析显示LcTTP的NOT1-BD结构域可能还是潜在的NES信号序列。ZFP36家族成员ZFP36L1和ZFP36L2缺失N端的NES信号序列, NES定位在C端替代TTP的NOT1-BD结构域(Ciais et al, 2013)。而在系统树中, LcTTP与其他硬骨鱼TTP聚在一起, 合并其他脊椎动物TTP聚为一支, 与哺乳动物的ZFP36L1、ZFP36L2、ZFP36L3等分离。这些结果表明本研究克隆的LcTTP属于典型的TTP蛋白, 也提示LcTTP可能与哺乳动物TTP有相似的功能。
基因组织表达水平与其生理功能密切相关, 调查目的基因的组织表达特异性可为其功能研究提供线索(Lu et al, 2004)。TTP mRNA在成年小鼠各种组织中表达量均较低, 肾和肺中TTP mRNA表达量相对较高, 脂肪、心、肝、脾相对较低, 而在骨骼肌、睾丸和脑中只有痕量表达(Lai et al, 1990)。在蛋白水平, 肝、卵巢和睾丸中TTP水平高, 而脑、心、肠、脾、胸腺、肾、骨骼肌TTP水平较低, 肺中几乎检测不到TTP蛋白(Lu et al, 2004)。本研究检测的9个大黄鱼组织器官中均检测到LcTTP mRNA表达, 这与小鼠中观察到TTP mRNA广泛表达现象类似, 提示LcTTP可能也如哺乳动物TTP广泛参与机体免疫反应的调节。但大黄鱼LcTTP基因的组织表达特异性与小鼠TTP基因存在差异, LcTTP mRNA在肌肉中的表达量最高, 其余依次为胃、腮、心、头肾、脾、肾、肝和脑, 提示TTP基因的组织表达特异性可能存在种间差异, 而种间差异的生理、病理意义还需在后续功能研究进一步探讨。
炎症是宿主清除入侵病原的必需生理反应, 但高炎症反应和炎症因子过量分泌也导致组织损伤甚至死亡, 因此, 免疫细胞炎症因子的表达在转录水平和转录后水平都受到严格调控(Akira et al, 2021)。TTP是在转录后水平通过促进炎症因子mRNA降解下调炎症因子表达的抑炎蛋白(Mukherjee et al, 2014)。哺乳动物巨噬细胞、树突状细胞、嗜中性粒细胞、T细胞、B细胞等免疫细胞中均表达TTP (Makita et al, 2021; Zhang et al, 2021)。哺乳动物巨噬细胞是TNF-α等炎症因子的主要分泌细胞, 研究显示LPS等炎症诱导物处理巨噬细胞早期TTP与炎症因子表达水平同步上调, 处理后期TTP通过促进炎症因子mRNA降解抑制炎症因子表达(Ostareck et al, 2019), 所以巨噬细胞是研究TTP调控炎症因子表达的首选细胞类型。本研究曾尝试转染大黄鱼原代单核/巨噬细胞过表达LcTTP研究LcTTP对TNF-α mRNA表达的调控效应及其机制, 但因转染效率低(< 10%)放弃。而大黄鱼头肾细胞系LYC-hK转染效率较高(50%~80%), 且LPS处理LYC-hK细胞早期, 炎症因子TNF-α (3 h)和IL-1β (2 h)mRNA表达量显著上调, 并在诱导6 h时回复到基础水平之下; LcTTP mRNA表达量小幅下调(1 h和2 h)后显著上调(3 h)。这与LPS诱导哺乳动物炎症因子和TTP mRNA表达的模式相似(Ostareck et al, 2019), 提示LYC-hK细胞中LcTTP可能参与调控LPS诱导的炎症因子表达。因此, 本研究选择大黄鱼LYC-hK细胞系研究LcTTP对炎症因子表达的调节效应及其作用机制。
小鼠巨噬细胞TTP的主要靶RNA是炎症因子(TNF-α、IL-1α、IL-1β、IL-6等)以及其他免疫相关蛋白的mRNA (Mukherjee et al, 2014; Sedlyarov et al, 2016)。TTP基因敲除巨噬细胞TNF-α (Carballo et al, 1997, 1998)、GM-CSF (Carballo et al, 2000) mRNA的稳定性增强, mRNA及其编码蛋白的表达量均上调; 反之, 过表达TTP则显著下调巨噬细胞LPS诱导的TNF-α、IL-1β、Cxcl2等mRNA的表达量(Patial et al, 2016b)。但在本研究中, LYC-hK细胞过表达LcTTP显著上调LPS诱导早期(1 h) TNF-α mRNA表达量, 之后迅速下降到对照组水平(2 h)。Lai等(1999)报道HEK293细胞低水平过表达TTP下调TNF-α mRNA表达量, 但高水平过表达TTP反而上调TNF-α mRNA表达量(Lai et al, 1999); 以重组nLuc-ARE为靶mRNA, 过表达野生型TTP和组成型激活TTP-AA也观察到相同现象, 即低水平过表达TTP下调nLuc-ARE mRNA表达量, 而高水平过表达TTP反而上调nLuc-ARE mRNA表达量(Mahmoud et al, 2019)。这些研究提示TTP作为核糖核酸外切酶复合体(exoribonuclease complex)支架蛋白, 高水平表达后可能由于复合体其他组分不足, 组装完全的活性复合体反而减少, 导致核糖核酸外切酶总活性降低, 上调靶mRNA表达量; 随着LPS诱导时间延长, 核糖核酸外切酶复合体其他结构组分表达水平上调, 组装完全活性复合体增多, 靶mRNA降解加速而急剧下降(Burack et al, 2000; Mahmoud et al, 2019)。同时在本研究中也发现: 放线菌素D抑制转录后, 过表达LcTTP促进TNF-α mRNA降解, 下调TNF-α mRNA的半衰期, 表明LcTTP也如哺乳动物TTP, 可通过促进靶RNA降解下调靶基因表达(Carballo et al, 1998; Deleault et al, 2008)。因此, LcTTP可能是大黄鱼炎症反应的关键调控蛋白。
4 结论本研究采用RACE技术克隆鉴定了LcTTP基因的全长cDNA序列, 序列分析显示哺乳动物TTP重要的功能结构域在LcTTP中保守; 组织表达特异性分析显示LcTTP基因在大黄鱼机体中广泛表达, 但不同组织表达水平差异较大; LYC-hK细胞过表达LcTTP发现LcTTP通过促进TNF-α mRNA降解调节LPS诱导的TNF-α表达。这些结果提示脊椎动物中TTP的结构和功能可能非常保守, 大黄鱼LcTTP可通过促进炎症因子mRNA降解调节炎症因子表达, 可能是大黄鱼炎症反应的关键调控蛋白, 在大黄鱼感染性病害防治策略开发中有潜在应用价值。
王凡, 廖碧钗, 孙敏秋, 等, 2019. 福建大黄鱼产业发展形势分析[J]. 中国水产, (3): 45-49. DOI:10.3969/j.issn.2095-1833.2019.03.006 |
农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会, 2022. 2022中国渔业统计年鉴[J]. 北京: 中国农业出版社: 22.
|
胡洋, 张旭, 王欢, 等, 2022. 药用植物资源在水产动物疾病控制中的研究进展[J]. 中国生物工程杂志, 42(11): 43-58. DOI:10.13523/j.cb.2209023 |
AKIRA S, MAEDA K, 2021. Control of RNA stability in immunity[J]. Annual Review of Immunology, 39: 481-509. DOI:10.1146/annurev-immunol-101819-075147 |
AO J Q, MU Y N, XIANG L X, et al, 2015. Genome sequencing of the perciform fish Larimichthys crocea provides insights into molecular and genetic mechanisms of stress adaptation[J]. PLoS Genetics, 11(4): e1005118. DOI:10.1371/journal.pgen.1005118 |
BATACLAN M, LEONI C, MONTICELLI S, 2021. RNA‐binding proteins and RNA methylation in myeloid cells[J]. Immunological Reviews, 304(1): 51-61. DOI:10.1111/imr.13025 |
BLACKSHEAR P J, PERERA L, 2014. Phylogenetic distribution and evolution of the linked RNA-binding and NOT1-binding domains in the tristetraprolin family of tandem CCCH zinc finger proteins[J]. Journal of Interferon & Cytokine Research, 34(4): 297-306. |
BLACKSHEAR P J, PHILLIPS R S, GHOSH S, et al, 2005. Zfp36l3, a rodent X chromosome gene encoding a placenta-specific member of the Tristetraprolin family of CCCH tandem zinc finger proteins[J]. Biology of Reproduction, 73(2): 297-307. DOI:10.1095/biolreprod.105.040527 |
BURACK W R, SHAW A S, 2000. Signal transduction: hanging on a scaffold[J]. Current Opinion in Cell Biology, 12(2): 211-216. DOI:10.1016/S0955-0674(99)00078-2 |
CARBALLO E, BLACKSHEAR P J, 2001. Roles of tumor necrosis factor-α receptor subtypes in the pathogenesis of the tristetraprolin-deficiency syndrome[J]. Blood, 98(8): 2389-2395. DOI:10.1182/blood.V98.8.2389 |
CARBALLO E, GILKESON G S, BLACKSHEAR P J, 1997. Bone marrow transplantation reproduces the tristetraprolin-deficiency syndrome in recombination activating gene-2 (-/-) mice. Evidence that monocyte/macrophage progenitors may be responsible for TNFalpha overproduction[J]. The Journal of Clinical Investigation, 100(5): 986-995. DOI:10.1172/JCI119649 |
CARBALLO E, LAI W S, BLACKSHEAR P J, 1998. Feedback inhibition of macrophage tumor necrosis factor-α production by tristetraprolin[J]. Science, 281(5379): 1001-1005. DOI:10.1126/science.281.5379.1001 |
CARBALLO E, LAI W S, BLACKSHEAR P J, 2000. Evidence that tristetraprolin is a physiological regulator of granulocyte-macrophage colony-stimulating factor messenger RNA deadenylation and stability[J]. Blood, 95(6): 1891-1899. DOI:10.1182/blood.V95.6.1891 |
CHOU K C, SHEN H B, 2010. A new method for predicting the subcellular localization of eukaryotic proteins with both single and multiple sites: Euk-mPLoc 2.0[J]. PLoS One, 5(4): e9931. DOI:10.1371/journal.pone.0009931 |
CIAIS D, CHERRADI N, FEIGE J J, 2013. Multiple functions of tristetraprolin/TIS11 RNA-binding proteins in the regulation of mRNA biogenesis and degradation[J]. Cellular and Molecular Life Sciences, 70(12): 2031-2044. DOI:10.1007/s00018-012-1150-y |
DAI Y F, LV Z M, YOU M X, et al, 2023. PPARα alleviates inflammation via inhibiting NF-κB/Rel pathway in Vibrio splendidus challenged Apostichopus japonicus[J]. Fish & Shellfish Immunology, 135: 108701. |
DE J, LAI W S, THORN J M, et al, 1999. Identification of four CCCH zinc finger proteins in Xenopus, including a novel vertebrate protein with four zinc fingers and severely restricted expression[J]. Gene, 228(1/2): 133-145. |
DELEAULT K M, SKINNER S J, BROOKS S A, 2008. Tristetraprolin regulates TNF TNF-α mRNA stability via a proteasome dependent mechanism involving the combined action of the ERK and p38 pathways[J]. Molecular Immunology, 45(1): 13-24. DOI:10.1016/j.molimm.2007.05.017 |
FAIRHURST A M, CONNOLLY J E, HINTZ K A, et al, 2003. Regulation and localization of endogenous human tristetraprolin[J]. Arthritis Research & Therapy, 5(4): 1-12. |
FU S C, IMAI K, HORTON P, 2011. Prediction of leucine-rich nuclear export signal containing proteins with NESsential[J]. Nucleic Acids Research, 39(16): e111. DOI:10.1093/nar/gkr493 |
JOHNSON M, ZARETSKAYA I, RAYTSELIS Y, et al, 2008. NCBI BLAST: a better web interface[J]. Nucleic Acids Research, 36: W5-W9. DOI:10.1093/nar/gkn201 |
KAFASLA P, SKLIRIS A, KONTOYIANNIS D L, 2014. Post-transcriptional coordination of immunological responses by RNA-binding proteins[J]. Nature Immunology, 15(6): 492-502. DOI:10.1038/ni.2884 |
KRATOCHVILL F, MACHACEK C, VOGL C, et al, 2011. Tristetraprolin‐driven regulatory circuit controls quality and timing of mRNA decay in inflammation[J]. Molecular Systems Biology, 7(1): 560. DOI:10.1038/msb.2011.93 |
KUMAR S, STECHER G, TAMURA K, 2016. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
LAI W S, CARBALLO E, STRUM J R, et al, 1999. Evidence that tristetraprolin binds to AU-rich elements and promotes the deadenylation and destabilization of tumor necrosis factor alpha mRNA[J]. Molecular and Cellular Biology, 19(6): 4311-4323. DOI:10.1128/MCB.19.6.4311 |
LAI W S, STUMPO D J, BLACKSHEAR P J, 1990. Rapid insulin-stimulated accumulation of an mRNA encoding a proline-rich protein[J]. Journal of Biological Chemistry, 265(27): 16556-16563. DOI:10.1016/S0021-9258(17)46259-4 |
LARKIN M A, BLACKSHIELDS G, BROWN N P, et al, 2007. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
LI S M, LIU S Y, CHEN R A, et al, 2022. Activation of the MKK3-p38-MK2-ZFP36 Axis by coronavirus infection restricts the upregulation of AU-rich element-containing transcripts in proinflammatory responses[J]. Journal of Virology, 96(5): e0208621. DOI:10.1128/jvi.02086-21 |
LIVAK K J, SCHMITTGEN T D, 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCt method[J]. Methods, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
LU J Y, SCHNEIDER R J, 2004. Tissue distribution of AU-rich mRNA-binding proteins involved in regulation of mRNA decay[J]. Journal of Biological Chemistry, 279(13): 12974-12979. DOI:10.1074/jbc.M310433200 |
MAHMOUD L, MOGHRABI W, KHABAR K S A, et al, 2019. Bi-phased regulation of the post-transcriptional inflammatory response by Tristetraprolin levels[J]. RNA Biology, 16(3): 309-319. DOI:10.1080/15476286.2019.1572437 |
MAHTANI K R, BROOK M, DEAN J L E, et al, 2001. Mitogen-activated protein kinase p38 controls the expression and posttranslational modification of tristetraprolin, a regulator of tumor necrosis factor alpha mRNA stability[J]. Molecular and Cellular Biology, 21(19): 6461-6469. DOI:10.1128/MCB.21.9.6461-6469.2001 |
MAKITA S, TAKATORI H, NAKAJIMA H, 2021. Post-transcriptional regulation of immune responses and inflammatory diseases by RNA-binding ZFP36 family proteins[J]. Frontiers in Immunology, 12: 711633. DOI:10.3389/fimmu.2021.711633 |
MARCHLER-BAUER A, BO Y, HAN L Y, et al, 2017. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures[J]. Nucleic Acids Research, 45(D1): D200-D203. DOI:10.1093/nar/gkw1129 |
MEDZHITOV R, 2008. Origin and physiological roles of inflammation[J]. Nature, 454(7203): 428-435. DOI:10.1038/nature07201 |
MUKHERJEE N, JACOBS N C, HAFNER M, et al, 2014. Global target mRNA specification and regulation by the RNA-binding protein ZFP36[J]. Genome Biology, 15(1): R12. DOI:10.1186/gb-2014-15-1-r12 |
OSTARECK D H, OSTARECK-LEDERER A, 2019. RNA-binding proteins in the control of LPS-induced macrophage response[J]. Frontiers in Genetics, 10: 31. DOI:10.3389/fgene.2019.00031 |
PATIAL S, BLACKSHEAR P J, 2016a. Tristetraprolin as a therapeutic target in inflammatory disease[J]. Trends in Pharmacological Sciences, 37(10): 811-821. DOI:10.1016/j.tips.2016.07.002 |
PATIAL S, CURTIS Ⅱ A D, LAI W S, et al, 2016b. Enhanced stability of tristetraprolin mRNA protects mice against immune-mediated inflammatory pathologies[J]. Proceedings of the National Academy of Sciences of the United States of America, 113(7): 1865-1870. |
SEDLYAROV V, FALLMANN J, EBNER F, et al, 2016. Tristetraprolin binding site atlas in the macrophage transcriptome reveals a switch for inflammation resolution[J]. Molecular Systems Biology, 12(5): 868. DOI:10.15252/msb.20156628 |
TAYLOR G A, CARBALLO E, LEE D M, et al, 1996. A pathogenetic role for TNFα in the syndrome of cachexia, arthritis, and autoimmunity resulting from tristetraprolin (TTP) deficiency[J]. Immunity, 4(5): 445-454. DOI:10.1016/S1074-7613(00)80411-2 |
TCHEN C R, BROOK M, SAKLATVALA J, et al, 2004. The stability of tristetraprolin mRNA is regulated by mitogen-activated protein kinase p38 and by tristetraprolin itself[J]. Journal of Biological Chemistry, 279(31): 32393-32400. DOI:10.1074/jbc.M402059200 |
TEUFEL F, ALMAGRO ARMENTEROS J J, JOHANSEN A R, et al, 2022. SignalP 6.0 predicts all five types of signal peptides using protein language models[J]. Nature Biotechnology, 40(7): 1023-1025. DOI:10.1038/s41587-021-01156-3 |
UCHIDA Y, CHIBA T, KURIMOTO R, et al, 2019. Post-transcriptional regulation of inflammation by RNA-binding proteins via cis-elements of mRNAs[J]. The Journal of Biochemistry, 166(5): 375-382. DOI:10.1093/jb/mvz067 |
XU K P, WANG Y S, YANG W X H, et al, 2022. Strategies for prevention and control of vibriosis in Asian fish culture[J]. Vaccines, 11(1): 98. DOI:10.3390/vaccines11010098 |
YE J, COULOURIS G, ZARETSKAYA I, et al, 2012. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction[J]. BMC Bioinformatics, 13: 134. DOI:10.1186/1471-2105-13-134 |
YOSHINAGA M, TAKEUCHI O, 2019. Post‐transcriptional control of immune responses and its potential application[J]. Clinical & Translational Immunology, 8(6): e1063. |
ZHANG Y W, ZHOU J, WEI Z Y, et al, 2021. TTP-mediated regulation of mRNA stability in immune cells contributes to adaptive immunity, immune tolerance and clinical applications[J]. RNA Biology, 18(12): 2150-2156. DOI:10.1080/15476286.2021.1917185 |
ZHOU S M, WANG Y, SHU F L, et al, 2023. Functional insights of a two-component system sensor kinase GacS in a fish pathogen, Pseudomonas plecoglossicida[J]. Aquaculture, 562: 738866. DOI:10.1016/j.aquaculture.2022.738866 |
 2023, Vol. 54
2023, Vol. 54