
中国海洋湖沼学会主办。
文章信息
- 李若晖, 张琳琳. 2024.
- LI Ruo-Hui, ZHANG Lin-Lin. 2024.
- 基于RNA-seq数据分析长牡蛎(Crassostrea gigas) 天然免疫的可变剪接事件
- ANALYSIS OF ALTERNATIVE SPLICING EVENTS FOR INNATE IMMUNITY OF CRASSOSTREA GIGAS INDUCED BASED ON RNA-SEQ
- 海洋与湖沼, 55(1): 182-192
- Oceanologia et Limnologia Sinica, 55(1): 182-192.
- http://dx.doi.org/10.11693/hyhz20230700148
文章历史
-
收稿日期:2023-07-14
收修改稿日期:2023-08-13
2. 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室 山东青岛 266237;
3. 中国科学院大学 北京 100049
2. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
免疫系统是生物体为了抵御病原体入侵, 在长期进化过程中形成的一套复杂的防御系统, 它可以维持机体的内稳态(蒲新明, 2008)。免疫系统通过一系列免疫应答反应参与免疫调节(王金星等, 2004)。免疫系统可以分为天然免疫系统(Innate immune system)和获得性免疫系统(Adaptive immune system)两种类型, 两种类型对应着不同的作用机制(李颖, 2011)。普遍认为无脊椎动物只存在非特异性的天然免疫, 缺乏获得性免疫(Litman et al, 2005)。无脊椎动物应对病原微生物的入侵主要依赖天然免疫系统。为应对各种各样的病原微生物入侵, 基因组需要通过不同的调控产生特定转录组和蛋白质组, 从而完成对病原体入侵做出有效和快速的天然免疫反应。
在漫长的自然进化过程中无脊椎动物进化出一套功效强大的先天防御体系, 通过细胞代谢的重编程和免疫系统的激活使它们能够有效地应对病原体入侵。许多细胞过程的正常进行在很大程度上取决于RNA代谢, 特别是RNA的成熟和降解。在真核生物中, 利用不同的剪接位点, 将相同的前体mRNA转化成多个信使RNA (mRNA)的过程, 被称作可变剪接(Alternative splicing, AS) (Kahles et al, 2016)。可变剪接通过使基因产生结构和功能不同的mRNA和蛋白质, 从而增强转录组可塑性和蛋白质功能多样性(Maniatis et al, 2002; Nilsen et al, 2010)。可变剪接可以从数量相对较少的基因中产生大量的mRNA和蛋白质异构体。可变剪接还可以改变转录本及其编码蛋白的结构, 从而调节蛋白质的功能, 而可变剪接产生的异构体甚至可能具有完全相反的功能(Stamm et al, 2005)。与哺乳动物中大量关于可变剪接的研究相比, 这一机制在无脊椎动物天然免疫中的作用和调控却知之甚少。
近年来, 高通量技术和生物信息学工具的快速发展, 特别是二代代测序技术, 如Illumina、PacBio和Oxford Nanopore直接RNA测序, 对研究基因表达变化和分析全基因组可变剪接事件做出巨大贡献。目前, 利用RNA-Seq技术分析了包括人(Homo sapiens)、线虫(Caenorhabditis elegans)、果蝇(Drosophila melanogaster)、拟南芥(Arabidopsis thaliana)等多个物种基因组的AS事件(Pan et al, 2008; Graveley et al, 2011; Ramani et al, 2011; Marquez et al, 2012)。利用生物信息学工具进一步深入挖掘转录组学数据来识别可变剪接事件, 将有助于阐明无脊椎动物在生物应激条件下的基因调控机制。
长牡蛎(Crassostrea gigas)既是世界上重要的经济贝类, 也是一种常见的模式无脊椎动物, 已被广泛用于研究(Zhang et al, 2012, 2015)。已有研究表明, AS在长牡蛎响应高温、高盐和空气暴露等非生物应激中发挥重要作用(Huang et al, 2016), 而AS在长牡蛎生物应激中的功能研究仍处于空白状态。因此, 本研究基于Illumina高通量测序的转录组数据(Zhang et al, 2015), 分析了长牡蛎在不同病原体和混合弧菌不同感染时间诱导下的可变剪接差异, 发现混合弧菌诱导可变剪接事件的增加, 分析了在不同病原体和混合弧菌诱导中差异表达的可变剪接基因的功能, 从而探究可变剪接对长牡蛎天然免疫多样性和特异性的调控作用。本研究为长牡蛎等无脊椎动物天然免疫多样性和特异性的深入研究提供了理论依据, 为后续长牡蛎病害防御提供了数据支持。
1 材料与方法 1.1 数据收集与处理收集来自NCBI的13个长牡蛎测序数据, 此数据由中国科学院海洋研究所2013年上传发布, 登录号为PRJNA194079和PRJNA194084。具体来说, 测序样品来自: (1) 5个混合弧菌感染(感染后0、6、12、24和48 h收集的感染组的鳃); (2) 8个不同致病菌[4个革兰氏阳性菌: 河口弧菌(Vibrio aestuarianus)、鳗弧菌(Vibrio anguillarum)、溶藻弧菌(Vibrio alginolyticus)和LPS, 1个革兰氏阴性菌: 藤黄微球菌(Micrococcus luteus), PBS和对照组的鳃]。
为了保证信息分析质量, 对原始数据利用FastQC质控, 使用Trimmomatic去除原始数据中带接头的、低质量的以及带N碱基的序列, 得到Clean reads数据。
1.2 可变剪接事件的鉴定将Clean reads与长牡蛎基因组利用软件STAR进行比对, 比对结果运用Cufflinks软件(Trapnell et al, 2012)进行组装, 并使用Cuffcompare与长牡蛎参考注释进行比较生成tmap文件(Trapnell et al, 2010)。使用ASprofile中的“extract-as”程序(Florea et al, 2013), 利用gtf、tmap文件在多个样本中鉴定12种可变剪接事件, 包括单外显子跳跃(skipped exon, SKIP)、单外显子跳跃(模糊边界) (approximate SKIP, XSKIP)、多外显子跳跃(multi-exon SKIP, MSKIP)、多外显子跳跃(模糊边界) (approximate MSKIP, XMSKIP)、单内含子滞留(intron retention, IR)、单内含子滞留(模糊边界) (approximate IR, XIR)、多内含子滞留(multi-IR, MIR)、多内含子滞留(模糊边界)(approximate MIR, XMIR)、可变5′或3′端剪切(alternative exonends, AE)、可变5′或3′端剪切(模糊边界) (approximate AE, XAE)、第1个外显子可变剪切(alternative 5′ first exon, TSS)、最后1个外显子可变剪切(alternative 3′ last exon, TTS)。
1.3 先天免疫相关基因分析及GO功能富集分析使用cuffdiff程序对混合弧菌不同感染时间和不同病原体样本进行不同表达基因分析(Trapnell et al, 2013)。然后使用参数|log2(fold_change)| > 1和P≤0.05和FDR≤0.05对生成的差异表达基因数据进行过滤。
利用DAVID数据库, 对显著差异表达的可变剪接基因ID进行GO分类, 获取GO编号, 并利用cluster Profiler对其进行功能富集, 以P≤0.05作为显著富集标准。
1.4 预测的长牡蛎AS基因亚型的表达分析根据Cuffnorm利用默认参数对每个样本中鉴定出的异构体进行定量。通过Cuffdiff计算混合弧菌感染条件下FPKM的转录表达量, 然后对所有样本进行归一化处理。
2 结果与分析 2.1 长牡蛎感染混合弧菌后不同时间可变剪接事件的鉴定为了研究混合弧菌感染下不同时间长牡蛎可变剪接的作用, 从NCBI数据库下载了5个长牡蛎Illumina高通量测序平台的转录组测序数据。这些数据集来自5个混合弧菌不同感染时间的RNA-seq读数, 包括0、6、12、24和48 h, 数据集的总碱基读数为15.27 Gb。
根据5个NCBI RNA-seq数据集, 计算了5个不同感染时间的AS事件的数量(表 1), 其中, 感染48 h后有最丰富的AS事件(80 566), 而感染6 h后的数量最少(74 573) (图 1)。在经混合弧菌感染后, 长牡蛎发生的可变剪接事件类型仍以TSS、TTS、AE和SKIP为主, 结果表明, 可变剪接在各时间点的数目存在差异, 随着时间的增长, 可变剪接事件也随着增多。
| 组别 | 0 h | 6 h | 12 h | 24 h | 48 h |
| AE | 204 | 152 | 165 | 140 | 138 |
| XAE | 32 | 22 | 21 | 12 | 20 |
| IR | 16 | 23 | 16 | 39 | 26 |
| XIR | 12 | 11 | 11 | 17 | 10 |
| MIR | 1 | 1 | 1 | 2 | 1 |
| XMIR | 2 | 1 | 3 | 0 | 0 |
| SKIP | 184 | 174 | 172 | 146 | 154 |
| XSKIP | 24 | 38 | 21 | 23 | 17 |
| MSKIP | 9 | 7 | 10 | 12 | 12 |
| XMSKIP | 0 | 0 | 3 | 2 | 1 |
| TSS | 38 641 | 36 931 | 37 469 | 39 675 | 40 022 |
| TTS | 38 941 | 37 213 | 37 651 | 39 863 | 40 165 |
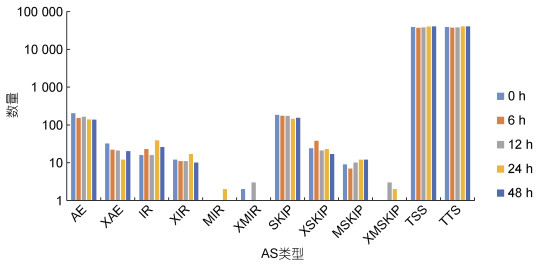 |
| 图 1 长牡蛎在混合弧菌胁迫下不同时间点的可变剪接事件 Fig. 1 Alternative splicing events of C. gigas under Vibrio challenge at different time points |
为了研究不同病原体长牡蛎可变剪接的作用, 从NCBI数据库下载了7个长牡蛎Illumina高通量测序平台的转录组测序数据。这些数据集来自不同病原体入侵的长牡蛎的RNA-seq读数, 包括LPS、河口弧菌(Vibrio aestuarianus)、鳗弧菌(Vibrio anguillarum)、溶藻弧菌(Vibrio alginolyticus)、塔氏弧菌(Vibrio tubiashii)、藤黄微球菌(Micrococcus luteus)。
根据7个NCBI RNA-seq数据集, 计算了7个不同病原体诱导的AS事件的数量(表 2)。结果表明, 不同病原体诱导后长牡蛎的可变剪接的总数目存在差异, 但长牡蛎发生的可变剪接事件类型仍为TSS、TTS、AE和SKIP四种主要类型(图 2)。
| 组别 | PBS | LPS | 河口弧菌 | 鳗弧菌 | 溶藻弧菌 | 塔式弧菌 | 藤黄微球菌 |
| AE | 152 | 156 | 122 | 150 | 162 | 139 | 175 |
| XAE | 28 | 20 | 28 | 30 | 32 | 24 | 26 |
| IR | 8 | 21 | 19 | 13 | 17 | 15 | 19 |
| XIR | 8 | 12 | 8 | 10 | 10 | 11 | 14 |
| MIR | 3 | 2 | 2 | 3 | 1 | 2 | 2 |
| XMIR | 1 | 2 | 0 | 1 | 0 | 0 | 0 |
| SKIP | 156 | 138 | 175 | 169 | 175 | 164 | 172 |
| XSKIP | 31 | 11 | 28 | 24 | 23 | 18 | 25 |
| MSKIP | 16 | 7 | 7 | 10 | 12 | 16 | 13 |
| XMSKIP | 3 | 0 | 1 | 4 | 2 | 1 | 3 |
| TSS | 36 477 | 35 159 | 35 767 | 36 792 | 35 327 | 34 901 | 36 580 |
| TTS | 36 716 | 35 396 | 36 002 | 37 050 | 35 688 | 35 144 | 36 874 |
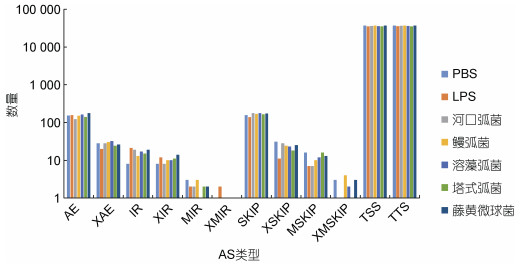 |
| 图 2 长牡蛎不同病原体入侵下的可变剪接事件 Fig. 2 Alternative splicing events of C. gigas challenged with different types of bacteria |
对感染混合弧菌的长牡蛎不同时间的可变剪接基因进行差异表达分析, 分别得到528、330、302和313个显著差异表达基因。对不同病原体诱导时发生可变剪接的基因进行差异表达分析, 分别得到528、330、302和313个显著差异表达基因。在这些显著差异基因中, TSS和TTS是主要的可变剪接方式, 这表明它们在响应弧菌的基因表达中可能起着重要的作用。
发生可变剪接事件的差异表达基因有多个基因为天然免疫相关基因家族成员, 混合弧菌不同感染时间发生可变剪接的显著差异表达基因中分别有38、28、30和31个天然免疫基因(表 3), 不同病原体入侵发生可变剪接的显著差异表达基因中分别有25、26、25、23、29和29个天然免疫基因(表 4), 这些基因家族包括RLR、TLR、SRCR、C1q等。但是混合弧菌不同感染时间发生可变剪接的天然免疫相关基因分别有450、466、482和501个, 占长牡蛎天然免疫相关基因总数的三分之一左右, 说明在病原体感染时, 机体中三分之一的天然免疫基因通过可变剪接发挥作用。
| 基因名称 | 6 h | 12 h | 24 h | 48 h |
| a2M | 0 | 1 | 0 | 0 |
| C1q | 6 | 5 | 8 | 6 |
| C-lectin | 8 | 7 | 5 | 5 |
| FREP | 3 | 2 | 2 | 2 |
| IKB | 1 | 1 | 1 | 1 |
| MBL | 1 | 1 | 1 | 1 |
| Galectin | 0 | 0 | 1 | 0 |
| MITA_id | 2 | 0 | 0 | 1 |
| NLR | 3 | 3 | 3 | 3 |
| SRCR | 3 | 2 | 2 | 4 |
| TNFR | 0 | 1 | 1 | 0 |
| CD36 | 1 | 0 | 0 | 0 |
| NF-kb | 1 | 0 | 0 | 0 |
| SARM | 1 | 0 | 0 | 0 |
| TNF | 1 | 0 | 0 | 0 |
| LRR-IG | 0 | 0 | 0 | 1 |
| SOD | 0 | 0 | 0 | 0 |
| TRAF | 1 | 0 | 0 | 0 |
| others | 6 | 5 | 6 | 7 |
| 总计 | 38 | 28 | 30 | 31 |
| 基因名称 | LPS | V. aes | V. ang | V. alg | V. tub | M. lut |
| C1q | 4 | 5 | 6 | 3 | 4 | 6 |
| C-lectin | 4 | 5 | 4 | 4 | 5 | 5 |
| FREP | 3 | 1 | 1 | 1 | 1 | 2 |
| IKB | 1 | 1 | 2 | 1 | 1 | 1 |
| MBL | 1 | 1 | 1 | 1 | 1 | 1 |
| Galectin | 0 | 0 | 0 | 0 | 1 | 0 |
| orphan | 0 | 0 | 0 | 2 | 0 | 1 |
| MITA_id | 0 | 1 | 0 | 0 | 1 | 1 |
| NLR | 3 | 3 | 3 | 3 | 3 | 3 |
| SRCR | 3 | 2 | 3 | 2 | 4 | 3 |
| SARM-like | 0 | 1 | 0 | 0 | 1 | 1 |
| SOD | 0 | 0 | 0 | 0 | 1 | 0 |
| TRAF | 0 | 0 | 0 | 0 | 1 | 0 |
| Others | 6 | 6 | 5 | 6 | 5 | 5 |
| 总计 | 25 | 26 | 25 | 23 | 29 | 29 |
为了解可变剪接对长牡蛎先天免疫的调控, 对弧菌诱导的5个不同时间基因组中发生AS事件的显著差异表达基因进行了GO富集, 以P≤0.05为显著富集标准, 发现得到143、179、153和164个条目(图 3)。
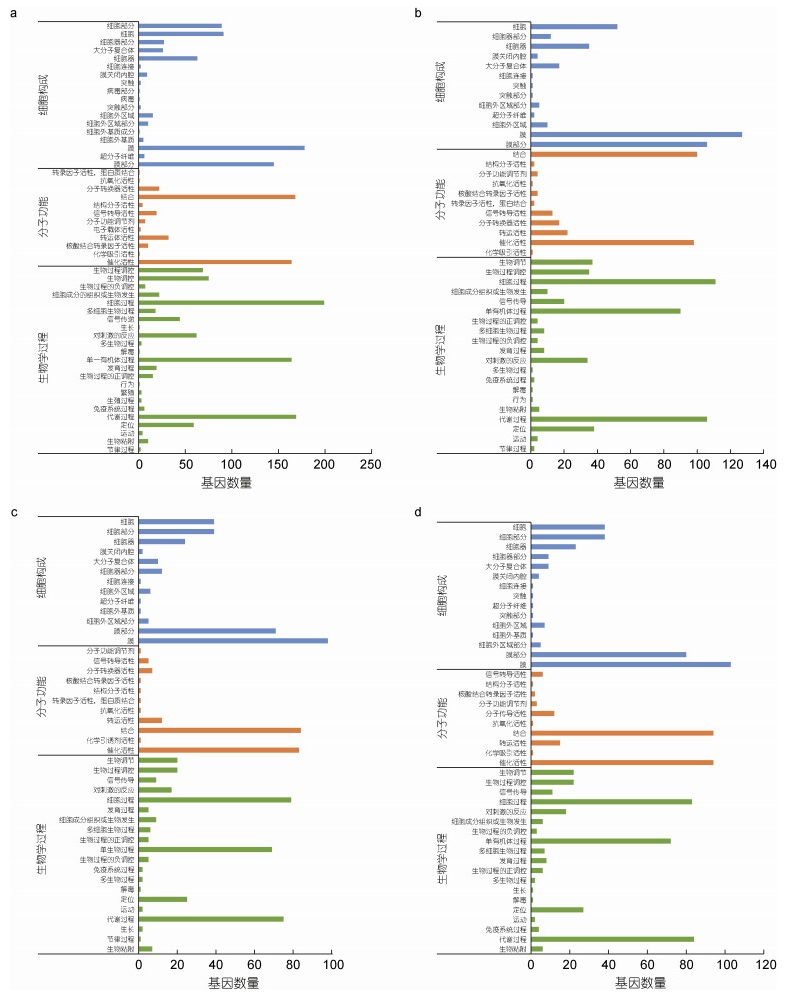 |
| 图 3 弧菌诱导下不同时间差异表达可变剪接基因GO富集 Fig. 3 Statistics of GO annotations for alternative splicing genes with variable expression in response to Vibrio challenge at different points 注: a. 6 h差异可变剪接基因GO富集结果; b. 12 h差异可变剪接基因GO富集结果; c. 24 h差异可变剪接基因GO富集结果; d. 48 h差异可变剪接基因GO富集结果 |
在0 h和6 h的差异基因中, 生物学过程、细胞构成和分子功能中显著富集的GO条目分别有36、35、72个。在生物学过程中, 发生可变剪接的差异基因主要集中于有机物的分解代谢过程(GO: 1901575)等代谢相关过程; 在分子功能中, 较多可变剪接基因被富集于催化活性(GO: 0003824)相关GO项中; 在细胞构成中, 发生可变剪接的基因主要集中于膜的固有成分(GO: 0031224)与膜相关条目。
在0 h和12 h的差异基因中, 在生物学过程、细胞构成和分子功能中显著富集的GO条目分别有101、20、58个。在生物学过程中, 发生可变剪接的差异基因主要集中于α-氨基酸代谢过程(GO: 1901605)等代谢相关过程; 在分子功能中, 较多可变剪接基因被富集于辅助因子结合(GO: 0048037)相关GO项中; 在细胞构成中, 发生可变剪接的基因主要集中于膜的固有成分(GO: 0031224)与膜相关条目。
在0 h和24 h的差异基因中, 在生物学过程、细胞构成和分子功能中显著富集的GO条目分别有101、10、72个。在生物学过程中, 发生可变剪接的差异基因主要集中于有机物的分解代谢过程(GO: 1901575)等代谢相关过程; 在分子功能中, 较多可变剪接基因被富集于催化活性(GO: 0003824)相关GO项中; 在细胞构成中, 发生可变剪接的基因主要集中于膜的固有成分(GO: 0031224)与膜相关条目。
在0 h和48 h的差异基因中, 在生物学过程、细胞构成和分子功能中显著富集的GO条目分别有112、13、39个。在生物学过程中, 发生可变剪接的差异基因主要集中于单一有机体代谢过程(GO: 0044710)等代谢相关过程; 在分子功能中, 较多可变剪接基因被富集于催化活性(GO: 0003824)相关GO项中; 在细胞构成中, 发生可变剪接的基因主要集中于膜的固有成分(GO: 0031224)与膜相关条目。
由此可见, 这些差异基因主要包括以下主要生物过程: (1) 细胞和代谢过程; (2) 生物过程的调控; (3) 细胞分泌物的产生; (4) 生物黏附过程; (5) 对刺激反应响应先天性免疫系统的启动; (6) 先天性免疫信号转导; (7) 细胞的生长等。其中与免疫相关的比例占据较小, 细胞和代谢过程以及生物调节过程的比例占据较高。这说明在长牡蛎生物应激过程中, 细胞、代谢过程、催化活性和生物调节等途径可能参与机体自身的免疫防御, 与机体的免疫途径共同响应病原体感染。
2.5 差异可变剪接基因的KEGG富集分析对混合弧菌入侵不同时间发生可变剪接的差异表达基因进行KEEG富集分析(图 4), 结果表明富集到与免疫相关通路, 在12 h富集到细胞凋亡(ko04210)、CD分子(ko04090)、信号分子与相互作用(ko09133)、细胞生长和死亡(ko09143)等; 在24 h富集到NOD-like受体信号通路(ko04621)和免疫系统(ko09151)等。
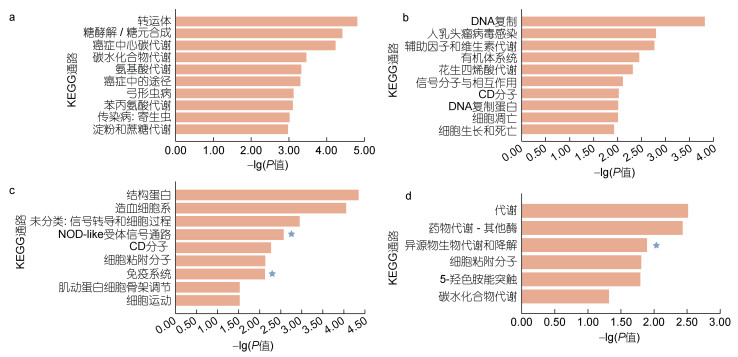 |
| 图 4 弧菌诱导下不同时间差异表达可变剪接基因KEGG富集 Fig. 4 Statistics of KEGG annotations for alternative splicing genes with variable expression in response to Vibrio challenge at different points 注: a. 6 h差异可变剪接基因KEGG富集结果; b. 12 h差异可变剪接基因KEGG富集结果; c. 24 h差异可变剪接基因KEGG富集结果; d. 48 h差异可变剪接基因KEGG富集结果; 星号表示免疫相关通路 |
对不同病原体发生可变剪接的差异表达基因进行KEEG富集分析(图 5), 结果表明富集到与免疫相关通路, 在LPS富集到PI3K-AKT通路(ko04151)等; 在V. aes富集到NF-kappaB细胞信号传导通路(ko04064); 在V. ang富集到Toll与Imd信号通路(ko04624)、NOD样受体信号通路(ko04621)、免疫系统(ko09151)和NF-kappaB细胞信号传导通路(ko04064); 在V. alg富集到NOD样受体信号通路(ko04621); 在V. tub富集到免疫系统(ko09151); 在M. lut富集到NOD样受体信号通路(ko04621)、Toll与Imd信号通路(ko04624)。
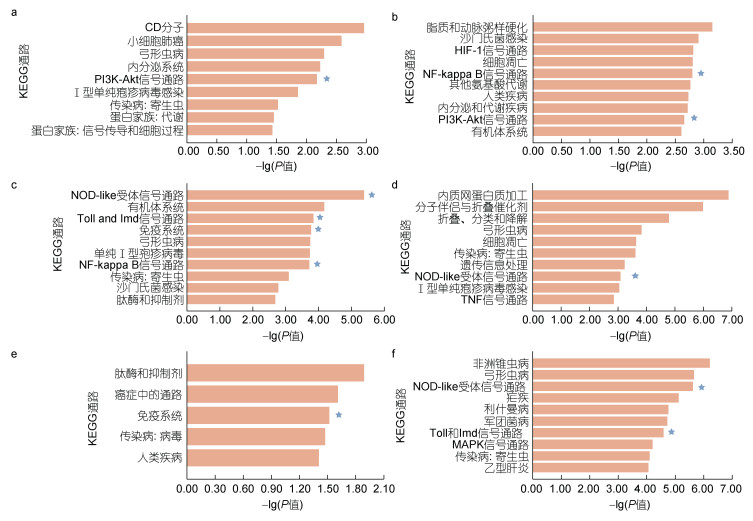 |
| 图 5 不同病原体诱导下差异表达可变剪接基因KEGG富集 Fig. 5 Statistics of KEGG annotations for alternative splicing genes with variable expression in response to various Vibrio challenges 注: a. LPS差异可变剪接基因KEGG富集结果; b. V. aes差异可变剪接基因KEGG富集结果; c. V. ang差异可变剪接基因KEGG富集结果; d. V. alg差异可变剪接基因KEGG富集结果; e. V. tub差异可变剪接基因KEGG富集结果; f: M. lut差异可变剪接基因KEGG富集结果; 星号表示免疫相关通路 |
综合差异表达筛选、GO富集分析和KEGG富集分析发现, 在混合弧菌感染6 h时富集到代谢相关通路(图 4a), 推测在感染初期, 机体对能量的需求增加, 迫使体内养分重新分配, 积极提供应激所需的能量物质和氨基酸; 在混合弧菌感染12 h时富集到细胞凋亡相关通路(图 4b), 推测在感染中期, 由于混合弧菌对长牡蛎鳃组织的急性感染, 导致鳃细胞出现凋亡, 以至于在感染6 h、12 h发生可变剪接事件总个数出现减少; 在混合弧菌感染24 h时富集到免疫相关通路、48 h时富集到异源物质代谢降解相关通路(图 4c, 4d), 推测随着感染时间推移, 鳃组织的天然免疫系统发挥作用, 消除了混合弧菌的急性感染, 并且及时清除机体内由于病原体入侵产生的异物, 逐步恢复鳃组织的免疫稳态。
2.6 混合弧菌胁迫下先天免疫基因的可变剪接事件异构体变化天然免疫在进化上是一个相对保守的系统, 主要通过PRRs识别PAMPs进行信号传递。RLRs是重要的RNA传感器分子。对两个RLR基因的分析显示, Cg02656和Cg26493基因都拥有六个可变剪接异构体。图 6a和图 7a显示了这两个基因的异构体经历了多种可变剪接事件, 表明可变剪接的调节可能非常复杂和多样, 例如, 异构体Cg02656E出现了TSS、IR和TTS。另外, 为了探索特定可变剪接异构体与致病菌胁迫之间的关系, 计算了不同感染时间可变剪接转录本的表达量(图 6b和图 7b)。RNA-seq数据的FPKM值表明, Cg02656和Cg26493基因的可变剪接异构体表达量在不同的感染时间存在差异表达。Cg02656和Cg26493基因的表达量均在48 h后达到最高值, 其中Cg02656基因在48 h时的表达量约是感染0 h时的2倍。
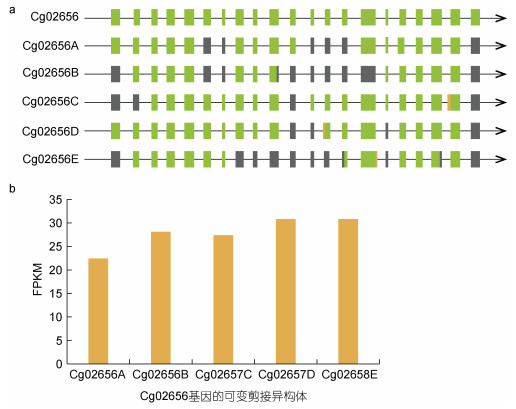 |
| 图 6 Cg02656基因的AS异构体(a)及其FPKM表达量(b) Fig. 6 The AS isoforms of the Cg02656 gene (a) and their FPKM expressions (b) 注: Cg02656A、B、C、D、E分别为0、6、12、24、48 h所对应的Cg02656基因异构体; FPKM: 每千个碱基的转录每百万映射读取的fragments (Fragments Per Kilobase of exon model per Million mapped fragments), 是基因相对表达量的衡量指标 |
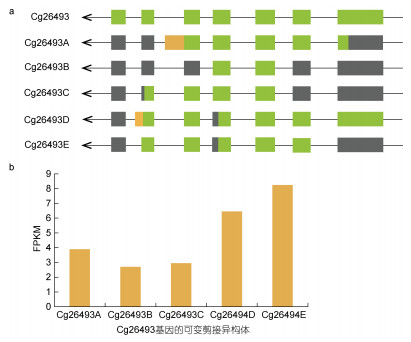 |
| 图 7 Cg26493基因的AS异构体(a)及其FPKM表达量(b) Fig. 7 The AS isoforms (a) of the Cg26493 gene and their FPKM expressions (b) 注: Cg26493A、B、C、D、E分别为0、6、12、24、48 h所对应的Cg26493基因异构体 |
基于这些发现, 我们认为长牡蛎可能具有复杂的可变剪接调节系统, 并且可变剪接可能是长牡蛎产生天然免疫多样性和特异性的重要途径。例如, 这些异构体可能作为Cg02656和Cg26493编码的完整蛋白的调节因子, 直接参与免疫反应或其他重要的生物学功能。不同感染时间中异构体表达的差异也表明可变剪接调控在不同免疫过程之间存在差异。
3 讨论机体的免疫是在长期进化过程中形成的, 免疫机制复杂性与各种生物大分子的种类与数量有关, 而在有限长度的遗传信息基础上产生更多的转录本, 无疑能在转录水平上大幅度提高免疫反应的效率, 进而实现对机体最大程度的保护。目前已有大量研究指出许多动物的天然免疫与可变剪接高度相关(Tan et al, 2019; Guo et al, 2022), 这种转录后水平上的调控与免疫应答中多种关键因子的生物合成过程密不可分。此外, 这种调控方式也会造成物种之间代谢通路的特异性。
本研究对感染混合弧菌后长牡蛎的鳃组织中的可变剪接事件进行检测。随着时间的增长, 可变剪接事件的数量也均呈现增加的趋势, 表明混合弧菌的感染会诱导长牡蛎可变剪接事件的产生。关于胁迫反应引起可变剪接事件数量增加的现象在长牡蛎中已有相关报道。Huang等(2016)的研究结果表明在长牡蛎在高温、高盐、空气暴露胁迫下, 可变剪接事件的数量都有所增加。由此可见, 可变剪接事件的产生可能是长牡蛎响应各种胁迫反应的一种重要调控机制。在本研究中, 12种类型的可变剪接事件均被鉴定出, 其中可变剪接事件数量最多的是TSS和TTS, 其次是AE和SKIP。
本研究识别了感染混合弧菌前后长牡蛎鳃组织中的发生可变剪接的差异表达基因, 并对这些基因进行了富集分析。其中GO的富集分析结果主要包含天然免疫应答、代谢过程。值得注意的是, 长牡蛎感染混合弧菌后12 h和24 h发生可变剪接的差异表达基因富集到了免疫系统通路上, 但在6 h的样本中并未发现此类富集, 这表明长牡蛎感染后可变剪接事件不会迅速响应。天然免疫应答是非特异性的、机体固有的、反应迅速的、广泛发生的免疫方式, 表明在感染混合弧菌后长牡蛎主要通过天然免疫应答清除入侵病原菌, 对机体进行保护。有机物代谢过程等GO词条被认为参与了机体能量代谢过程, 表明免疫应答过程会进行大量能量消耗与物质代谢。在KEGG富集分析结果中, 发生可变剪接的差异表达基因被显著富集在细胞凋亡、NOD-like受体信号通路和免疫系统等信号通路上。细胞凋亡是为了维持内环境的稳定, 由基因控制的非被动的细胞死亡, 并不只在机体受到胁迫时才发挥作用, 也是正常细胞代谢的生理现象之一(Elmore, 2007)。细胞凋亡也是免疫系统的一种作用方式。Nagata等(2017)的研究表明, 许多凋亡调节因子均在免疫系统中发挥着重要作用。NOD-like受体(NLR)信号通路是生物体的一种重要免疫防御机制。作为天然免疫系统的关键组成部分之一, NLR信号通路能够识别并响应各种外来病原体、细胞损伤和自身核酸等危险信号, 从而启动免疫防御反应, 维护机体的健康稳定(Van Gorp et al, 2014)。NLR信号通路主要包括模式识别受体(PRR)、炎症小体、caspase-1及相关炎性因子等多个组成部分, 这些部分在免疫防御反应中相互协作, 发挥着重要的作用。
本研究识别了不同病原体入侵长牡蛎鳃组织中的发生可变剪接的差异表达基因, 并对这些基因进行了富集分析。在KEGG富集分析结果中, 发生可变剪接的差异表达基因被显著富集在PI3K-AKT通路、NOD-like受体信号通路、NF-kappaB细胞信号传导通路和Toll与Imd信号通路等信号通路上。核转录因子κB (NF-κB)是转录因子蛋白家族中的一员, 在免疫系统中发挥非常重要的调节作用(Hayden et al, 2006)。NF-κB可以调控细胞因子、生长因子以及效应蛋白酶的表达, 从而参与到免疫应答中。在哺乳动物中, Toll样受体(TLRs)直接识别病原微生物的病原体相关分子模式, 通过NF-κB触发信号反应, 启动先天免疫和适应性免疫反应(Takeda et al, 2003)。Toll信号途径最先被发现于果蝇, 它主要与机体对真菌和革兰氏阳性细菌的免疫反应有关(Kim et al, 2003; Kaneko et al, 2005)。果蝇Toll蛋白能与多种PMAPs结合, 从而活化胞内Tube、Pele等多种胞内信号分子, 进而调控免疫相关基因的表达。Imd信号转导途径是一条与机体响应革兰氏阴性细菌的免疫反应相关的信号转导途径, 其在果蝇体内可调控220种基因的表达, 其中就有革兰氏抗阴性细菌的多肽(Romeo et al, 2008)。最近的研究发现, Imd信号通路还可以激活JNK (c-Jun N-terminal kinase), 实现细胞骨架蛋白的诱导表达。这表明Imd信号转导途径不仅在天然免疫应答中, 而且也在组织修复中起着关键作用。
4 结论本研究基于RNA-seq测序数据, 鉴定了长牡蛎病原体诱导过程中发生的可变剪接事件类型及数目, 筛选并分析在不同病原体和混合弧菌诱导中差异表达的可变剪接基因的功能, 从而探究可变剪接对长牡蛎天然免疫多样性和特异性的调控作用。本研究为长牡蛎等无脊椎动物天然免疫多样性和特异性的深入研究提供了理论依据, 为后续长牡蛎病害防御提供了数据支持。
致谢 本文获得中国科学院海洋研究所海洋大数据中心的支持, 谨致谢忱。
王金星, 赵小凡, 2004. 无脊椎动物先天免疫模式识别受体研究进展[J]. 生物化学与生物物理进展, 31(2): 112-117. DOI:10.3321/j.issn:1000-3282.2004.02.003 |
李颖, 2011. 细胞抗病毒天然免疫信号转导的调控机制[D]. 武汉: 武汉大学.
|
蒲新明, 2008. 职业环境与机体免疫[J]. 职业与健康, 24(10): 985-987. DOI:10.3969/j.issn.1004-1257.2008.10.046 |
ELMORE S, 2007. Apoptosis: A review of programmed cell death[J]. Toxicologic Pathology, 35(4): 495-516. DOI:10.1080/01926230701320337 |
FLOREA L, SONG L, SALZBERG S L, 2013. Thousands of exon skipping events differentiate among splicing patterns in sixteen human tissues[J]. F1000 Research, 2: 188. DOI:10.12688/f1000research.2-188.v1 |
GRAVELEY B R, BROOKS A N, CARLSON J W, et al, 2011. The developmental transcriptome of Drosophila melanogaster[J]. Nature, 471(7339): 473-479. DOI:10.1038/nature09715 |
GUO Y N, SUN Z D, ZHANG Y T, et al, 2022. Molecular identification and function characterization of four alternative splice variants of trim25 in Japanese flounder (Paralichthys olivaceus)[J]. Fish & Shellfish Immunology, 120: 142-154. |
HAYDEN M S, WEST A P, GHOSH S, 2006. NF-κB and the immune response[J]. Oncogene, 25(51): 6758-6780. DOI:10.1038/sj.onc.1209943 |
HUANG B, ZHANG L, TANG X, et al, 2016. Genome-wide analysis of alternative splicing provides insights into stress adaptation of the Pacific oyster[J]. Marine Biotechnology, 18: 598-609. DOI:10.1007/s10126-016-9720-x |
KAHLES A, ONG C S, ZHONG Y, et al, 2016. SplAdder: identification, quantification and testing of alternative splicing events from RNA-Seq data[J]. Bioinformatics, 32(12): 1840-1847. DOI:10.1093/bioinformatics/btw076 |
KANEKO T, GOLENBOCK D, SILVERMAN N, 2005. Peptidoglycan recognition by the Drosophila Imd pathway[J]. Journal of Endotoxin Research, 11(6): 383-389. DOI:10.1177/09680519050110060201 |
KIM M S, BYUN M, OH B H, 2003. Crystal structure of peptidoglycan recognition protein LB from Drosophila melanogaster[J]. Nature Immunology, 4(8): 787-793. DOI:10.1038/ni952 |
LITMAN G W, CANNON J P, DISHAW L J, 2005. Reconstructing immune phylogeny: new perspectives[J]. Nature Reviews Immunology, 5(11): 866-879. DOI:10.1038/nri1712 |
MANIATIS T, TASIC B, 2002. Alternative pre-mRNA splicing and proteome expansion in metazoans[J]. Nature, 418(6894): 236-243. DOI:10.1038/418236a |
MARQUEZ Y, BROWN J W S, SIMPSON C, et al, 2012. Transcriptome survey reveals increased complexity of the alternative splicing landscape in Arabidopsis[J]. Genome Research, 22(6): 1184-1195. DOI:10.1101/gr.134106.111 |
NAGATA S, TANAKA M, 2017. Programmed cell death and the immune system[J]. Nature Reviews Immunology, 17(5): 333-340. DOI:10.1038/nri.2016.153 |
NILSEN T W, GRAVELEY B R, 2010. Expansion of the eukaryotic proteome by alternative splicing[J]. Nature, 463(7280): 457-463. DOI:10.1038/nature08909 |
PAN Q, SHAI O, LEE L J, et al, 2008. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing[J]. Nature Genetics, 40(12): 1413-1415. DOI:10.1038/ng.259 |
RAMANI A K, CALARCO J A, PAN Q, et al, 2011. Genome-wide analysis of alternative splicing in Caenorhabditis elegans[J]. Genome Research, 21(2): 342-348. DOI:10.1101/gr.114645.110 |
ROMEO Y, LEMAITRE B, 2008. Drosophila immunity [M] // EWBANK J, VIVIER E. Innate Immunity. Totowa, NJ: Humana Press: 379-394.
|
STAMM S, BEN-ARI S, RAFALSKA I, et al, 2005. Function of alternative splicing[J]. Gene, 344: 1-20. DOI:10.1016/j.gene.2004.10.022 |
TAKEDA K, KAISHO T, AKIRA S, 2003. Toll-like receptors[J]. Annual Review of Immunology, 21(1): 335-376. DOI:10.1146/annurev.immunol.21.120601.141126 |
TAN S X, WANG W W, TIAN C X, et al, 2019. Post-transcriptional regulation through alternative splicing after infection with Flavobacterium columnare in channel catfish (Ictalurus punctatus)[J]. Fish & Shellfish Immunology, 91: 188-193. DOI:10.11964/jfc.20180811427 |
TRAPNELL C, HENDRICKSON D G, SAUVAGEAU M, et al, 2013. Differential analysis of gene regulation at transcript resolution with RNA-seq[J]. Nature Biotechnology, 31(1): 46-53. DOI:10.1038/nbt.2450 |
TRAPNELL C, ROBERTS A, GOFF L, et al, 2012. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J]. Nature Protocols, 7(3): 562-578. DOI:10.1038/nprot.2012.016 |
TRAPNELL C, WILLIAMS B A, PERTEA G, et al, 2010. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 28(5): 511-515. DOI:10.1038/nbt.1621 |
VAN GORP H, KUCHMIY A, VAN HAUWERMEIREN F, et al, 2014. NOD-like receptors interfacing the immune and reproductive systems[J]. FEBS Journal, 281(20): 4568-4582. DOI:10.1111/febs.13014 |
ZHANG G F, FANG X D, GUO X M, et al, 2012. The oyster genome reveals stress adaptation and complexity of shell formation[J]. Nature, 490(7418): 49-54. DOI:10.1038/nature11413 |
ZHANG L L, LI L, GUO X M, et al, 2015. Massive expansion and functional divergence of innate immune genes in a protostome[J]. Scientific Reports, 5(1): 8693. DOI:10.1038/srep08693 |
 2024, Vol. 55
2024, Vol. 55


