
中国海洋湖沼学会主办。
文章信息
- 王庚申, 颜懿, 李彤, 董鹏生, 谢建军, 许文军, 张德民, 张化俊. 2024.
- WANG Geng-Shen, YAN Yi, LI Tong, DONG Peng-Sheng, XIE Jian-Jun, XU Wen-Jun, ZHANG De-Min, ZHANG Hua-Jun. 2024.
- 漂白粉消毒后对虾养殖源水细菌群落的响应特征研究
- EFFECTS OF BLEACHING POWDER DISINFECTION FOR SOURCE WATER OF SHRIMP FARMING ON BACTERIAL COMMUNITY
- 海洋与湖沼, 55(1): 202-212
- Oceanologia et Limnologia Sinica, 55(1): 202-212.
- http://dx.doi.org/10.11693/hyhz20230800163
文章历史
-
收稿日期:2023-08-08
收修改稿日期:2023-10-29
2. 宁波大学海洋学院 浙江宁波 315211;
3. 浙江省海洋水产研究所 浙江省海水增养殖重点实验室 浙江舟山 316021
2. School of Marine Sciences, Ningbo University, Ningbo 315211, China;
3. Zhejiang Marine Fisheries Research Institute, Key Laboratory of Mariculture and Enhancement of Zhejiang Province, Zhoushan 316021, China
为了防控凡纳滨对虾(Litopenaeus vannamei)的传染性疾病, 对源水消毒是养殖的关键环节之一。漂白粉是对虾养殖源水消毒中常用的一种卤素类消毒剂, 具有价格低、杀菌力强、使用方便等特点。漂白粉是氢氧化钙、氯化钙、次氯酸钙的混合物, 溶解在水中时水解成次氯酸离子和氯离子。次氯酸离子的强氧化作用可以杀死细菌, 氯离子也表现出一定的杀菌功能(Ma et al, 2019)。研究表明, 大多数病原对次氯酸钙敏感, 且杀菌效果与剂量有关(Park et al, 2004; Limsuwan et al, 2008; Cha et al, 2012)。一般认为, 对养殖源水消毒可以有效去除病原微生物, 但由于消毒机制的非特异性, 水体中的有益微生物也会被杀灭。由于细菌类群生理及群落组成的多样性, 水体中不同的微生物类群会对消毒表现出不同的响应或恢复能力(Becerra-Castro et al, 2016)。目前, 对消毒后养殖源水细菌群落的恢复动态仍知之甚少。
抗生素抗性基因(antibiotic resistant genes, ARGs)可以通过水平转移将移动遗传元件转至病原菌中, 使病原菌产生抗生素抗性, 威胁公共卫生健康(Jo et al, 2021)。由于河流汇入及水产养殖活动, ARGs在近海海区环境中普遍存在(Hedberg et al, 2018), 近海养殖源水是对虾养殖池塘环境中ARGs的主要来源(夏涛涛等, 2022), 在华南及华东地区的对虾养殖源水中均检测到了多种养殖中常见抗生素的ARGs (Wang et al, 2019a; Yuan et al, 2019)。近几年, 城市污水、饮用水处理领域的研究显示, 氯消毒可以有效降低水中的ARGs丰度(Yoon et al, 2017; Wang et al, 2020), 水产养殖用水消毒的研究也证实了这一点(赵晓雨等, 2021)。但氯消毒不能完全去除水体中的ARGs (Yuan et al, 2015), 即使携带ARGs的细菌被杀灭, ARGs仍可以释放到水环境中通过水平转移进行传播(姜瀚集等, 2019)。然而, 关于养殖源水消毒后ARGs丰度的动态变化规律尚未见报道。
养殖水体细菌群落是对虾养殖系统的重要组成, 对维持水质稳定及对虾健康具有重要意义(杜世聪等, 2019)。一般认为, 源水进入养殖系统后除改善水体理化因子外, 也会影响水体微生物群落组成(Heyse et al, 2021)。有研究报道氯消毒会引起水体细菌类群的异常增殖, 当消毒后病原菌和耐抗生素细菌增殖时, 可能导致潜在的微生物健康风险(Lin et al, 2016; Deng et al, 2019; Wang et al, 2021a)。因此, 了解漂白粉消毒后养殖源水细菌群落、病原菌及抗生素抗性基因的恢复动态, 尤其是不同消毒浓度的影响, 对优化对虾养殖系统微生物管控策略具有重要的指导作用。基于此, 本研究利用不同浓度漂白粉消毒养殖源水, 通过高通量测序比较消毒前后养殖源水的理化因子、细菌群落结构及病原菌丰度变化, 同时利用RT-qPCR检测养殖源水ARGs的丰度变化, 探究源水细菌群落对不同浓度漂白粉消毒后的响应特征, 以期为对虾养殖生产中漂白粉的使用提供理论基础。
1 材料与方法 1.1 实验材料与设计本实验在浙江省海洋水产研究所试验场进行, 实验用对虾养殖源水取自周边海域, 温度27.3~ 27.9 ℃, 盐度25.5~25.7, pH 8.25~8.21。漂白粉购自湖北省应城市利必康化工有限公司, 有效氯含量28%~35%。
实验在室内300 L塑料水槽中进行, 共设置2个高、低漂白粉消毒浓度: 60、20 mg/L, 每组4个重复。消毒前(DC)、消毒后余氯衰减至0.1 mg/L时0~3 d (D0、D1、D2、D3)定时取2 L水样, 1 L用于水质理化指标测定; 1 L用0.22 μm滤膜过滤后保存于–80 ℃, 用于检测微生物和抗性基因。
采用HANNA HI-97734余氯和总氯便携光度计(DPD法, 量程0.00~10.00 mg/L, 精度为±0.03 mg/L)测定水体中的余氯浓度; 用YSI Pro2030测定水体溶解氧, YSI Pro10测定水体pH。水体理化因子化学需氧量(COD)采用碱性高锰酸钾法, 氨氮(NH4+-N)采用靛酚蓝分光光度比色法, 亚硝酸盐氮(NO2-N)采用萘乙二胺分光光度法, 硝酸盐氮(NO3-N)采用锌-铬还原法, 活性磷酸盐(AP)用磷钼蓝分光光度法测定。
1.2 DNA提取、高通量测序使用PowerSoil ®Pro Kit (Qiagen)试剂盒提取水样DNA, 通过0.8%琼脂糖凝胶电泳检测提取DNA的质量, 用紫外分光光度计检测DNA的浓度和纯度。用带Barcode的338F (5′-ACTCCTACGGGAGGCAGCAG-3′)和806R (5′-GGACTA CHVGGGTWTCTAAT-3′)引物对细菌16S rRNA的V3~V4区进行PCR扩增。PCR扩增条件: 98 ℃预变性30 s; 98 ℃变性15 s, 50 ℃退火30 s, 72 ℃延伸30 s, 27个循环; 72 ℃延伸5 min。反应体系(25 μL): 5 μL 5×High GC Buffer、5 μL 5×Reaction Buffer、2 μL 10 mmol/L dNTPs、2 μL引物(10 μmol/L)、0.25 μL FastPfu聚合酶; 2 μL模板DNA, 灭菌双蒸水补足25 μL。PCR产物经纯化、定量后, 使用TruSeq Nano DNA LT Library Prep Kit建库, 构建的文库经质检和定量后, 使用Illumina NovaSeq平台PE250进行双端测序。
1.3 测序数据处理使用QIIME2 2021.2版本对原始数据进行质量控制, 去除接头, 利用DADA2插件去噪, 并生成扩增子序列变体(ASV)表。以每个ASV中丰度最高的作为代表序列, 在SILVA138数据库中比对确定其所属类群。去除叶绿体(Chloroplast)、线粒体(Mitochondria)及未被鉴定的ASVs, 共获得了2933个ASVs。
1.4 水环境细菌病原数据库分析将实验样品中每个ASV的代表序列在水环境细菌病原数据库(DPiWE, dayuz.com) (董鹏生等, 2021)进行BLAST比对、注释, 参数阈值E-value < 1×10–6, 序列同源性 > 98.5%, 最后输出病原菌比对信息, 共比对获得病原菌169个ASV。由于数据库注释信息的差异, 所得病原菌序列以病原菌库的注释信息为准。
1.5 抗性基因检测采用Real-time qPCR方法对常见的8种ARGs (表 1)进行定量分析。qPCR在罗氏LightCycler480上使用AceQ qPCR SYBR Green Master Mix (Vazyme)进行, 反应体系: 10 μL 2×SYBR real-time PCR premixture、0.8引物(10 μmol/L)、1 μL模板DNA, 灭菌双蒸水补足20 μL。PCR反应条件: 95 ℃ 5 min; 95 ℃ 15 s, 55~62 ℃ 30 s, 72 ℃ 30 s, 40个循环, 之后进行熔解曲线分析。通过Ct值和标准曲线计算基因拷贝数, 以每毫升养殖源水的拷贝数来测定ARGs的浓度。
| 抗生素类型 | 抗性基因 | 引物序列5′~3′ | 退火温度/℃ | 片段长度/bp |
| 磺胺类(Pei et al, 2006) | sul1 | FW GCACCGGAAACATCGCTGCAC RV GAAGTTCCGCCGCAAGGCTCG |
62 | 163 |
| sul2 | FW TCCGGTGGAGGCCGGTATCTGG RV CGGGAATGCCATCTGCCTTGAG |
62 | 191 | |
| 氯霉素类(Li et al, 2013) | cmlA | FW GCCAGCAGTGCCGTTTAT RV GGCCACCTCCCAGTAGAA |
55 | 158 |
| floR | FW CGGTCGGTATTGTCTTCACG RV TCACGGGCCACGCTGTAT |
56 | 171 | |
| cfr | FW TGTGCTACAGGCAACATTGGAT RV CAAATACTTGACGGTTGGCTAGAG |
55 | 148 | |
| 四环素类(Aminov et al, 2001) | tetA | FW GCGCGATCTGGTTCACTCG RV AGTCGACAGYRGCGCCGGC |
62 | 164 |
| tetQ | FW AGAATCTGCTGTTTGCCAGTG RV CGGAGTGTCAATGATATTGCA |
55 | 169 | |
| 氨基糖苷类(Zhu et al, 2018) | strB | FW GCTCGGTCGTGAGAACAATCT RV CAATTTCGGTCGCCTGGTAGT |
56 | — |
通过Bray-Curtis距离进行非度量多维尺度分析(NMDS)分析, 展示漂白粉消毒后水体细菌群落的演替规律。基于Bray-Curtis距离的相似性分析(ANOSIM)分析各组细菌群落的相似性。通过R中的“pheatmap”软件包绘制热图。使用“psych”软件包计算类群相关性矩阵, 保留皮尔森相关性系数|r| > 0.6, 且P < 0.05的数据矩阵, 使用Gephi(v0.9.2)构建微生物群落的共现网络, 并进行加权随机模块化网络分析。利用SPSS 20.0在用单因素方差分析(one-way ANOVA)分析样品间ARGs丰度的差异(P < 0.05)。
2 结果 2.1 消毒前后对虾养殖源水水体理化因子变化实验期间, 对虾养殖源水温度在27.5~32.9 ℃, 盐度在漂白粉消毒后小幅升高, 高浓度组始终高于低浓度组; 溶解氧、化学需氧量浓度在消毒后(D0)迅速下降并保持稳定; pH、硝酸盐浓度在消毒后(D0)升高随后保持稳定; 亚硝酸盐浓度在消毒后(D0)升高, 之后大幅震荡, 氨氮浓度则在消毒后缓慢上升; 活性磷酸盐浓度在消毒后出现分化, 低浓度消毒组始终高于高浓度组(图 1)。
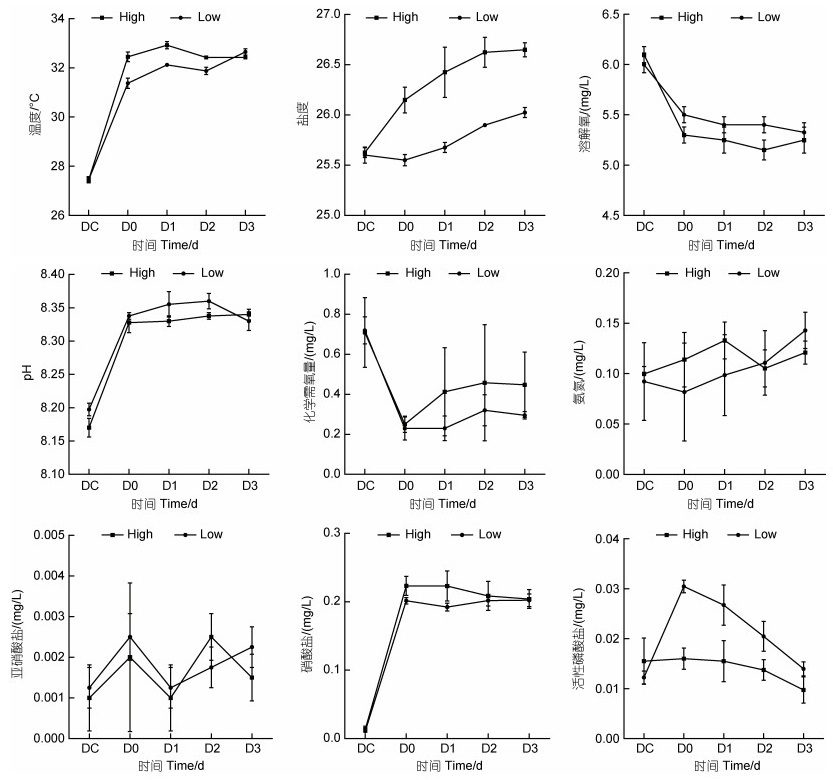 |
| 图 1 消毒前后对虾养殖源水水质理化因子变化 Fig. 1 Changes of physicochemical factors of shrimp source water before and after bleaching powder disinfection |
消毒后, 高、低浓度组细菌类群门水平组成均发生了显著的变化, 拟杆菌门(Bacteroidota)的相对丰度较消毒前均显著升高(P < 0.05), 高浓度组变形菌门(Proteobacteria)的相对丰度则显著下降(P < 0.05) (图 2a)。消毒前(DC), 高浓度组养殖源水的主要门类包括变形菌门(53.71%)、拟杆菌门(27.15%)、放线菌门(Actinobacteriota, 9.32%)、Patescibacteria (1.84%)及蓝细菌门(Cyanobacteria, 1.46%)等; 消毒后余氯衰减至0.1 mg/L时(D0), 优势菌门为变形菌门(25.54%)、拟杆菌门(73.91%)。而低浓度组在消毒前(DC)的主要门类为变形菌门(74.74%)、拟杆菌门(12.76%)、蓝菌门(7.34%)、放线菌门(2.76%)及Patescibacteria (1.55%)等; 消毒后(D0), 优势菌门为变形菌门(69.26%)、拟杆菌门(29.19%)。消毒后第1~2天, 高浓度组的优势菌门以变形菌门(79.90%、51.17%)、拟杆菌门(19.83%、44.85%)为主, 而低浓度组则以变形菌门为优势菌, 相对丰度达到了98.78%、95.78%。至第3天, 高浓度组的优势菌门均以变形菌门(91.58%)、蛭弧菌门(Bdellovibrionota) (5.17%)、拟杆菌门(2.19%)为主, 低浓度组的优势菌门为变形菌门(87.92%)、蛭弧菌门(6.34%)、拟杆菌门(2.79%)、Patescibacteria (1.67%)。
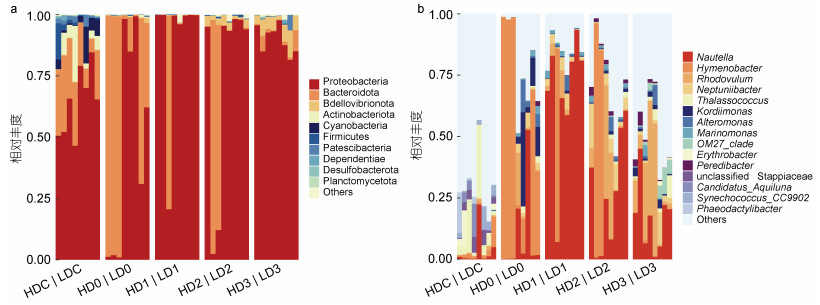 |
| 图 2 消毒前后对虾养殖源水细菌群落相对丰度图 Fig. 2 Relative abundance of bacterial community in shrimp source water before and after bleach disinfection 注: a. 门水平(相对丰度 > 0.1%); b. 属水平(相对丰度 > 0.2%)。H代表高浓度组, L代表低浓度组。下同 |
从属水平看(图 2b), 消毒前高低浓度组养殖源水的优势菌属包括Thalassococcus、Nautella、unclassified Stappiaceae、Candidatus_Aquiluna、Erythrobacter、Synechococcus_CC9902、Hymenobacter、Phaeodactylibacter, 以上菌属的总相对丰度在高、低浓度组分别占比27.80%、29.61%。消毒后第0~2天, 高浓度组中薄层菌属(Hymenobacter, 19.77%~73.88%)和Nautella (5.26%~55.98%)交替占据优势, 第3天, 薄层菌属丰度迅速下降。而低浓度组中薄层菌属(29.06%)仅在第0天占据优势, Nautella (37.40%~78.34%)则在第1~2天维持优势。消毒后第2天, 高、低浓度组的小红卵菌属(Rhodovulum)丰度开始升高, 第3天在两组中均占据优势(24.08%、14.52%), 其次是Nautella (22.00%、12.12%)。
在ASV水平上(图 3), 源水细菌群落中相对丰度最高的50个ASVs主要归类于红杆菌科Rhodobacteraceae (16个)、交替单胞菌科Alteromonadaceae (4个)、黄杆菌科Flavobacteriaceae (3个)和Nitrincolaceae (3个), 其中21个ASVs在消毒后相对丰度下降, 22个ASVs相对丰度升高。消毒后, ASV848 (Nautella)在高、低浓度组的相对丰度均较消毒前显著上升, 归类于薄层菌属的ASV1561、ASV953的相对丰度在第1天显著升高, 之后开始下降; 红杆菌科的ASV1480、ASV1249、ASV2053仅在高浓度组显著下降, ASV1764 (小红卵菌属)则在高浓度组中显著上升。
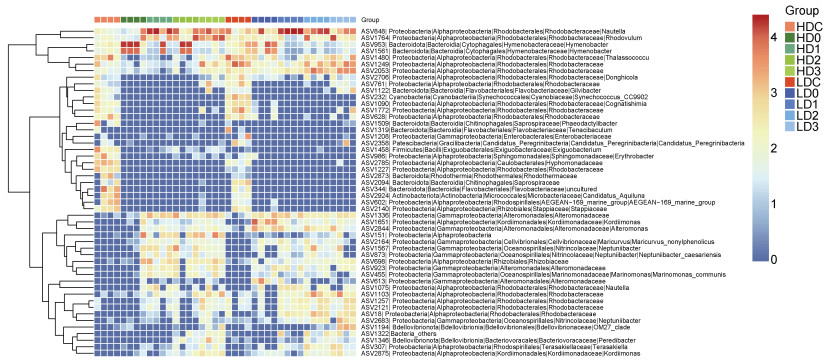 |
| 图 3 消毒前后对虾养殖源水中的优势ASVs热图(相对丰度前50个ASVs) Fig. 3 Dominant ASVs of shrimp source water before and after bleaching powder disinfection (top 50 ASVs in relative abundance) |
基于Bray-Curtis距离的NMDS分析发现消毒后水体细菌群落与消毒前分区明显, 而高、低浓度组未完全分离(图 4); ANOSIM分析也发现, 消毒前、后细菌群落差异显著(P < 0.05), 而消毒后高、低浓度之间差异不显著(P > 0.05)。
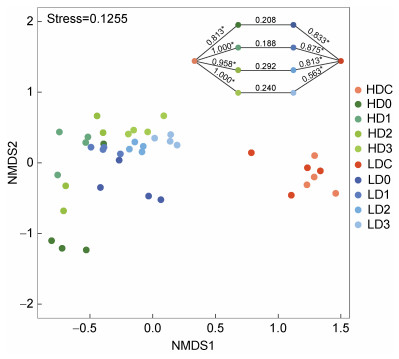 |
| 图 4 基于Bray-Curtis距离的NMDS分析水体细菌群落结构 Fig. 4 NMDS analysis of bacterial community structure in source water based on the Bray-Curtis distance 注: 图中右上角数值即为ANOSIM的R值。R值越接近1, 表明组间差异越大于组内差异。*表示统计具有显著性(P < 0.05) |
消毒前及高、低浓度组消毒后的水体细菌群落的共现网络包含425、238、348个节点, 分别由1 776、1 137、1 958个边连接(图 5a~5c, 表 2), 说明消毒后节点减少, 低浓度消毒会增加群落间的相互作用。相比消毒前, 消毒后水体细菌群落共现网络的模块化系数、平均聚类系数降低, 而其他网络拓扑学参数则升高; 消毒后, 除了网络直径外, 高浓度组共现网络的平均聚类系数、平均路径长度、平均度均低于低浓度组。高、低浓度组的网络分别由6个主要模块构成, 分别占整个网络的69.74% (9.24%~13.87%)、79.38% (8.33%~18.39%), 而对照组模块较为平均, 模块Ⅰ到模块Ⅵ仅占整个网络的26.6% (4%~4.71%)。
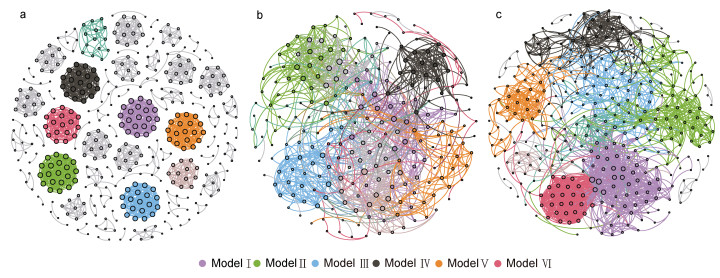 |
| 图 5 消毒前后水体微生物群落共现网络 Fig. 5 Co-occurrence network of bacterial communities 注: a. 消毒前; b. 高浓度消毒后; c. 低浓度消毒后 |
| 时间 | 节点 | 边 | 模块化系数 | 平均聚类系数 | 网络直径 | 平均路径长度 | 平均度 |
| 消毒前 | 425 | 1 776 | 0.94 | 0.934 | 5 | 1.077 | 8.358 |
| 高浓度组消毒后 | 238 | 1 137 | 0.747 | 0.487 | 14 | 4.176 | 9.555 |
| 低浓度组消毒后 | 348 | 1 958 | 0.738 | 0.604 | 11 | 4.519 | 11.253 |
使用DPiWE数据库对消毒前后的水体细菌高通量测序数据进行序列比对和病原菌物种注释, 消毒前水体病原菌有23属, 总序列数为6 461; 消毒后高浓度组病原菌有37属, 总序列数为2 912, 低浓度组有31属, 总序列数为6 584, 说明消毒后病原菌种类增加, 而高浓度组总序列数显著下降。
对虾养殖源水的优势病原菌(平均相对丰度大于0.01%)丰度占总病原菌丰度的95.59%, 其中50%为鱼类病原, 50%为跨宿主共患病病原。消毒前, 优势病原菌为假埃希氏菌(Pseudescherichia, 1.56%)、微球菌属(Micrococcus, 0.53%)、Thalassobius (0.23%)等; 消毒后, 高浓度组的优势病原菌Thalassobius (0.32%)、不动杆菌属(Acinetobacter, 0.10%), 低浓度组则为Thalassobius (0.88%)、假交替单胞菌属(Pseudoalteromonas, 0.13%)等。水中优势病原菌在消毒后具有一定时间上的差异, 而Thalassobius在高、低浓度组均逐渐占据绝对优势。值得一提的是, 弧菌属(Vibro)病原菌的相对丰度在消毒后明显下降, 但在源水中仍存在(图 6)。
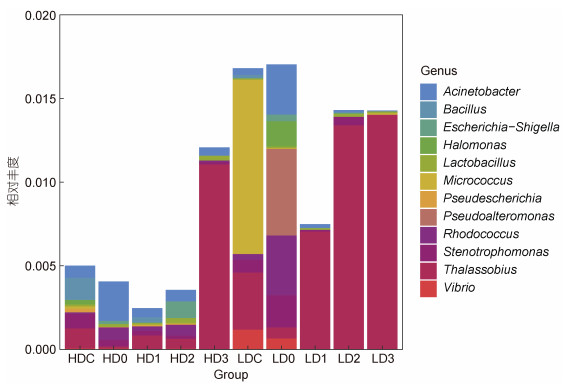 |
| 图 6 消毒前后病原菌的相对丰度堆积图 Fig. 6 Relative abundance of pathogenic bacteria before and after bleaching powder disinfection |
本研究中, 4大类8个ARGs在消毒前的养殖源水中均有检出(图 7), tetA是丰度最高的ARG, 达到107 copies/mL, cfr是丰度最低的ARG (102 copies/mL), 其他ARGs的丰度在103~104 copies/mL水平。消毒后, 余氯衰减至0.1 mg/L时(D0), 高、低浓度组sul1、floR、cfr、tetQ的丰度较消毒前(DC)显著下降(P < 0.05), 而tetA、strB、cmlA、sul2的丰度变化趋势在高、低浓度组出现分化。sul1、floR的丰度均在随后的1~2 d内恢复到消毒前的数量级水平, 而cfr、tetQ基因在实验期间未能恢复到消毒前的数量级。
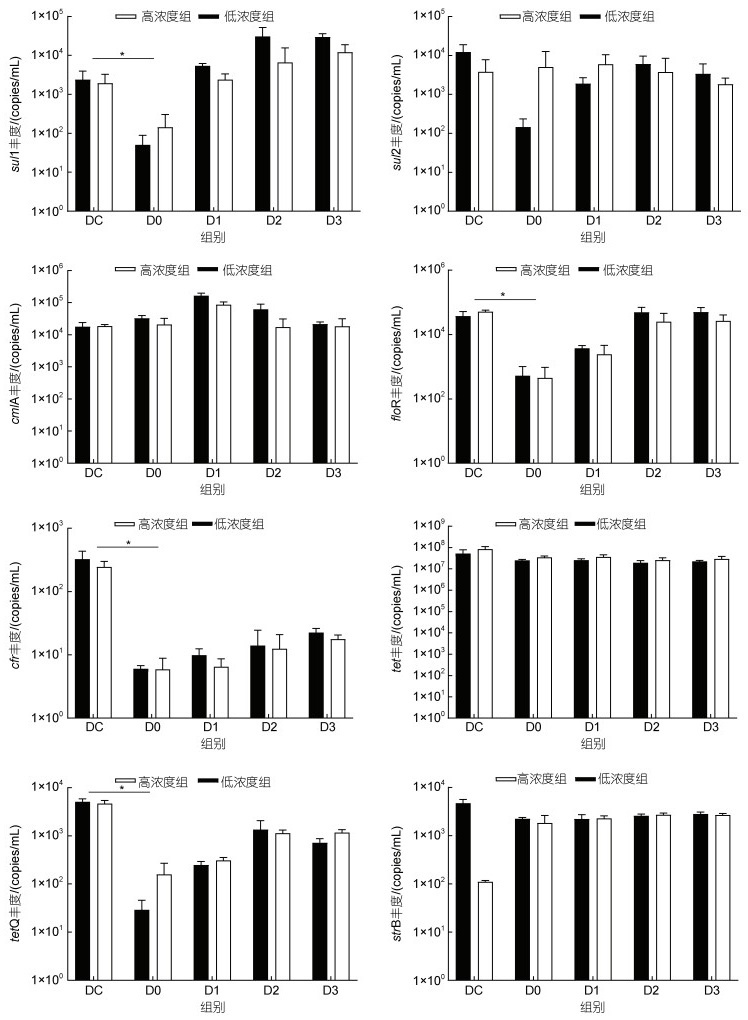 |
| 图 7 消毒前后8种ARGs的动态变化 Fig. 7 Changes of abundances of 8 ARGs before and after bleaching powder disinfection |
含氯消毒剂在海水养殖疾病预防中的应用已被养殖业者广泛接受, 但由于水体中不同细菌对消毒剂的敏感性存在差异, 消毒后水体的细菌群落结构的恢复动态值得关注。研究表明, 氯消毒会改变饮用水的细菌群落组成, 降低群落结构多样性(Holinger et al, 2014; Dai et al, 2020)。Becerra-Castro等(2016)研究发现紫外线、臭氧及光催化臭氧化等多种方式消毒城市污水和地表水, 3 d后的两种类型水体的变形菌门丰度明显上升。Duan等(2020)报道漂白粉消毒对虾育苗用水5 d后, 变形菌门和拟杆菌门丰度上升, 放线菌门丰度下降。本研究中, 漂白粉消毒后养殖源水的细菌群落与消毒前差异显著, 但高低浓度组之间无显著差异。消毒后(D0), 高浓度组变形菌门丰度明显下降, 拟杆菌门占据主导地位, 而低浓度组变形菌门丰度无明显变化, 说明高浓度漂白粉对变形菌门的杀灭效果更强, 存在剂量依赖效应。消毒后第3天, 高、低浓度组细菌群落组成趋于一致, 变形菌门丰度占据优势, 表明消毒后变形菌门类群可快速恢复。共现网络的复杂性代表了不同样本微生物群落之间的根本差异(Shi et al, 2016), 网络的拓扑学参数可以表征微生物之间的相互作用(Barberán et al, 2012)。漂白粉消毒后, 细菌网络节点数量减少, 模块化程度降低, 说明消毒降低了细菌的共现网络复杂程度。低浓度组共现网络节点之间的连接最为复杂, 并且网络中心性最高, 表明消毒后低浓度的水体细菌群落稳定性高于高浓度组。
从属水平看, 消毒后(D0)养殖源水优势属的数量减少, 但总相对丰度增加, 与Wan等(2022)的研究结果一致。属水平的变化趋势与ASV水平相似, 主要由单个或几个优势ASV的丰度变化主导。消毒后0~2 d, 薄层菌属和Nautella在占据主导地位, 高浓度组薄层菌属丰度高于低浓度组; 第3天, 小红卵菌属在高低浓度组均占据优势, 其次是Nautella丰度, 且高浓度丰度均高于低浓度组。有研究认为, 薄层菌属具有辐射抗性, 能适应多种极端环境(Collins et al, 2000)。Nautella常见于海水养殖环境及对虾肠道中(李秋芬等, 2011; Sakami et al, 2014; Zheng et al, 2016), 但尚未发现对凡纳滨对虾的致病性。小红卵菌属是一类光养细菌, 分泌多种杀菌活性物质, 对水产动物有益生作用(Loo et al, 2013; Chang et al, 2019; Koga et al, 2021)。
DPiWE是一个基于高通量测序数据分析水体环境病原菌群落特征和溯源的综合数据库, 提供了人类介水传染病、水产动物疾病及跨宿主共患病等病害的细菌病原的分类学和宿主信息(董鹏生等, 2021)。通过DPiWE数据库比对, 养殖源水中优势病原菌为埃希氏菌、微球菌属及Thalassobius等, 与象山大黄鱼网箱养殖区水体中优势病原菌(弧菌属、黏着杆菌属Tenacibaculum、埃希氏-志贺氏菌属Escherichia-Shigella和不动杆菌属)不同, 且总体丰度偏低(Hou et al, 2021), 说明本实验源水环境受到水产养殖活动影响的程度低。漂白粉消毒后病原菌总丰度降低, 但种类增加, 与病原菌对氯消毒的抗性有关(Song et al, 2019; Wang et al, 2019b), 如消毒后新增链球菌Streptococcus、梭状芽孢杆菌Clostridium、埃希氏-志贺氏菌等病原菌被普遍报道具有氯消毒抗性(Wang et al, 2021b)。高浓度组病原菌在消毒后初期丰度下降, 而低浓度组在D0时高于消毒前, 说明高浓度漂白粉对病原菌有更好地杀灭作用。源水病原菌组成与丰度存在明显的动态演替, 实验后期均以Thalassobius占主导地位, 有研究认为Thalassobius中包含多种鱼类病原菌, 但未见对虾类致病的报道(Kanehisa, 2018)。
本研究中, 养殖源水中丰度最高的ARG是tetA, 其次是cmlA与floR, 与文献报道对虾养殖近海水源水主要为磺胺类ARGs (sul2)不同, 养殖源水中ARGs丰度与该水产养殖区常用的抗生素类型有关(夏涛涛等, 2022), 同时受人类活动、生活污水排入、降雨等多种因素影响(Shao et al, 2018; Liu et al, 2020)。有研究表明, 氯消毒能有效去除水体中的ARGs (Yoon et al, 2017), 去除效率与有效氯浓度有关(Huang et al, 2013)。本实验中, 漂白粉消毒后(D0)仅对sul1、floR、cfr、tetQ有一定的去除效果, 且在有效氯衰减后短时间内恢复至消毒前水平, 说明漂白粉消毒养殖源水不能有效去除ARGs (Tang et al, 2023)。值得注意的是, 消毒后高浓度组sul1、sul2、tetA、tetQ等ARGs的丰度高于低浓度组, 分析原因可能与消毒后携带相关抗性基因的细菌快速再生有关(Sullivan et al, 2017)。氯消毒对水体ARGs的去除具有选择性, 对引用水的研究发现, 氯消毒除对sul1有明显的去除作用外, 对ampC、aphA2、blatem1、tetA、tetG、ermA、ermB等ARGs均有富集作用(Shi et al, 2013)。Wan等(2022)也报道氯消毒30 min对多种ARGs的丰度没有影响。本实验也有类似结果, tetA、cmlA和strB的丰度在消毒后无明显变化, 但具体机制有待进一步研究。
4 结论研究发现, 漂白粉消毒改变了源水的细菌群落结构及共现网络的复杂性。消毒后, 两个浓度组源水的拟杆菌门丰度上升, 而60 mg/L漂白粉组变形菌门的丰度显著降低; 属水平上, 薄层菌属和Nautella在消毒后源水中占据优势; 至第3天, 两组的细菌群落组成在门和属水平趋于一致。60 mg/L漂白粉对病原菌丰度和恢复速度的抑制作用优于20 mg/L。漂白粉消毒对养殖源水中sul1、floR、cfr、tetQ有一定的去除作用, 但对其他ARGs几乎无影响, 显示了对ARGs去除的选择性, 且高低浓度组之间无差异。综合分析认为, 相比20 mg/L组, 60 mg/L漂白粉消毒可以显著降低变形菌门的丰度, 抑制病原菌快速增殖, 可以作为凡纳滨对虾养殖中更好地消毒选择。
杜世聪, 黄雷, 杨坤杰, 等, 2019. 凡纳滨对虾健康状态分化前后养殖水体浮游细菌群落的比较[J]. 生态学杂志, 38(8): 2456-2465. |
李秋芬, 傅雪军, 张艳, 等, 2011. 循环水养殖系统生物滤池细菌群落的PCR-DGGE分析[J]. 水产学报, 35(4): 579-586. |
赵晓雨, 苏浩昌, 徐煜, 等, 2021. 渔用氧化剂对水源水和池塘水中磺胺类抗性基因sul1的去除作用[J]. 南方水产科学, 17(3): 46-53. |
姜瀚集, 付佳伦, 金敏, 等, 2019. 细菌耐药基因在斑马鱼体内转移规律的初步研究[J]. 环境与健康杂志, 36(5): 382-387. |
夏涛涛, 苏浩昌, 胡晓娟, 等, 2022. 强氯精对对虾养殖用水中抗生素抗性基因的影响[J]. 南方水产科学, 18(6): 85-92. |
董鹏生, 郭海朋, 王艳婷, 等, 2021. 水环境细菌病原数据库的构建及应用[J]. 水产学报, 45(11): 1921-1933. |
AMINOV R I, GARRIGUES-JEANJEAN N, MACKIE R I, 2001. Molecular ecology of tetracycline resistance: development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins[J]. Applied and Environmental Microbiology, 67(1): 22-32. DOI:10.1128/AEM.67.1.22-32.2001 |
BARBERÁN A, BATES S T, CASAMAYOR E O, et al, 2012. Using network analysis to explore co-occurrence patterns in soil microbial communities[J]. The ISME Journal, 6(2): 343-351. DOI:10.1038/ismej.2011.119 |
BECERRA-CASTRO C, MACEDO G, SILVA A M T, et al, 2016. Proteobacteria become predominant during regrowth after water disinfection[J]. Science of the Total Environment, 573: 313-323. DOI:10.1016/j.scitotenv.2016.08.054 |
CHA C N, LEE Y E, KANG I J, et al, 2012. Bactericidal efficacies of an aquatic disinfectant tablet composed to Calcium hypochlorite against Vibrio anguillarum and Streptococcus iniae[J]. Journal of Food Hygiene and Safety, 27(3): 290-294. DOI:10.13103/JFHS.2012.27.3.290 |
CHANG B V, LIAO C S, CHANG Y T, et al, 2019. Investigation of a farm-scale multitrophic recirculating aquaculture system with the addition of Rhodovulum sulfidophilum for milkfish (Chanos chanos) coastal aquaculture[J]. Sustainability, 11(7): 1880. DOI:10.3390/su11071880 |
COLLINS M D, HUTSON R A, GRANT I R, et al, 2000. Phylogenetic characterization of a novel radiation-resistant bacterium from irradiated pork: description of Hymenobacter actinosclerus sp. nov[J]. International Journal of Systematic and Evolutionary Microbiology, 50(2): 731-734. DOI:10.1099/00207713-50-2-731 |
DAI Z H, SEVILLANO-RIVERA M C, CALUS S T, et al, 2020. Disinfection exhibits systematic impacts on the drinking water microbiome[J]. Microbiome, 8(1): 42. DOI:10.1186/s40168-020-00813-0 |
DENG S X, YAN X T, ZHU Q Q, et al, 2019. The utilization of reclaimed water: Possible risks arising from waterborne contaminants[J]. Environmental Pollution, 254: 113020. DOI:10.1016/j.envpol.2019.113020 |
DUAN Y F, TANG Y P, HUANG J H, et al, 2020. Changes in the microbial community of Litopenaeus vannamei larvae and rearing water during different growth stages after disinfection treatment of hatchery water[J]. Journal of Microbiology, 58(9): 741-749. DOI:10.1007/s12275-020-0053-0 |
HEDBERG N, STENSON I, PETTERSSON M N, et al, 2018. Antibiotic use in Vietnamese fish and lobster sea cage farms; implications for coral reefs and human health[J]. Aquaculture, 495: 366-375. DOI:10.1016/j.aquaculture.2018.06.005 |
HEYSE J, PROPS R, KONGNUAN P, et al, 2021. Rearing water microbiomes in white leg shrimp (Litopenaeus vannamei) larviculture assemble stochastically and are influenced by the microbiomes of live feed products[J]. Environmental Microbiology, 23(1): 281-298. DOI:10.1111/1462-2920.15310 |
HOLINGER E P, ROSS K A, ROBERTSON C E, et al, 2014. Molecular analysis of point-of-use municipal drinking water microbiology[J]. Water Research, 49: 225-235. DOI:10.1016/j.watres.2013.11.027 |
HOU D D, HONG M, WANG Y T, et al, 2021. Assessing the risks of potential bacterial pathogens attaching to different microplastics during the summer–autumn period in a mariculture cage[J]. Microorganisms, 9(9): 1909. DOI:10.3390/microorganisms9091909 |
HUANG J J, HU H Y, WU Y H, et al, 2013. Effect of chlorination and ultraviolet disinfection on tetA-mediated tetracycline resistance of Escherichia coli[J]. Chemosphere, 90(8): 2247-2253. DOI:10.1016/j.chemosphere.2012.10.008 |
JO H, RAZA S, FAROOQ A, et al, 2021. Fish farm effluents as a source of antibiotic resistance gene dissemination on Jeju Island, South Korea[J]. Environmental Pollution, 276: 116764. DOI:10.1016/j.envpol.2021.116764 |
KANEHISA M, 2018. Inferring antimicrobial resistance from pathogen genomes in KEGG [M] // MAMITSUKA H. Data Mining for Systems Biology. New York: Humana Press: 225-239.
|
KOGA A, TANI Y, OZAKI K I, et al, 2021. The effects of a marine photosynthetic bacteria Rhodovulum sulfidophilum on the growth and survival rate of Marsupenaeus japonicus (kuruma shrimp)[J]. Journal of Aquaculture, Fisheries & Fish Science, 3(2): 245-249. |
LI J, SHAO B, SHEN J Z, et al, 2013. Occurrence of chloramphenicol-resistance genes as environmental pollutants from swine feedlots[J]. Environmental Science & Technology, 47(6): 2892-2897. |
LIMSUWAN C, CHUCHIRD N, LAISUTISAN K, 2008. Efficacy of Calcium hypochlorite on the prevalence of Microsporidiosis (Thelohania) in pond-reared Litopenaeus vannamei[J]. Kasetsart Journal - Natural Science, 42(2): 282-288. |
LIN Y W, LI D, GU A Z, et al, 2016. Bacterial regrowth in water reclamation and distribution systems revealed by viable bacterial detection assays[J]. Chemosphere, 144: 2165-2174. DOI:10.1016/j.chemosphere.2015.10.071 |
LIU S, SU H C, PAN Y F, et al, 2020. Spatial and seasonal variations of antibiotics and antibiotic resistance genes and ecological risks in the coral reef regions adjacent to two typical islands in South China Sea[J]. Marine Pollution Bulletin, 158: 111424. DOI:10.1016/j.marpolbul.2020.111424 |
LOO P L, CHONG V C, VIKINESWARY S, 2013. Rhodovulum sulfidophilum, a phototrophic bacterium, grown in palm oil mill effluent improves the larval survival of marble goby Oxyeleotris marmorata (Bleeker)[J]. Aquaculture Research, 44(3): 495-507. DOI:10.1111/j.1365-2109.2012.03193.x |
MA X C, ZHU F, JIN Q R, 2019. Antibiotics and chemical disease-control agents reduce innate disease resistance in crayfish[J]. Fish & Shellfish Immunology, 86: 169-178. |
PARK J H, SEOK S H, CHO S A, et al, 2004. Safety and protective effect of a disinfectant (STEL water) for white spot syndrome viral infection in shrimp[J]. Diseases of Aquatic Organisms, 60(3): 253-257. |
PEI R T, KIM S C, CARLSON K H, et al, 2006. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Research, 40(12): 2427-2435. DOI:10.1016/j.watres.2006.04.017 |
SAKAMI T, KOISO M, SUGAYA T, 2014. Characterization of bacterial community composition in rotifer cultures under unexpected growth suppression[J]. Fisheries Science, 80(4): 757-765. DOI:10.1007/s12562-014-0741-y |
SHAO S C, HU Y Y, CHENG J H, et al, 2018. Research progress on distribution, migration, transformation of antibiotics and antibiotic resistance genes (ARGs) in aquatic environment[J]. Critical Reviews in Biotechnology, 38(8): 1195-1208. DOI:10.1080/07388551.2018.1471038 |
SHI P, JIA S Y, ZHANG X X, et al, 2013. Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water[J]. Water Research, 47(1): 111-120. DOI:10.1016/j.watres.2012.09.046 |
SHI S J, NUCCIO E E, SHI Z J, et al, 2016. The interconnected rhizosphere: high network complexity dominates rhizosphere assemblages[J]. Ecology Letters, 19(8): 926-936. DOI:10.1111/ele.12630 |
SONG P, ZHOU B, FENG G, et al, 2019. The influence of chlorination timing and concentration on microbial communities in labyrinth channels: implications for biofilm removal[J]. Biofouling, 35(4): 401-415. DOI:10.1080/08927014.2019.1600191 |
SULLIVAN B A, VANCE C C, Gentry T J, et al, 2017. Effects of chlorination and ultraviolet light on environmental tetracycline resistant bacteria and tet (W) in water[J]. Journal of Environmental Chemical Engineering, 5(1): 777-784. DOI:10.1016/j.jece.2016.12.052 |
TANG Y W, ZHANG H J, YAN J J, et al, 2023. Assessing the efficacy of bleaching powder in disinfecting marine water: Insights from the rapid recovery of microbiomes[J]. Water Research, 241: 120136. DOI:10.1016/j.watres.2023.120136 |
WAN K, ZHENG S K, YE C S, et al, 2022. Ancient oriental wisdom still works: removing ARGs in drinking water by boiling as compared to chlorination[J]. Water Research, 209: 117902. DOI:10.1016/j.watres.2021.117902 |
WANG M N, ATEIA M, AWFA D, et al, 2021a. Regrowth of bacteria after light-based disinfection — What we know and where we go from here[J]. Chemosphere, 268: 128850. DOI:10.1016/j.chemosphere.2020.128850 |
WANG J L, CHU L B, WOJNÁROVITS L, et al, 2020. Occurrence and fate of antibiotics, antibiotic resistant genes (ARGs) and antibiotic resistant bacteria (ARB) in municipal wastewater treatment plant: an overview[J]. Science of the Total Environment, 744: 140997. DOI:10.1016/j.scitotenv.2020.140997 |
WANG L L, SU H C, HU X J, et al, 2019a. Abundance and removal of antibiotic resistance genes (ARGs) in the rearing environments of intensive shrimp aquaculture in South China[J]. Journal of Environmental Science and Health, Part B, 54(3): 211-218. DOI:10.1080/03601234.2018.1550310 |
WANG H B, WU Y H, LUO L W, et al, 2021b. Risks, characteristics, and control strategies of disinfection-residual-bacteria (DRB) from the perspective of microbial community structure[J]. Water Research, 204: 117606. DOI:10.1016/j.watres.2021.117606 |
WANG H, XU J J, TANG W, et al, 2019b. Removal efficacy of opportunistic pathogens and bacterial community dynamics in two drinking water treatment trains[J]. Small, 15(2): 1804436. DOI:10.1002/smll.201804436 |
YOON Y, CHUNG H J, DI D Y W, et al, 2017. Inactivation efficiency of plasmid-encoded antibiotic resistance genes during water treatment with chlorine, UV, and UV/H2O2[J]. Water Research, 123: 783-793. DOI:10.1016/j.watres.2017.06.056 |
YUAN Q B, GUO M T, YANG J, 2015. Fate of antibiotic resistant bacteria and genes during wastewater chlorination: implication for antibiotic resistance control[J]. PLoS One, 10(3): e0119403. DOI:10.1371/journal.pone.0119403 |
YUAN J L, NI M, LIU M, et al, 2019. Occurrence of antibiotics and antibiotic resistance genes in a typical estuary aquaculture region of Hangzhou Bay, China[J]. Marine Pollution Bulletin, 138: 376-384. DOI:10.1016/j.marpolbul.2018.11.037 |
ZHENG Y F, YU M, LIU Y, et al, 2016. Comparison of cultivable bacterial communities associated with Pacific white shrimp (Litopenaeus vannamei) larvae at different health statuses and growth stages[J]. Aquaculture, 451: 163-169. DOI:10.1016/j.aquaculture.2015.09.020 |
ZHU D, AN X L, CHEN Q L, et al, 2018. Antibiotics disturb the microbiome and increase the incidence of resistance genes in the gut of a common soil collembolan[J]. Environmental Science & Technology, 52(5): 3081-3090. |
 2024, Vol. 55
2024, Vol. 55


