中国海洋湖沼学会主办。
文章信息
- 梁日深, 何浩斌, 郜梓涵, 张景琪, 朱梓锋, 陈厚桦, 周萌, 黄燕华, 张凯. 2024.
- LIANG Ri-Shen, HE Hao-Bin, GAO Zi-Han, ZHANG Jing-Qi, ZHU Zi-Feng, CHEN Hou-Hua, ZHOU Meng, HUANG Yan-Hua, ZHANG Kai. 2024.
- 我国裸胸鳝属鱼类新记录种——奥迪萨裸胸鳝(Gymnothorax odishi)形态与线粒体基因组分析
- MORPHOLOGAL CHARACTERISTICS AND MITOCHONDRIAL GENOME OF GYMNOTHORAX ODISHI, A NEW-RECORD GYMNOTHORAX SPECIES OF CHINA
- 海洋与湖沼, 55(1): 279-291
- Oceanologia et Limnologia Sinica, 55(1): 279-291.
- http://dx.doi.org/10.11693/hyhz20230900181
文章历史
-
收稿日期:2023-09-02
收修改稿日期:2023-11-02
2. 仲恺农业工程学院动物科技学院 广东广州 510225;
3. 广东省水环境与水产品安全工程技术研究中心 广东广州 510225;
4. 深圳大学生命与海洋科学学院 广东深圳 518060;
5. 华南农业大学海洋学院 广东广州 510642
2. College of Animal Science and Technology, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China;
3. Guangdong Provincial Water Environment and Aquatic Products Security Engineering Technology Research Center, Guangzhou 510225, China;
4. The College of Life Sciences and Oceanography, Shenzhen University, Shenzhen 518060, China;
5. College of Marine Sciences, South China Agricultural University, Guangzhou 510642, China
裸胸鳝鱼类隶属于鳗鲡目(Anguilliformes), 海鳝科(Muraenidae), 海鳝亚科(Muraeninae), 裸胸鳝属(Gymnothorax) (成庆泰等, 1987), 广泛分布于全球热带、亚热带及温带海区, 主要栖息于珊瑚礁及岩礁区域, 部分物种生活在近海沙泥底质海域, 为凶猛肉食性鱼类(申屠垠等, 2009)。裸胸鳝在体表颜色和斑纹上呈现出明显的多样化, 从体表纯色无斑纹到各种鲜艳颜色与独特的斑纹、条带、网纹及其他不规则斑纹图案。目前全球裸胸鳝属记录约120多种(Smith, 2012; Smith et al, 2019; Froese et al, 2023), 是海鳝科中物种数量最多的一个属。我国分布有30余种(朱元鼎等, 1962, 1963; 成庆泰等, 1987; 黄宗国, 2008; 刘瑞玉, 2008; 张春光, 2010; 陈大刚等, 2016)。其中《中国海洋鱼类》(陈大刚等, 2016)与《中国海洋生物名录》(刘瑞玉, 2008)记录的裸胸鳝属种类最多, 均为37种。
由于裸胸鳝栖息环境较为隐秘, 并具凶猛攻击性, 故目前有关裸胸鳝鱼类的基础研究较少(Reece et al, 2010)。部分裸胸鳝的物种有效性、信息名录与种质资源分布还存空白, 有待进一步更新与完善(Böhlke et al, 2000; Smith, 2012; Tang et al, 2013; Huang et al, 2019)。近年来, 在印度-西太平洋部分国家和地区的海域陆续发现一些裸胸鳝的新种或新记录种, 如中国台湾澎湖列岛Gymnothorax poikilospilus (Huang et al, 2022)、越南坚江省Gymnothorax longinquus (Huang et al, 2020)、印度安达曼群岛Gymnothorax aurocephalus (Nashad et al, 2020)、印度喀拉拉邦Gymnothorax smithi (Sumod et al, 2019)、印度西孟加拉邦Gymnothorax pseudotile (Chakraborty et al, 2018)等。自2018年以来, 全球共有10个裸胸鳝新种或新记录种被发现, 揭示目前裸胸鳝属鱼类还存在许多未被开发的种质资源, 物种分类信息与地理分布数据有待进一步的更新和确认。国内近两年也有鞍头裸胸鳝(Gymnothorax sagmacephalus) (何浩斌等, 2022)、黏裸胸鳝(Gymnothorax mucifer) (张国庆等, 2022)等裸胸鳝新记录种鉴定的报道, 表明国内裸胸鳝种质资源研究同样不足, 存在较大的可挖掘潜力。
本研究于2021年11月在海南省陵水黎族自治县水产码头采集裸胸鳝样品一尾, 其主要特征为: 体型较短, 粗大, 体棕褐色, 眼睛后缘及鳃孔处有一明显黑斑, 鳃部至吻端区域分布橘黄色斑点, 头部之后整个躯干单一褐色, 无其他斑纹、条带。该物种在我国分类资料均无记载。形态特征与Mohapatra等2018年采集于东印度奥迪萨邦海岸的裸胸鳝新种——奥迪萨裸胸鳝Gymnothorax odishi sp. nov.特征描述较为一致, 初步鉴定为奥迪萨裸胸鳝G. odishi, 为我国裸胸鳝新记录种。研究针对奥迪萨裸胸鳝进行详细的形态特征测量, 并与Mohapatra采集的模式种进行比较。同时, 分子水平测定奥迪萨裸胸鳝线粒体全基因组, 针对奥迪萨裸胸鳝的分子分类地位深入探讨, 明确其在裸胸鳝属的分类地位, 验证其物种有效性, 为我国裸胸鳝属鱼类的系统分类以及物种名录修订提供分类基础。
1 材料与方法 1.1 实验材料奥迪萨裸胸鳝样品标本于2021年11月在海南省陵水黎族自治县水产码头采集, 共1尾, 全长864 mm。样品鲜活状态进行高清拍摄, 并取少量鳃丝样品于95%乙醇中保存, 用于基因组DNA提取。鱼体标本浸泡于福尔马林溶液, 保存于仲恺农业工程学院动物科技学院水产养殖系实验室, 编号为ZKAT-GO20211101。
1.2 实验方法 1.2.1 形态学特征研究分析采用传统形态学测量方法, 参考张春光(2010)、Mohapatra等(2018)测量方式, 对采集的奥迪萨裸胸鳝样品的形态特征进行描述。可量性状利用直尺及游标卡尺进行准确测量, 分度值精确到1 mm; 测量要素包括全长、体长、体高、肛前体长、肛后体长、头长、头高、吻长等; 可数性状采用直接计数法进行统计, 包括前颌齿、上颌齿、下颌齿、犁骨齿、前颌骨、中央齿及脊椎骨数等。
1.2.2 DNA提取与线粒体基因组测序取约20 mg鳃丝组织样品, 利用海洋动物组织基因组DNA提取试剂盒(天根生化), 按照说明书步骤提取样品DNA。提取的DNA用80 µL灭菌水溶解, 采用1%琼脂糖凝胶电泳检测、NanoDrop 2000分光光度计分析, 筛选出质量、浓度优良的DNA样品, 于–20 ℃冰箱保存备用。选取的样品送往广州天一辉远生物科技有限公司, 基于Illumina NovaSeq 6000测序平台, 运用第二代测序技术(Next generation Sequencing, NGS)对样品线粒体基因组进行高通量测序。
1.2.3 序列拼接组装与注释分析测序所得有效数据利用SPAdes 3.11.0 (Bankevich et al, 2012)软件进行组装。组装完成的线粒体基因组于在线注释工具MITOs (Bernt et al, 2013)网站(http://mitos.bioinf.unileipzig.de/)进行识别与注释, 利用CGView (Stothard et al, 2019) (https://cgview.ca)工具绘制线粒体基因组图谱。利用tRNAScan-SESearchServer (Lowe et al, 1997)软件对tRNA二级结构进行预测, 利用MEGA X (Kumar et al, 2018)软件分析裸胸鳝线粒体基因组碱基组成、遗传距离、基因的密码子偏好性等信息。
1.2.4 分子系统进化分析基于奥迪萨裸胸鳝线粒体基因组中12个蛋白编码基因序列(ND6除外), 结合GenBank上公布的24种海鳝科鱼类以及作为外类群的海鳗科(Muraenesocidae)海鳗(Muraenesox cinereus)线粒体基因组12个蛋白编码基因共同分析。此外, 为了验证奥迪萨裸胸鳝物种有效性及其在裸胸鳝属中的分类地位, 从GenBank下载全球分布的71种裸胸鳝属鱼类DNA条形码COI基因序列, 利用Clustal W (Thompson et al, 1994)软件进行序列比对, 生成统一的数据矩阵。利用MEGA X (Kumar et al, 2018)软件计算各物种遗传距离, 基于RAxML 8.0软件(Stamatakis, 2014)利用最大似然法(Maximum likelihood method)和邻接法(Neighbor-joining method)构建分子系统进化树, 利用jModelTest 2.1.5 (Posada, 2008)进行进化树最佳模型预测, 进化树各分支节点的置信度采用1 000次Bootstrap自举法验证。
2 实验结果 2.1 奥迪萨裸胸鳝形态特征 2.1.1 新种记录奥迪萨裸胸鳝标本样品ZKAT-GO20211101于2021年11月1日采集于海南省陵水黎族自治县水产码头, 全长864 mm。
 |
| 图 1 奥迪萨裸胸鳝外部形态特征图 Fig. 1 External morphological characteristics of G. odishi |
体棕褐色, 体型较粗大, 吻端至鳃孔部分布有橘黄色小斑点, 眼睛后缘存在比眼睛大的黑色斑块, 鳃孔边缘黑色, 背鳍始于鳃孔之前, 身体无其他明显斑纹或条带, 体纯色, 前颌齿、上颌齿、中央齿单行, 下颌齿两行, 椎骨式为4-52-135。
2.1.3 性状描述奥迪萨裸胸鳝形态测量数据见表 1。该裸胸鳝全长为864 mm, 全长为体高(99 mm)的8.73倍, 为背鳍前距(81.5 mm)的10.60倍, 为臀鳍前距(378.8 mm)的1.82倍, 为头长(121.4 mm)的7.12倍。头长是上颌长(59 mm)的2.06倍, 下颌长(68 mm)的1.79倍, 眼径(9.1 mm)的13.34倍, 眼间距(18.3 mm)的6.63倍。
| 测量性状 | ZKAT-GO20211101 | 模式标本 (Mohapatra et al, 2018) |
| 全长/mm | 864 | 420~700 |
| 全长占其他测量性状的倍数 | ||
| 体长 | 1.01 | — |
| 体高 | 8.73 | 11.5~18.4 |
| 头长 | 7.12 | 6.7~7.6 |
| 背鳍前距 | 10.60 | 8.4~10.8 |
| 臀鳍前距 | 1.82 | — |
| 背鳍基长 | 1.20 | — |
| 臀鳍基长 | 2.28 | — |
| 肛前体长 | 1.99 | 1.9~2.1 |
| 肛后体长 | 2.20 | — |
| 头长占其他测量性状的倍数 | ||
| 头高 | 1.41 | — |
| 吻长 | 4.88 | 4.3~5.4 |
| 上颌长 | 2.06 | — |
| 下颌长 | 1.79 | 2.2~2.8 |
| 眼径 | 13.34 | 8.6~10.8 |
| 眼间距 | 6.63 | 6.0~7.9 |
| 背鳍鳍条数/条 | 317 | — |
| 臀鳍鳍条数/条 | 198 | — |
| 背前脊椎骨数/个 | 4 | 4 |
| 肛门前脊椎骨数/个 | 52 | 55~58 |
| 总脊椎骨数/个 | 135 | 133~138 |
| 前颌齿/颗1 | 6 | 4~8 |
| 中央齿/颗 | 3 | 3 |
| 犁骨齿/颗 | — | 5~6 |
| 上颌齿外侧/颗1 | 13 | 12~14 |
| 上颌齿内侧/颗1 | 0 | 0 |
| 下颌齿外侧/颗1 | 25 | 20~24 |
| 下颌齿内侧/颗1 | 2 | 1 |
| 注: 测量性状中上标“1”表示牙齿单侧的数据, “—”表示无数据 | ||
背鳍鳍条数为317, 臀鳍鳍条数为198。锥体呈双凹型, 总脊椎数为135, 背前脊椎数为4, 肛门前脊椎数为52, 椎骨式4-52-135。牙齿呈锥状, 前颌齿一行, 每侧具6~7颗较大犬齿; 上颌齿一行, 牙齿较前颌齿稍小, 每侧13颗; 中央齿尖长弯曲, 单行, 3颗; 无犁骨齿; 下颌齿两行, 外侧每侧牙齿25颗, 内侧每侧2颗, 靠近吻端。
体棕褐色, 体型粗壮, 吻长较短, 前鼻孔呈长细管状, 位于上颌前端, 后鼻孔圆形, 位于眼前缘上方。吻端至鳃孔区域分布有零散橘黄色斑点, 眼睛后缘存在比眼睛大的明显黑斑, 鳃孔边缘也为黑色。背鳍始于鳃孔之前, 体色单一, 无其他斑纹或条带。背鳍、臀鳍均与尾鳍相连, 无胸鳍, 肛门位于体中央之后。
2.2 奥迪萨裸胸鳝与近缘裸胸鳝种类形态差异比较根据目前国内分类资料所收录的裸胸鳝属鱼类物种信息(成庆泰等, 1987; 张春光, 2010; 陈大刚等, 2016; Froese et al, 2023), 排除体色鲜艳, 身体具复杂斑点、条纹或条带的种类, 保留与奥迪萨裸胸鳝形态接近、体色偏纯色, 无复杂斑纹的种类比较分析, 结果如表 2所示。奥迪萨裸胸鳝可根据体型短粗、眼后存在黑斑、鳃孔黑色、头部分布橘黄色斑点等典型特征, 与其他裸胸鳝区分开来。
| 区分特征 | 体型 | 体表颜色 | 眼后有无黑斑 | 鳃孔是否黑色 | 头部有无橙色斑点 | 总椎骨数 |
| 奥迪萨裸胸鳝 G. odishi | 短体 | 棕褐色 | 有 | 是 | 有 | 133~138 |
| 鞍头裸胸鳝 G. sagmacephalus | 修长 | 浅褐色 | 有 | 无 | 无 | 170~176 |
| 海氏裸胸鳝 G. herrei | 短体 | 红褐色 | 无 | 无 | 无 | 114~116 |
| 白缘裸胸鳝 G. albimarginatus | 修长 | 紫褐色 | 无 | 无 | 无 | 171~181 |
| 长身裸胸鳝 G. prolatus | 修长 | 灰褐色 | 无 | 无 | 无 | 182~187 |
| 平达裸胸鳝 G. pindae | 短体 | 褐色 | 无 | 无 | 无 | 110~124 |
测序获得奥迪萨裸胸鳝线粒体基因组全长为16 580 bp (Genbank登录号: OR465051), 闭合双链(重链和轻链)环状结构。整个基因组由37个基因组成, 13个蛋白编码基因、22个tRNA基因、2个rRNA基因和1个D-Loop非编码控制区序列(见图 2, 表 3)。9个基因[tRNA-Gln、tRNA-Ala、tRNA-Asn、tRNA-Cys、tRNA-Tyr、tRNA-Ser (UCN)、tRNA-Glu、tRNA-Pro和ND6]位于轻链上, 其余基因均位于重链。基因排列顺序与大多数鱼类相似, 无重排现象。基因组4种碱基A、T、C、G含量分别为: 30.05%、26.86%、25.94%、17.15%, A+T (56.91%)大于G+C (43.09%), 表现为A+T偏好性和反G偏倚。
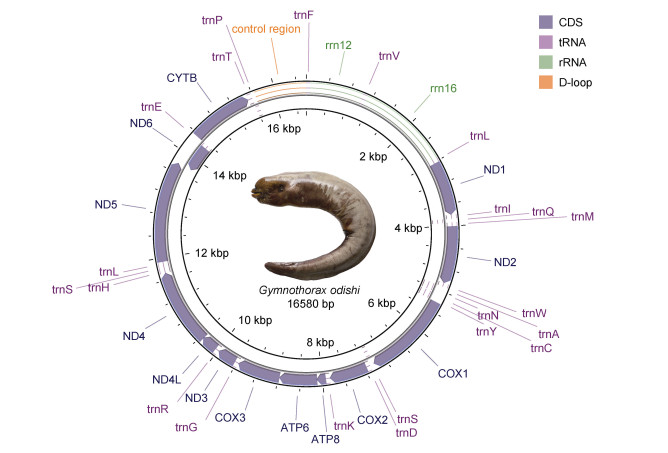 |
| 图 2 奥迪萨裸胸鳝线粒体基因组结构图 Fig. 2 Mitogenome map of G. odishi |
| 基因 | 编码链 | 位置 | 长度/bp | 间隔碱基 | 起始密码子 | 终止密码子 |
| tRNAPhe | H | 1~69 | 69 | 0 | ||
| 12S rRNA | H | 70~1 018 | 949 | 0 | ||
| tRNAVal | H | 1 019~1 089 | 71 | 0 | ||
| 16S rRNA | H | 1 090~2 748 | 1 659 | 0 | ||
| tRNALeu(UUR) | H | 2 749~2 824 | 76 | 0 | ||
| ND1 | H | 2 825~3 796 | 972 | 1 | ATG | TAA |
| tRNAIle | H | 3 798~3 868 | 71 | –1 | ||
| tRNAGln | L | 3 868~3 938 | 71 | –1 | ||
| tRNAMet | H | 3 938~4 006 | 69 | 0 | ||
| ND2 | H | 4 007~5 048 | 1 042 | 0 | ATG | T-- |
| tRNATrp | H | 5 049~5 117 | 69 | 1 | ||
| tRNAAla | L | 5 119~5 187 | 69 | 1 | ||
| tRNAAsn | L | 5 189~5 261 | 73 | 22 | ||
| tRNACys | L | 5 284~5 350 | 67 | 0 | ||
| tRNATyr | L | 5 351~5 421 | 71 | 1 | ||
| COI | H | 5 423~7 025 | 1 603 | 1 | GTG | T-- |
| tRNASer(UCN) | L | 7 027~7 097 | 71 | 5 | ||
| tRNAAsp | H | 7 103~7 172 | 70 | 0 | ||
| COII | H | 7 173~7 863 | 691 | 0 | ATG | T-- |
| tRNALys | H | 7 864~7 936 | 73 | 1 | ||
| ATPase8 | H | 7 938~8 105 | 168 | –10 | ATG | TAA |
| ATPase6 | H | 8 096~8 779 | 684 | –1 | ATG | TAA |
| COIII | H | 8 779~9 563 | 785 | 0 | ATG | TA- |
| tRNAGly | H | 9 564~9 634 | 71 | 0 | ||
| ND3 | H | 9 635~9 983 | 349 | 0 | ATG | T-- |
| tRNAArg | H | 9 984~10 053 | 70 | 0 | ||
| ND4L | H | 10 054~10 350 | 297 | –7 | ATG | TAA |
| ND4 | H | 10 344~11 721 | 1 378 | 0 | ATG | T-- |
| tRNAHis | H | 11 722~11 790 | 69 | 0 | ||
| tRNASer(AGY) | H | 11 791~11 858 | 68 | 0 | ||
| tRNALeu(CUN) | H | 11 859~11 931 | 73 | 0 | ||
| ND5 | H | 11 932~13 773 | 1 842 | –4 | ATG | TAA |
| ND6 | L | 13 770~14 288 | 519 | 0 | ATG | TAA |
| tRNAGlu | L | 14 289~14 357 | 69 | 6 | ||
| Cytb | H | 14 364~15 503 | 1 140 | 5 | ATG | TAA |
| tRNAThr | H | 15 509~15 580 | 72 | 1 | ||
| tRNAPro | L | 15 582~15 651 | 70 | 0 | ||
| D-loop | H | 15 652~16 580 | 929 | 0 |
奥迪萨裸胸鳝线粒体基因组存在一定的间隔与重叠(表 3)。其中间隔区出现11次, 间隔长度为1~22 bp, 总长度为45 bp, 最大间隔区在tRNA-Asn和tRNA-Cys之间。重叠区出现6次, 重叠长度为1~10 bp, 总长度24 bp, 最大重叠区在ATPase8与ATPase6之间。其他基因排列紧密, 无间隔与重叠情况。
2.3.2 蛋白编码基因分析奥迪萨裸胸鳝线粒体基因组13个蛋白编码基因总长度为11 470 bp, 占线粒体全长的69.18%。除COI基因利用GTG作为起始密码子, 其他蛋白编码基因起始密码子均为ATG; 终止密码子类型在不同基因中存在多样化, 除了ATPase8、ATPase6、ND1、ND4L、ND5、ND6和Cyt b基因使用TAA外, 其他基因均为不完全终止密码子, 如COIII基因使用TA, ND2、COI、COII、ND3和ND4基因使用T作为终止密码子。终止密码子的缺失一般认为是由多聚腺苷酸化引起。而利用TA或T这两种不完全终止密码子, 在其他鱼类线粒体基因组也是常见的现象。
计算奥迪萨裸胸鳝蛋白编码基因相对密码子使用频率(relative synonymous codon usage, RSCU)如图 3所示。结果表明, 奥迪萨裸胸鳝13个蛋白编码基因中存在31个偏好密码子(RSCU > 1)。其中, 使用频率最高的密码子是CGA (RSCU=2.40), 最低为GCG (RSCU=0.19)。各密码子的RSCU值相差较大, 表明密码子使用频率具有一定的偏向性。在密码子第三位中, A的使用频率最高(1 356个), AT使用频率均高于GC。
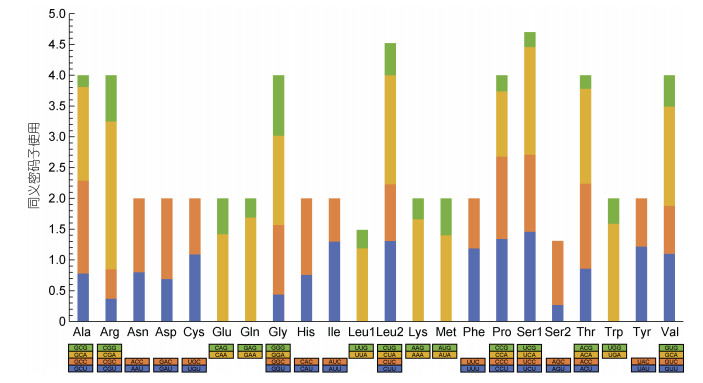 |
| 图 3 奥迪萨裸胸鳝蛋白编码基因同义密码子使用情况 Fig. 3 Relative synonymous codon usage (RSCU) of the mitogenome of G. odishi |
在氨基酸组成方面, 总长度为11 470 bp的蛋白编码序列中, 除去终止密码子, 共编码氨基酸3 814个, 其中亮氨酸(Leu)含量最高, 为15.86%, 其次是丙氨酸(Ala), 含量为8.76%, 而半胱氨酸(Cys)含量最小, 仅为0.87% (图 4)。
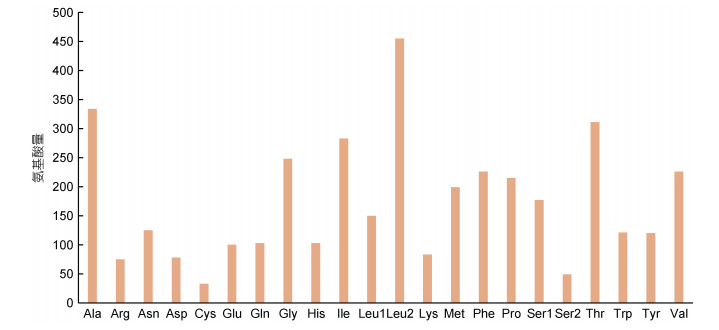 |
| 图 4 奥迪萨裸胸鳝蛋白编码基因氨基酸数 Fig. 4 Number of amino acids in protein-coding genes of G. odishi |
奥迪萨裸胸鳝rRNA基因由12S rRNA和16S rRNA组成。12S rRNA基因长度为949 bp, 16S rRNA为1 646 bp, 两者位于tRNA-Phe和tRNA-Leu (UUR)之间, 中间由tRNA-Val基因隔开。22个tRNA基因长度在67 bp (tRNA-Cys)至76 bp (tRNA-Leu)之间, 总长度为1 552 bp。其中8个tRNA基因位于轻链, 14个位于重链。利用tRNAscan-SE软件进行tRNA二级结构进行预测, 除tRNA-Ser (AGY)缺少一个DHU臂, 其余21个tRNA基因均能形成典型的三叶草形结构。
2.4 海鳝科分子系统进化分析 2.4.1 海鳝科蛋白编码基因序列分子系统进化关系基于线粒体基因组12个蛋白编码基因序列(ND6基因除外), 以海鳗科海鳗作为外类群, 利用最大似然法(ML)及邻接法(NJ)将奥迪萨裸胸鳝与Genbank下载的24种海鳝科鱼类序列一起构建分子系统进化树。构建ML树之前, 使用Modeltest软件预测最佳核苷酸替代模型为GTR+G+I。两种方法构建的进化树拓扑结构基本一致, 将两种进化树合并, 树上节点分别为ML法与NJ法的bootstrap支持率(图 5)。进化树上, 25种海鳝科鱼类主要形成海鳝亚科与鳍尾鳝亚科两个独立分支, 海鳝亚科包括裸胸鳝属(Gymnothorax)、蛇鳝属(Echidna)、勾吻鳝属(Enchelycore)、裸海鳝属(Gymnomuraena)、管鼻海鳝属(Rhinomuraena)和弯牙海鳝属(Strophidon), 鳍尾鳝亚科(Uropterygiinae)包含鞭尾鳝属(Scuticaria)和高眉鳝属(Anarchias), 与形态学划分一致。在海鳝亚科分支中, 物种数量最多的裸胸鳝属并未形成单系, 除了裸海鳝属单独一支位于海鳝亚科分支基部, 蛇鳝属、勾吻鳝属、管鼻鳝属与弯牙海鳝属的种类均分散分布于裸胸鳝属内部。奥迪萨裸胸鳝进化地位主要位于小裸胸鳝、网纹裸胸鳝等6种海鳝鱼类组成的分支中, 并以100%的支持率与匀斑裸胸鳝和淡网纹裸胸鳝形成的分支聚在一起, 揭示其与匀斑裸胸鳝/淡网纹裸胸鳝的关系最近。
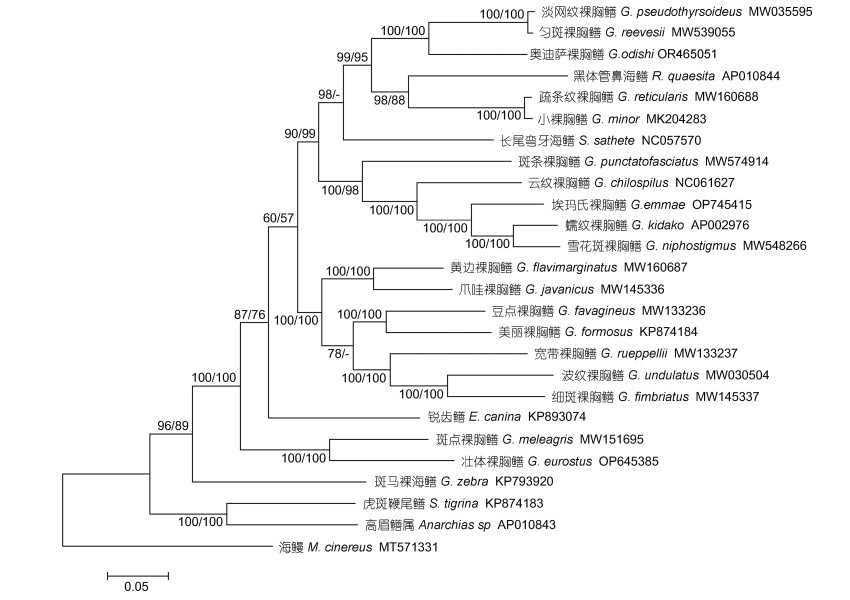 |
| 图 5 基于12个蛋白编码基因序列利用最大似然法和邻接法构建的25种海鳝科的分子系统进化树 Fig. 5 Molecular phylogenetic trees of 26 Muraenidae species constructed using the maximum likelihood and neighbor-joining methods based on 12 protein-coding genes |
为了从分子水平分析奥迪萨裸胸鳝物种有效性及其在裸胸鳝属鱼类的分类地位, 选择鱼类DNA条形码COI基因进行分析。从奥迪萨裸胸鳝线粒体基因组中获取COI基因序列, 结合GenBank下载的71种裸胸鳝属鱼类COI基因序列共同比对分析。利用MEGA X软件, 基于Kimura-2-parameter模型计算奥迪萨裸胸鳝与其他裸胸鳝的种间遗传距离如表 4所示。奥迪萨裸胸鳝与其他裸胸鳝的COI遗传距离范围为0.082~0.263, 其中与肝色裸胸鳝的遗传距离最小(0.082); 与细尾裸胸鳝遗传距离最大(0.263)。所有遗传距离数值均远大于2% (0.020)的最小物种区分遗传距离, 揭示奥迪萨裸胸鳝是区分于其他裸胸鳝的独立物种。为进一步分析奥迪萨裸胸鳝在裸胸鳝属的分类地位, 根据遗传距离数据, 从中选择与奥迪萨裸胸鳝亲缘关系较近的20种裸胸鳝(根据COI遗传距离从小到大的顺序), 以大头尾鳝和虎斑鞭尾鳝为外群, 采用最大似然法(ML)和邻接法(NJ)法进行分子系统进化树构建(图 6)。结果显示, 两棵进化树上, 奥迪萨裸胸鳝与9种裸胸鳝(肝色裸胸鳝、黄纹裸胸鳝、匀斑裸胸鳝、异纹裸胸鳝、爪哇裸胸鳝等)紧密聚为一支。在该分支中, 奥迪萨裸胸鳝先以高支持率与肝色裸胸鳝紧密聚成姐妹分支, 亲缘关系最近; 该分支再与黄纹裸胸鳝聚在一起, 最后再与淡网纹裸胸鳝、匀斑裸胸鳝形成的分支相聚。
| 物种 | 距离 | 物种 | 距离 | 物种 | 距离 | 物种 | 距离 | |||
| G. hepaticus | 0.082 | G. javanicus | 0.190 | G. neglectus | 0.207 | G. margaritophorus | 0.226 | |||
| G. monochrous | 0.103 | G. kolpos | 0.191 | G. maderensis | 0.207 | G. emmae | 0.226 | |||
| G. shaoi | 0.133 | G. enigmaticus | 0.192 | G. nubilus | 0.207 | G. griseus | 0.229 | |||
| G. pseudothyrsoideus | 0.142 | G. miliaris | 0.193 | G. zonipectis | 0.207 | G. thyrsoideus | 0.230 | |||
| G. reevesii | 0.142 | G. reticularis | 0.193 | G. elegans | 0.207 | G. moringa | 0.231 | |||
| G. flavimarginatus | 0.166 | G. rueppelliae | 0.195 | G. porphyreus | 0.209 | G. robinsi | 0.231 | |||
| G. phalarus | 0.166 | G. longinquus | 0.195 | G. mucifer | 0.210 | G. nudivomer | 0.234 | |||
| G. tile | 0.167 | G. meleagris | 0.196 | G. obesus | 0.212 | G. pseudoherrei | 0.236 | |||
| G. albimarginatus | 0.175 | G. chilospilus | 0.199 | G. prasinus | 0.212 | G. niphostigmus | 0.236 | |||
| G. angusticauda | 0.178 | G. minor | 0.199 | G. buroensis | 0.214 | G. polygonius | 0.237 | |||
| G. equatorialis | 0.178 | G. nigromarginatus | 0.200 | G. fimbriatus | 0.219 | G. johnsoni | 0.239 | |||
| G. richardssonii | 0.180 | G. mordax | 0.200 | G. australicola | 0.221 | G. kidako | 0.240 | |||
| G. saxicola | 0.184 | G. breedeni | 0.201 | G. pictus | 0.221 | G. undulatus | 0.243 | |||
| G. ocellatus | 0.185 | G. vicinus | 0.203 | G. melatremus | 0.221 | G. taiwanensia | 0.248 | |||
| G. pindae | 0.187 | G. isingteena | 0.204 | G. pharaonis | 0.223 | G. fuscomaculatus | 0.253 | |||
| G. prolatus | 0.187 | G. favagineus | 0.204 | G. castlei | 0.223 | G. punctatus | 0.256 | |||
| G. randalli | 0.188 | G. formosus | 0.206 | G. eurostus | 0.224 | G. gracilicauda | 0.263 | |||
| G. sagmacephalus | 0.189 | G. funebris | 0.206 | G. polyuranodon | 0.225 |
 |
| 图 6 基于COI基因序列利用最大似然法和邻接法构建的裸胸鳝属鱼类分子系统进化树 Fig. 6 Molecular phylogenetic trees of Gymnothorax species constructed using the maximum likelihood and neighbor-joining methodd based on COI genes |
奥迪萨裸胸鳝最早于2018年Mohapatra等(2018)在东印度孟加拉湾作为新种发现, 至今世界其他地区均无记录。目前, 有关奥迪萨裸胸鳝的研究资料, 除了Mohapatra等(2018)的新种记录文章之外, 暂无其他数据信息, 世界鱼类数据库fishbase (Froese et al, 2023)也未有该裸胸鳝收录的资料。我国形态学分类书籍与文献中, 同样无奥迪萨裸胸鳝的物种记录信息。本研究在中国海南省陵水黎族自治县水产码头采集奥迪萨裸胸鳝的样品一尾, 形态学分析结果与Mohapatra等(2018)鉴定的奥迪萨裸胸鳝的特征及图片信息基本吻合, 初步鉴定为奥迪萨裸胸鳝, 为中国裸胸鳝属新记录种, 也是继Mohapatra之后第二次记录该裸胸鳝的信息。研究结果将奥迪萨裸胸鳝的分布范围从孟加拉湾扩展到中国南海, 为我国裸胸鳝属鱼类物种名录更新与完善提供依据, 充实我国裸胸鳝鱼类物种数据信息。
形态上, 裸胸鳝鱼类体色斑纹存在明显的物种差异多样化, 从体色单一无斑纹到多种复杂体色与斑纹特征。在体色单一纯色的裸胸鳝种类中, Böhlke (2000)曾根据脊椎骨数的多少将其划分为短体型及长体型两大类型, 短体型裸胸鳝脊柱骨数少于150, 而长体型类群脊椎骨数大于150。Mohapatra等(2018)根据奥迪萨裸胸鳝脊椎骨数为133–138, 将其归为短体型类型。学者Böhlke曾将全球裸胸鳝进行形态学归类, 汇总出属于短体型裸胸鳝的有8种(Böhlke, 1997; Böhlke et al, 2000), 分别为Gymnothorax herrei、Gymnothorax kontodontos、Gymnothorax panamensi、Gymnothorax atolli、Gymnothorax australicola、Gymnothorax microstictus、Gymnothorax pinda以及Gymnothorax pseudoherrei。近两年有学者在印度孟加拉湾另外发现了包括奥迪萨裸胸鳝在内的短体型裸胸鳝新种3个Gymnothorax mishrai sp. nov (Ray et al, 2015)、Gymnothorax odishi sp. nov (Mohapatra et al, 2018)以及Gymnothorax andamanensis sp. nov (Mohapatra et al, 2019)。而我国有记录的短体型裸胸鳝仅有G. herrei以及G. pindae两种。奥迪萨裸胸鳝与上述其他短体型裸胸鳝相比, 除了G. panamensis之外, 其他裸胸鳝均不具有眼睛后缘存在大黑斑, 吻端至鳃孔部分布有橘黄色小斑点的特征。G. panamensis眼后虽具明显黑斑, 但它的背鳍起点开始于鳃孔之后, 而奥迪萨裸胸鳝背鳍起点始于鳃孔之前, 可有效区分开来。
3.2 奥迪萨裸胸鳝的线粒体基因组结构分析奥迪萨裸胸鳝线粒体基因组全序列, 全长为16 580 bp, 符合目前研究表明鳗鲡目鱼类线粒体基因组大小为15~22 kb的长度范围(15~22 kb) (申欣等, 2014)。基因组结构由13个蛋白编码基因、2个rRNA基因、22个tRNA基因和1个非编码控制区(D-loop区)组成, 与其他鳗鲡目鱼类以及大部分硬骨鱼类结构一致(Teng et al, 2009; Zhong et al, 2018; 王开杰等, 2022), 在13个蛋白编码基因中, 除了ATPase8、ATPase6、ND1、ND4L、ND5、ND6和Cyt b基因利用TAA外、其他基因均以TA或T作为不完全终止密码子, 这种不完全终止密码子在其他动物线粒体基因组中也普遍存在(Peng et al, 2006)。有研究表明, 不完全终止密码子可在多聚腺苷酸化和多核转录切割作用形成完整的功能性终止密码子(Ojala et al, 1981; Du et al, 2016)。奥迪萨裸胸鳝线粒体基因组A+T含量(56.91%), C+G含量(43.09%), 核苷酸组成具有明显的AT偏向性, 与其他鳗鲡目及大多数硬骨鱼类碱基组成相似(申欣等, 2014; 毛明光等, 2019; 刘凯等, 2020), 这种碱基分布的不平衡性主要是在种群演化过程中出现了自然突变以及选择压力而导致的结果(钟东等, 2002)。值得注意的是, 本研究的奥迪萨裸胸鳝线粒体基因组未发现基因结构的重排, 这与部分前期研究鳗鲡目中蛇鳗科Ophichthyidae、海鳗科Muraenesocidae、康吉鳗科Congridae等多个科鱼类线粒体基因组存在的各种基因重排现象不同(申欣等, 2014; Lü et al, 2019; Zhang et al, 2021; 宁子君等, 2022)。将奥迪萨裸胸鳝线粒体基因组与GenBank中已公布的海鳝科线粒体基因组结构分析发现, 所有海鳝科鱼类线粒体基因组均无基因重排现象, 揭示鳗鲡目mtDNA中普遍的基因重排现象并不包括海鳝科鱼类。
3.3 奥迪萨裸胸鳝分子系统分类地位基于线粒体基因组12个蛋白编码基因与COI基因构建的分子系统进化树中, 奥迪萨裸胸鳝与匀斑裸胸鳝、淡网纹裸胸鳝、肝色裸胸鳝、黄纹裸胸鳝等裸胸鳝种类的亲缘关系最为密切。在12个蛋白编码基因构建的海鳝科鱼类进化树上, 奥迪萨裸胸鳝以100%的支持率与匀斑裸胸鳝和淡网纹裸胸鳝形成的分支聚在一起。而基于COI基因构建的裸胸鳝属进化树上, 奥迪萨裸胸鳝先以高支持率与肝色裸胸鳝聚为一支, 该分支再与黄纹裸胸鳝、匀斑裸胸鳝和淡网纹裸胸鳝形成的分支聚在一起。在COI遗传距离上, 奥迪萨裸胸鳝与其他71种裸胸鳝属鱼类遗传距离在0.082~0.263之间, 均大于Hebert等(2003)提出的最小物种鉴定遗传距离0.020 (2%), 从而在分子水平上揭示奥迪萨裸胸鳝是区分于目前其他裸胸鳝鱼类的独立物种。
3.4 海鳝科鱼类分子系统进化关系在构建的海鳝科鱼类分子进化树中, 进化树将25种海鳝划分为海鳝亚科Muraeninae (裸胸鳝属+蛇鳝属+勾吻鳝属+裸海鳝属+管鼻鳝属+弯牙海鳝属)与鳍尾鳝亚科Uropterygiinae (鞭尾鳝属+高眉鳝属)两个分支, 与形态学划分结果一致。在海鳝亚科分支中, 我们发现物种数量最多的裸胸鳝属鱼类未能形成单系。除了裸海鳝属单独形成一支, 独立于裸胸鳝属类群之外, 蛇鳝属、勾吻鳝属、管鼻鳝属与弯牙海鳝属等其他属的种类均位于裸胸鳝属内部, 并将裸胸鳝属分割成多个分支。对于裸胸鳝属的非单系性, 前期研究也有报道, Reece等(2010)、Tang等(2013)、陈铭等(2022)利用不同的分子标记构建海鳝科分类关系, 结果显示, 除了裸海鳝属, 海鳝亚科其他属的种类均聚在裸胸鳝属内部, 揭示裸胸鳝属可能为多系起源。Reece等(2010)曾提出, 要体现裸胸鳝属的单系性, 需要将其他属的种类归为裸胸鳝属。本研究基于12个蛋白编码基因序列得出的分类结果, 进一步验证裸胸鳝属的非单系性。而对于不同属的种类在裸胸鳝属中具体的位置, 需要后续更多的海鳝科线粒体全基因组全面综合分析才能最终体现。
4 结论本研究采集分析了我国裸胸鳝属鱼类新记录种——奥迪萨裸胸鳝, 此前该物种仅在东印度孟加拉湾有记录, 其主要特征为: 体型粗壮, 体棕褐色, 吻端至鳃孔区域分布橘黄色斑点, 眼睛后缘存在黑色斑块, 鳃孔边缘黑色, 身体无其他条带或斑纹。在线粒体基因组方面, 奥迪萨裸胸鳝线粒体全长为16 580 bp, 由38个基因组成, 基因组结构特征与其他海鳝科鱼类相似。分子系统进化关系表明, 奥迪萨裸胸鳝与匀斑裸胸鳝、淡网纹裸胸鳝、肝色裸胸鳝与黄纹裸胸鳝的关系较为密切。COI遗传距离揭示奥迪萨裸胸鳝在分子水平上也是区分于世界上其他裸胸鳝的独立物种。研究结果为我国裸胸鳝属鱼类的系统分类及物种名录更新提供了有效的形态与分子水平分类依据。
王开杰, 姜燕, 徐永江, 等, 2022. 黄带拟鲹线粒体基因组测序及鲹科鱼类系统发育分析[J]. 水产学报, 46(11): 2017-2027. |
毛明光, 顾杰, 刘瑞婷, 等, 2019. 太平洋鳕线粒体全基因组测序及结构特征分析[J]. 水生生物学报, 43(1): 17-26. |
申欣, 田美, 孟学平, 等, 2014. 鳗鲡目鱼类线粒体蛋白质编码基因易位及系统演化关系分析[J]. 海洋学报, 36(4): 73-81. DOI:10.3969/j.issn.0253-4193.2014.04.009 |
申屠垠, 黄海, 尹绍武, 等, 2009. 裸胸鳝属鱼类的研究现状与开发利用[J]. 现代渔业信息, 24(7): 18-21. DOI:10.3969/j.issn.1004-8340.2009.07.005 |
宁子君, 刘玉萍, 张书飞, 等, 2022. 艾氏蛇鳗线粒体基因组全序列结构分析和系统发育关系探讨[J]. 中国水产科学, 29(9): 1264-1276. |
成庆泰, 郑葆珊, 1987. 中国鱼类系统检索[M]. 北京: 科学出版社.
|
朱元鼎, 张春霖, 成庆泰, 1962. 南海鱼类志[M]. 北京: 科学出版社.
|
朱元鼎, 张春霖, 成庆泰, 1963. 东海鱼类志[M]. 北京: 科学出版社.
|
刘凯, 冯晓宇, 马恒甲, 等, 2020. 钱塘江三角鲂线粒体基因组测序及其结构特征分析[J]. 浙江农业学报, 32(9): 1591-1608. DOI:10.3969/j.issn.1004-1524.2020.09.08 |
刘瑞玉, 2008. 中国海洋生物名录[M]. 北京: 科学出版社.
|
何浩斌, 杨杰銮, 杨森, 等, 2022. 我国大陆裸胸鳝属鱼类新记录种——鞍头裸胸鳝的形态与分子系统学[J]. 水产学报: 1-11. [2022-08-09]. https://kns.cnki.net/kcms2/article/abstract?v=rNedIcCUbLBHw6itYc12oIYsd5660-_MXATinHYH_4sdltdT5bpXdYu73pAyRMnt86PN5m6RIz2fYLKkZw0atOtAOvr5V5H_FHgVwDfrzYV9iOltjkIIUw==&uniplatform=NZKPT&language=gb.
|
张国庆, 杨杰銮, 李培源, 等, 2022. 我国近海裸胸鳝属鱼类新记录种——黏裸胸鳝(Gymnothorax mucifer)形态与分子系统学研究[J]. 海洋学报, 44(7): 112-121. |
张春光, 2010. 中国动物志-硬骨鱼纲, 鳗鲡目 背棘鱼目[M]. 北京: 科学出版社.
|
陈大刚, 张美昭, 2016. 中国海洋鱼类[M]. 青岛: 中国海洋大学出版社.
|
陈铭, 范蔓桦, 谢瑞琳, 等, 2022. 基于16S rRNA序列探讨我国海鳝科鱼类分子系统进化关系[J]. 水产学报, 46(2): 183-195. |
钟东, 赵贵军, 张振书, 等, 2002. 基因组内碱基分布整体均衡与局部不均衡的研究进展[J]. 遗传, 24(3): 351-355. |
黄宗国, 2008. 中国海洋生物种类与分布[M]. 北京: 海洋出版社.
|
BANKEVICH A, NURK S, ANTIPOV D, et al, 2012. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing[J]. Journal of Computational Biology, 19(5): 455-477. DOI:10.1089/cmb.2012.0021 |
BERNT M, DONATH A, JÜHLING F, et al, 2013. MITOS: improved de novo metazoan mitochondrial genome annotation[J]. Molecular Phylogenetics and Evolution, 69(2): 313-319. DOI:10.1016/j.ympev.2012.08.023 |
BÖHLKE E B, 1997. Notes on the identity of elongate unpatterned indo-pacific morays, with description of a new species (muraenidae, subfamily muraninae)[J]. Proceedings of the Academy of Natural Sciences of Philadelphia, 147: 89-109. |
BÖHLKE E B, 2000. Notes on the identity of small, brown, unpatterned indo-pacific moray eels, with descriptions of three new species (anguilliformes: muraenidae)[J]. PacificSciencs, 54(4): 395-416. |
BÖHLKE E B, RANDALL J E, 2000. A review of the moray eels (Angulliformes: Muraenidae) of the Hawaiian Islands, with descriptions of two new species[J]. Proceedings of the Academy of Natural Sciences of Philadelphia, 150: 203-278. |
CHAKRABORTY P, SAREN S C, SENGUPTA A, et al, 2018. Notes on the Record of Gymnothorax pseudotile Mohapatra et al., 2018 (Muraenidae: Muraeninae) from the Sundarbans, West Bengal, India[J]. Records of the Zoological Survey of India, 118(3): 318-321. |
DU C, HE S L, SONG X H, et al, 2016. The complete mitochondrial genome of Epicauta chinensis (Coleoptera: Meloidae) and phylogenetic analysis among coleopteran insects[J]. Gene, 578(2): 274-280. DOI:10.1016/j.gene.2015.12.036 |
FROESE R, PAULY D, 2023. FishBase. World Wide Web Electronic Publication [M/OL]. 2023-06. www.Fishbase.org.
|
HEBERT P D N, CYWINSKA A, BALL S L, et al, 2003. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society B: Biological Sciences, 270(1512): 313-321. DOI:10.1098/rspb.2002.2218 |
HUANG W C, CHEN H M, LIAO T Y, 2019. Revalidation of a moray eel, Gymnothorax mucifer Snyder, 1904 (Teleostei: Anguilliformes: Muraenidae), with a revised distribution[J]. Zootaxa, 4559(1): 151-165. |
HUANG W C, LOH K H, CHEN H M, 2022. Gymnothorax poikilospilus, a new moray eel (Teleostei: Anguilliformes: Muraenidae) from Penghu Islands, western Taiwan[J]. Zootaxa, 5189(1): 87-102. DOI:10.11646/zootaxa.5189.1.11 |
HUANG W C, THU P T, LIAO T Y, 2020. A new record of the long moray, Gymnothorax longinquus (Actinopterygii: Anguilliformes: Muraenidae), from southern Vietnam, supporting the uncertain record in the Gulf of Thailand[J]. Acta Ichthyologica et Piscatoria, 50(2): 201-207. DOI:10.3750/AIEP/02790 |
KUMAR S, STECHER G, LI M, et al, 2018. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 35(6): 1547-1549. DOI:10.1093/molbev/msy096 |
LOWE T M, EDDY S R, 1997. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Research, 25(5): 955-964. DOI:10.1093/nar/25.5.955 |
LÜ Z M, ZHU K H, JIANG H, et al, 2019. Complete mitochondrial genome of Ophichthus brevicaudatus reveals novel gene order and phylogenetic relationships of Anguilliformes[J]. International Journal of Biological Macromolecules, 135: 609-618. DOI:10.1016/j.ijbiomac.2019.05.139 |
MOHAPATRA A, KIRUBA-SANKAR R, PRAVEENRAJ J, et al, 2019. A new short brown unpatterned moray eel Gymnothorax andamanensis (Muraenidae: Muraeninae) from Andaman waters, India[J]. Zootaxa, 4661(1): 189-196. |
MOHAPATRA A, MOHANTY S R, et al, 2018. Gymnothorax odishi sp. nov. (Muraenidae: Muraeninae), a short brown unpatterned moray eel from Bay of Bengal, India[J]. Zootaxa, 4420(1): 123-130. |
NASHAD M, MOHAPATRA A, VARGHESE S P, et al, 2020. A new white-spotted moray eel, Gymnothorax aurocephalus sp. nov. (Muraenidae: Muraeninae) from Andaman Sea, India[J]. Zootaxa, 4877(2): 361-372. |
OJALA D, MONTOYA J, ATTARDI G, 1981. tRNA punctuation model of RNA processing in human mitochondria[J]. Nature, 290(5806): 470-474. DOI:10.1038/290470a0 |
PENG Z G, WANG J, HE S P, 2006. The complete mitochondrial genome of the helmet catfish Cranoglanis bouderius (Siluriformes: Cranoglanididae) and the phylogeny of otophysan fishes[J]. Gene, 376(2): 290-297. DOI:10.1016/j.gene.2006.04.014 |
POSADA D, 2008. jModelTest: phylogenetic model averaging[J]. Molecular Biology and Evolution, 25(7): 1253-1256. DOI:10.1093/molbev/msn083 |
RAY D, MOHAPATRA A, SMITH D G, 2015. A new species of short brown unpatterned moray eel of the genus Gymnothorax (Anguilliformes: Muraenidae) from the Bay of Bengal[J]. Zootaxa, 4027(1): 140-144. DOI:10.11646/zootaxa.4027.1.8 |
REECE J S, BOWEN B W, SMITH D G, et al, 2010. Molecular phylogenetics of moray eels (Muraenidae) demonstrates multiple origins of a shell-crushing jaw (Gymnomuraena, Echidna) and multiple colonizations of the Atlantic Ocean[J]. Molecular Phylogenetics and Evolution, 57(2): 829-835. DOI:10.1016/j.ympev.2010.07.013 |
SMITH D G, 2012. A checklist of the moray eels of the world (Teleostei: Anguilliformes: Muraenidae)[J]. Zootaxa, 3474(1): 1-64. DOI:10.11646/zootaxa.3474.1.1 |
SMITH D G, BOGORODSKY S V, MAL A O, et al, 2019. Review of the moray eels (Anguilliformes: Muraenidae) of the Red Sea, with description of a new species[J]. Zootaxa, 4704(1): 1-87. |
STAMATAKIS A, 2014. RAxML version 8: A tool for phylogenetic analysis and post-analysis of large phylogenies[J]. Bioinformatics, 30(9): 1312-1313. DOI:10.1093/bioinformatics/btu033 |
STOTHARD P, GRANT J R, VAN DOMSELAAR G, 2019. Visualizing and comparing circular genomes using the CGView family of tools[J]. Briefings in Bioinformatics, 20(4): 1576-1582. DOI:10.1093/bib/bbx081 |
SUMOD K S, MOHAPATRA A, SANJEEVAN V N, et al, 2019. A new species of white-spotted moray eel, Gymnothorax smithi (Muraenidae: Muraeninae) from deep waters of Arabian Sea, India[J]. Zootaxa, 4652(2): 359-366. |
TANG K L, FIELITZ C, 2013. Phylogeny of moray eels (Anguilliformes: Muraenidae), with a revised classification of true eels (Teleostei: Elopomorpha: Anguilliformes)[J]. Mitochondrial DNA, 24(1): 55-66. |
TENG H Y, LIN Y S, TZENG C S, 2009. A new Anguilla species and a reanalysis of the phylogeny of freshwater eels[J]. Zoological Studies, 48(6): 808-822. |
THOMPSON J D, HIGGINS D G, GIBSON T J, 1994. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Research, 22(22): 4673-4680. |
ZHANG K, ZHU K H, LIU Y F, et al, 2021. Novel gene rearrangement in the mitochondrial genome of Muraenesox cinereus and the phylogenetic relationship of Anguilliformes[J]. Scientific Reports, 11(1): 2411. |
ZHONG L Q, WANG M H, LI D M, et al, 2018. Complete mitochondrial genome of freshwater goby Rhinogobius cliffordpopei (Perciformes, Gobiidae): genome characterization and phylogenetic analysis[J]. Genes & Genomics, 40(11): 1137-1148. |
 2024, Vol. 55
2024, Vol. 55


