中国海洋湖沼学会主办。
文章信息
- 邵帅, 陶玉林, 颜光祥, 汤昌盛, 廖智, 严小军, 何建瑜. 2024.
- SHAO Shuai, TAO Yu-Lin, YAN Guang-Xiang, TANG Chang-Sheng, LIAO Zhi, YAN Xiao-Jun, HE Jian-Yu. 2024.
- 厚壳贻贝(Mytilus coruscus)组织及其海水环境中真菌群落结构差异分析
- DIVERSITY OF FUNGAL COMMUNITY IN TISSUES OF MYTILUS CORUSCUS AND ITS CULTIVATION SEAWATER
- 海洋与湖沼, 55(2): 451-461
- Oceanologia et Limnologia Sinica, 55(2): 451-461.
- http://dx.doi.org/10.11693/hyhz20231100252
文章历史
-
收稿日期:2023-11-22
收修改稿日期:2024-01-15
海洋真菌分布广泛, 物种多样性高, 全球范围内已发现和报道的超过10 000种, 作为维持海洋生态系统平衡的重要组成部分, 在生态学过程中起着重要作用。作为分解者, 海洋真菌参与海洋动植物尸体的分解, 提高海洋初级生产力(Hyde et al, 1998); 参与包括碳、氮等元素在内的生物地球化学循环(Hyde et al, 1998; Wegley et al, 2007; Baltar et al, 2021; Orsi et al, 2022); 还可与海洋动植物形成共生或寄生关系, 在宿主体表和内部组织甚至细胞内(Zhang et al, 2009) 参与宿主的生理过程, 影响宿主的生长过程和生物量(Gutiérrez et al, 2016), 或参与宿主摄食和代谢(Phillips, 1984; Gatesoupe, 1999; Bärlocher et al, 2004), 提高宿主免疫能力(Kiran et al, 2018), 或通过侵染宿主成为致病菌(Franchini et al, 2005; Van Dover et al, 2007; Hatai, 2012; Kovačić et al, 2018; Carella et al, 2023)。因此, 研究宿主真菌群落对深入了解真菌的生态学作用具有重要的意义。
具高滤食性的海洋双壳纲贝类与周围水体微生物关系密切且作用复杂。贝类分布生境多样, 潮间带(Yahya et al, 2020), 深海热液(谢林青等, 2022), 冷泉(Lin et al, 2021)等均有分布, 在海洋生态系统中占据重要的生态位(Venier et al, 2011), 参与诸多重要的生态过程(Pruzzo et al, 2005; Telesca et al, 2021), 还可作为环境中重要的指示生物(Dailianis, 2011), 海洋贝类已经成为研究海洋微生物与宿主相互作用的重要模式生物(Pruzzo et al, 2005)。双壳贝类中已发现的主要共附生真菌为丝状真菌(Grovel et al, 2003; Zvereva et al, 2005; Santos et al, 2017; Borzykh et al, 2018), 例如链格孢属(Alternaria)、曲霉属(Aspergillus)、木霉属(Trichoderma)、青霉属(Penicillium)等。“双壳软体动物-丝状真菌”的相互作用已成为真菌生态学和群落学研究的重要领域(Borzykh et al, 2018)。目前对贝类相关真菌的研究多集中在生物化学和分子生物学, 其主要目的在于发现新的真菌来源的生物活性分子, 用于研发新物质或解释对抗病原体作用的防御机制。然而贝类体内抗菌活性物质的来源复杂, 贝类与真菌的相互作用关系尚不全面, 因此开展贝类宿主体内海洋真菌多样性研究为解析真菌在宿主免疫和海洋生态中的作用提供基础是十分必要的。
厚壳贻贝(Mytilus coruscus)营养价值高(何建瑜等, 2012), 具有较强的环境适应性和免疫能力(Liao et al, 2013; Yang et al, 2022)。作为我国第四大水产养殖贝类, 其年产量超过80万t。浙江嵊泗被誉为“贻贝之乡”, 其厚壳贻贝产量约占全国产量的1/4。作为一种高经济价值的贝类, 厚壳贻贝也具有不可忽视的生态价值, 如它可以去除水体中的悬浮沉积物, 净化水质(Neori et al, 2004); 也常作为监测污染物的指示生物(刘硕博, 2019)。厚壳贻贝组织表面附着大量细菌, 具有明显的组织差异性(何治江等, 2022), 可以影响宿主贻贝的生理代谢过程(吴冠举等, 2023), 影响周围海域水体细菌群落(He et al, 2023)和沉积物理化性质(吴紫琪等, 2023)。目前已有研究表明厚壳贻贝组织中存在共附生真菌(Komatsu et al, 2000), 且被认为是机会致病菌(Guarro et al, 1999; Borzykh et al, 2018), 但有关其组织差异性的研究还尚未见报道。
早期的真菌群落结构的研究手段主要为形态学观察, 且局限于特定基质上的可培养真菌(Coates et al, 1985), 也可通过气相色谱分析脂肪酸组成进行群落结构分析的情况(Bobbie et al, 1980)。作为多拷贝DNA片段、兼具保守区域和蕴含系统发育信息的可变区域, 核糖体DNA (rDNA)操纵子被视为真菌系统发育研究的优秀靶点(Kauserud, 2023)。ITS是内源转录间隔区(Internally Transcribed Spacer), 位于真菌18S rRNA、5.8S rRNA和28S rRNA基因之间。尽管核糖体大亚基(LSU)和小亚基(SSU) DNA和ITS片段都曾用于系统发育研究, 但ITS片段受自然选择压力小, 进化更快、差异明显(Gardes et al, 1991; Bruns et al, 1992; Gardes et al, 1993; Hibbett et al, 1993), 目前已成为真菌DNA条形码和群落多样性研究的重要标记基因(Schoch et al, 2012; De Beeck et al, 2014; Vitali et al, 2016; Quek et al, 2021; Walker et al, 2021; Zeng et al, 2023; Longley et al, 2024)。
为了解厚壳贻贝组织及其养殖区不同距离海水环境的真菌群落差异性, 本研究以浙江嵊泗厚壳贻贝养殖区为研究海域, 通过ITS rDNA扩增子测序对养殖区内、边缘及外围海域真菌群落多样性调查; 采集包括足、消化腺、后闭壳肌、肾脏、性腺、外套膜、血淋巴和鳃等不同组织进行真菌群落结构解析, 为深入了解微生物-贻贝相互作用以及海洋真菌在海水养殖活动中的变化提供科学依据。
1 材料与方法 1.1 样本采集在前期有关贻贝养殖区细菌群落的研究基础上(He et al, 2023), 2021年3月, 对浙江省舟山市嵊泗县枸杞岛(122°45′E, 30°42′N)厚壳贻贝养殖区内(BA)、边缘(MBA)及外围(离养殖区约1 km) (OBA)海域利用标准采水器收集深度约5 m的表层海水2 L, 各4组平行样本; 同时在养殖区内和边缘区各随机采集厚壳贻贝(2龄)样本30只。所有样本现场标记后立即返回实验室。海水样本使用0.22 μm醋酸纤维素滤膜进行抽滤。厚壳贻贝样本则在返回实验室后解剖, 获取每个海域厚壳贻贝后闭壳肌、消化腺、足、鳃、性腺、血淋巴、肾脏、外套膜8种组织样本, 经无菌水充分漂洗分装在4个50 mL离心管中标记待用。所有组织和滤膜样本置于–80 ℃保存, 以备后续实验。
1.2 样品总DNA提取所有样本用TIANGEN Genomic DNA Kit (DP305, 中国)试剂盒提取总DNA, 方法参照试剂盒说明书进行, 通过1%琼脂糖凝胶电泳和Nanodrop 2000 (THERMO, 美国)分别检测样本总DNA的分子量、浓度和纯度。经检验合格后, 一部分置于–20 ℃保存, 另一部分用于开展ITS基因绝对定量和测序文库构建。
1.3 ITS rDNA绝对定量为量化样品中的真菌丰度, 参照相关文献(Song et al, 2014; Liu et al, 2021b), 采用扩增通用引物(White et al, 1990): ITS1-F (5′-TCCGTAGGTGAACC TGCGG-3′)和ITS2-R (5′-GCTGCGTTCTTCATCGA TGC-3′)进行ITS基因绝对定量(拷贝数)。首先制作ITS基因拷贝数-Ct标准曲线。PCR扩增体系(20 μL)为: DNA模板20 ng, TB Green Premix Ex Taq Ⅱ (Tli RNaseH Plus) 10 μL, ITS1-F (10 μmol/L) 0.8 μL, ITS2-R (10 μmol/L) 0.8 μL, ddH2O补充至20 μL。PCR反应条件为: 95 ℃预变性30 s, 40个反应循环(95 ℃变性5 s, 60 ℃退火延伸1 min)。PCR扩增产物经1.2%琼脂糖凝胶电泳检测后进行胶回收, 其操作参照TIANGEN胶回收试剂盒(DP209)说明书进行。利用pLB零背景快速克隆试剂盒(TIANGEN, VT205)构建重组质粒, 方法参照试剂盒说明书进行。挑取阳性克隆进行液体恒温震荡(150 r/min, 37 ℃)培养过夜。采用质粒提取试剂盒(TIANGEN, DP105)提取质粒DNA, 利用试剂盒(TIANGEN, VT205)所提供的pLB-simple forward primer (10 μmol/L)和pLB-simple reverse primer (10 μmol/L)进行PCR扩增和产物测序验证。将验证无误的质粒测定DNA浓度后进行等梯度稀释, 利用Nanodrop测定其纯度和浓度, 计算基因拷贝数(copies/μL)= [质粒浓度(ng/μL)×10–9 ×6.02× 1023]/[样本分子量(g/mol)], 其中样本分子量为碱基数×324, 获得质粒标准拷贝数, 并以102~109 copies/μL的质粒标准品制作ITS基因拷贝数-Ct标准曲线。样本绝对定量PCR反应体系和扩增条件同标准曲线, 其中样本DNA加入量均为20 ng, 所有样本重复4次。
1.4 ITS rDNA文库构建及高通量测序为揭示真菌群落结构特征, 本文采用ITS rDNA高通量测序方法。样本总DNA经Qubit Fluorometer检测其浓度和纯度合格后进行测序文库构建。文库构建采用真菌通用引物ITS3 (5′-GCATCGTGAAGAA CGCAGC-3′)和ITS4 (5′-TCCTCCGCTTATTGATAT GC-3′)进行, 扩增体系和反应条件参照KAPA HiFi HotStart PCR Kit试剂盒(KAPA Biosystems, Roche, 美国)进行。测序文库定量检测依托KAPA Library Quantification Kit试剂盒进行, 经检验合格后进行两端并行测序(PE250, Illumina MiSeq测序平台)。ITS rDNA高通量测序在浙江杭州开泰生物技术有限公司进行。
1.5 生物信息学分析利用Vsearch v2.13.6对原始序列(Raw reads)进行拼接, 通过Cutadapt v2.4去除序列标签(Barcode), 然后采用Vsearch对低质量序列、嵌合体序列以及长度 < 100 bp的序列进行去除, 进而对高质量序列(Clean reads)进行统计。进一步地, 采用MOTHUR按照序列相似度 > 97%进行OTU (operational taxonomic unite)聚类。OTU代表序列的物种注释采用UNITE数据库。去除低丰度OTU (Reads < 2)后进行抽平分析, 获得OTU_TAX表。真菌群落多样性分析及数据可视化均在R v4.1.3环境下调用phyloseq (McMurdie et al, 2013)、microeco (Liu et al, 2021a)、vegan (Dixon, 2003)、ggplot2等包进行。
1.6 统计分析样本间Alpha多样性指数及基因绝对定量的显著性差异主要采用单因素方差分析(one way ANOVA), 组间多重比较采用Student-Newman-Keuls (SNK)检验进行。P < 0.05表示具有显著差异。
2 结果 2.1 厚壳贻贝养殖区及组织真菌ITS-1序列丰度差异明显通过构建ITS基因拷贝数-Ct标准曲线(图 1a)量化组织真菌丰度分布差异。定量结果显示厚壳贻贝养殖区内及边缘海域海水真菌生物量分别为2.92×108和3.03×108 copies/L, 显著高于外围海域(0.89×108 copies/L, P < 0.05, 图 1b)。对后闭壳肌, 消化腺, 血淋巴等8种组织进行真菌生物量测定, 结果表明厚壳贻贝不同组织真菌丰度差异显著(P < 0.05, 图 1c), 其中肾脏最高(6.63×106 copies/ng DNA), 其次为后闭壳肌(2.20× 106 copies/ng DNA), 性腺(1.96×106 copies/ng DNA)、足(1.90×106 copies/ng DNA)、外套膜(1.74×106 copies/ng DNA), 消化腺(1.25×106 copies/ng DNA)和血淋巴(1.03×106 copies/ng DNA), 最低为鳃, 仅有0.65×106 copies/ng DNA。
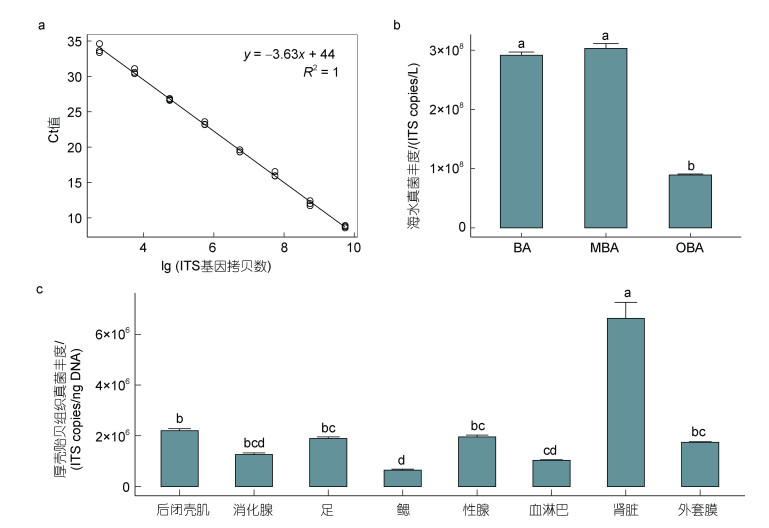 |
| 图 1 厚壳贻贝养殖海域及不同组织真菌丰度分析 Fig. 1 Fungal abundance in M. coruscus cultivation seawater and tissues 注: a. ITS基因拷贝数-Ct标准曲线; b. 厚壳贻贝养殖区内、边缘及外围海域真菌ITS基因拷贝数定量分析; c. 厚壳贻贝8种组织真菌ITS基因拷贝数定量分析。BA: 养殖区内海水; MBA: 养殖区边缘海水; OBA: 养殖区外围海水, 下同。柱形图上方的字母不同代表具有显著性差异 |
通过ITS rDNA高通量测序分析厚壳贻贝养殖区水体及组织真菌群落结构。从8种厚壳贻贝组织和3种养殖区海水共计76个样本中共获得2 480 003条高质量序列, 去除只包含1条序列的低丰度OTU后进行抽平分析, 在97%序列相似度上共获得1 409个OTUs, 分属于3个门(图 2a), 分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和接合菌门(Zygomycota)。丰度较高的子囊菌门相对丰度分布为养殖中心海域(26.76%) < 边缘海域(49.37%) < 外围海域(43.91%)。担子菌门在厚壳贻贝养殖中心海域的相对丰度较高(5.27%), 边缘海域的相对丰度仅为1.28%。在厚壳贻贝不同组织中, 子囊菌门在足、肾、外套膜中的相对丰度较高, 分别为22.50%、17.36%和10.38%。担子菌门在闭壳肌和血淋巴中的相对丰度较高, 分别为8.15%和6.94%, 最低为肾脏, 仅为0.77%。接合菌门只在厚壳贻贝的肾脏中被注释到, 其相对丰度仅占0.002 1%。可能受限于真菌数据库, 结果中仍有大量OTUs不能被注释, 其中厚壳贻贝组织真菌中未注释OTUs比例高于养殖区海水真菌, 主要为消化腺(92.78%) > 鳃(90.86%) > 性腺(88.64%) > 外套膜(88.52%) > 血淋巴(86.78%) > 后闭壳肌(84.56%) > 肾(81.87%) > 足(76.41%) > 中心海域(67.97%) > 边缘海域(49.37%) > 外围海域(43.91%)。
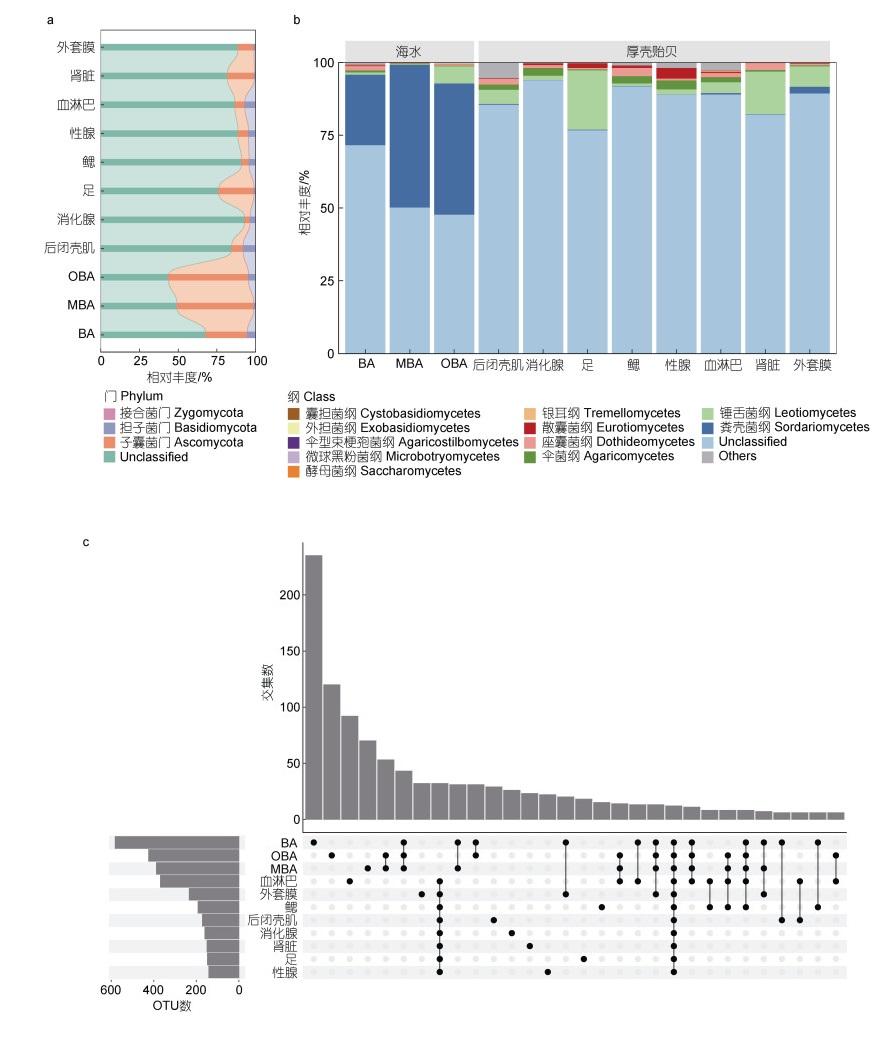 |
| 图 2 厚壳贻贝养殖海域及不同组织真菌群落结构分析 Fig. 2 Fungal community structure in M. coruscus cultivation seawater and different tissues 注: a. 厚壳贻贝8种组织和养殖区不同海域门水平下相对丰度分析; b. 纲水平下相对丰度分析; c. Upset图展示厚壳贻贝不同组织和养殖区海域真菌OTUs的共有和特有情况 |
在纲水平进行真菌群落分析结果显示, 除不能被注释的OTUs以外, 厚壳贻贝养殖区海域均以粪壳菌纲(Sordariomycetes)为优势菌纲(图 2b), 其中养殖区边缘(48.88%) > 养殖区外围(45.11%) > 养殖区内(24.18%)。在厚壳贻贝不同组织中, 主要以锤舌菌纲(Leotiomycetes)、座囊菌纲(Dothideomycetes)、散囊菌纲(Eurotiomycetes)、伞菌纲(Agaricomycetes)为主, 锤舌菌纲在足(20.13%)和肾脏(14.72%)中占比较高, 座囊菌纲在鳃(2.89%)、肾脏(2.31%)、后闭壳肌(1.92%)和血淋巴(1.36%)中占比较高, 散囊菌纲在性腺(3.59%)和足(1.57%)中占比较高, 伞菌纲相对丰度在消化腺(2.71%)、性腺(3.09%)和鳃(2.50%)中占比较高。对厚壳贻贝养殖区及不同组织独有和共有真菌类群进行分析, 结果(图 2c)表明厚壳贻贝养殖区内的OTUs数最高(581个), 其次为养殖区外围(425个)及边缘海域(388个)。厚壳贻贝血淋巴和外套膜中的真菌OTUs数最高, 分别为369个和234个, 而性腺最少, 仅有141个OTUs。厚壳贻贝和养殖区海域中均存在的OTUs仅有10个。独有OTUs数最高的为贻贝养殖区内海域(230个), 其次为外围海域(129个)、血淋巴(95个)和边缘海域(76个)。
2.3 厚壳贻贝养殖区及组织真菌群落多样性分析在了解厚壳贻贝养殖区海水及组织真菌组成基础上, 本文继续对其多样性进行分析。Alpha多样性结果显示厚壳贻贝养殖区不同区域的海水真菌的多样性差异较小, 仅中心区域真菌在OTU数和香农指数显著高于边缘海域(P < 0.05), 但仍高于厚壳贻贝组织真菌(图 3a~3d)。厚壳贻贝不同组织的真菌群落多样性分析发现血淋巴的多样性最高, 其次是鳃和外套膜, 而肾脏最低(图 3a~3d)。基于Bray-Curtis相似距离分析表明厚壳贻贝8个组织的真菌群落相似性较高, 且接近于养殖区内海水真菌群落, 但显著有异于养殖区边缘及外围海域的真菌群落(图 3e)。
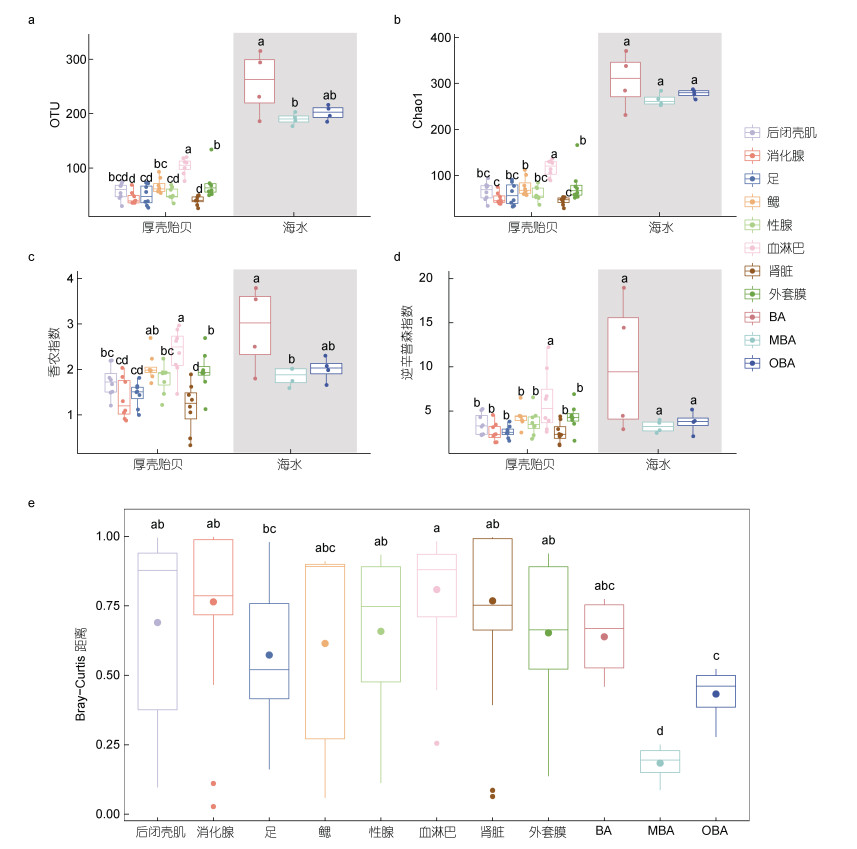 |
| 图 3 厚壳贻贝养殖海域及不同组织真菌群落Alpha多样性及Bray-curtis距离分析 Fig. 3 Alpha diversity and Bray-curtis analysis of fungal community from cultivation seawater and M. coruscus tissues 注: a. 厚壳贻贝不同组织和不同海域OTUs数比较; b. 厚壳贻贝不同组织和不同海域Chao1指数比较; c. 厚壳贻贝不同组织和不同海域香农指数比较; d. 厚壳贻贝不同组织和不同海域逆辛普森指数比较; e. 厚壳贻贝不同组织和不同海域逆Bray-Curtis距离比较。箱形图上方的字母不同代表具有显著性差异 |
主坐标分析(Principal Coordinates Analysis, PCoA)显示厚壳贻贝组织真菌群落显著不同于海水真菌群落(图 4a), 这与PERMANOVA差异分析结果一致(P < 0.05)。Beta多样性分解表明, 真菌群落物种替换和丰富度差异导致了厚壳贻贝养殖区海水与不同组织真菌群落组成的差异(图 4b)。物种替换和丰富度差异过程是导致养殖区海水真菌和厚壳贻贝组织真菌群落差异的主要因素(P < 0.01, 图 4c, 4d)。具体来说, 厚壳贻贝养殖区不同海域之间真菌群落组成差异以物种替换过程为主, 平均贡献了总差异的83.48%, 厚壳贻贝不同组织之间的差异是由物种替换过程导致, 占65.20%, 而养殖区海水真菌和厚壳贻贝组织真菌之间群落差异则大部分为丰富度差异过程驱动(61.99%)。
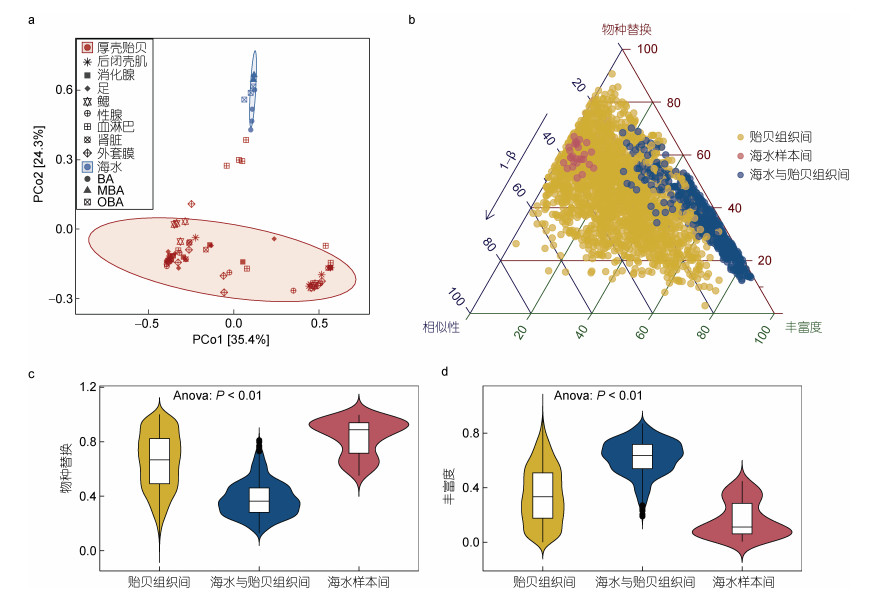 |
| 图 4 厚壳贻贝养殖海域及不同组织真菌群落Beta多样性分析 Fig. 4 Beta diversity of fungal community from farming seawater and M. coruscus tissues 注: a. PCoA分析; b. 分解Beta多样性的三元图。图中每个点代表一对样本间差异, 其位置由相似性、物种替换和丰富度差异矩阵的值决定; c. 样本间物种替换; d. 样本间丰富度差异分析 |
厚壳贻贝的养殖活动显著提高了养殖区内及边缘海水的真菌生物量。已有研究发现贻贝的养殖活动可改变周围海域中的藻类群落(陈明超等, 2011; 邓明星, 2016; 包炎琳等, 2020), 藻类群落影响水体有机质组成(王旭, 2015), 为真菌的生长提供了必要营养物质和更多的生态位。从门水平看, 厚壳贻贝养殖区海域主要为子囊菌门、担子菌门和接合菌门等。子囊菌门是真菌界中种类最多的一个门, 粪壳菌纲是其中第二大纲, 广泛存在于海洋环境中, 在沿海和海洋空气中占优势(于显东, 2019; 江建平等, 2022), 与王雪等(2021)在该海域温暖季节的真菌群落结构类似。段瑛博(2018)等采用高通量测序对卡罗来纳州沿海表层水的真菌群落进行分析, 结果显示, 该区域表层水真菌群落全年以子囊门中的粪壳菌纲为主导。任娜等(2022)从浅海、半深海和深海带分离的可培养真菌中, 粪壳菌纲真菌占25%。粪壳菌纲是有机质的分解者, 编码大量的碳水化合物活性酶, 对降解植物碎屑具有重要作用(Bugni et al, 2004; Strickland et al, 2009; Baltar et al, 2021; Clocchiatti et al, 2023)。粪壳菌纲也存在植物内生菌、植物病原体及昆虫和其他动物的致病菌(Zhang et al, 2015)。然而厚壳贻贝组织中粪壳菌纲平均相对丰度(0.46%)显著低于环境水样本(39.39%)中的, 说明厚壳贻贝对海水中的粪壳菌有一定的选择性。厚壳贻贝组织中具有较高丰度的锤舌菌纲, 已有相关文献报道在赤潮爆发后, 该纲真菌丰度增加, 且被认为在藻际环境中具有较强的竞争力(谢伟娟等, 2021), 因此厚壳贻贝组织中锤舌菌纲的增加可能与贻贝对藻类的滤食有关。座囊菌纲真菌也被认为与海洋中褐藻和红藻有关(Gnavi et al, 2017)。
厚壳贻贝不同组织中的真菌类群丰度和多样性存在较为明显的组织差异性, 其中肾脏所含有的真菌丰度最高, 而血淋巴中的多样性最高。该结果也与何治江等(2022)在厚壳贻贝血淋巴中细菌多样性的研究结果一致。如此的组织差异性一方面受到养殖水体的环境影响, 另一方面也可能受到厚壳贻贝免疫系统的影响。如血淋巴被认为可能具有从环境中获取组织中不存在的细菌的能力(Canesi et al, 2002; Pierce et al, 2018), 对于真菌而言, 也可能有类似的机制使血淋巴的真菌群落与周围海水相似。贻贝是已知抗菌肽分子多样性最为丰富的海洋软体动物之一(Mitta et al, 1999a, 1999b; Gerdol et al, 2012; Sonthi et al, 2012; Leoni et al, 2017; Yang et al, 2022), 不同组织中的抗菌肽存在显著的表达差异性且对真菌具有持续性的强免疫反应。厚壳贻贝抗菌肽Mytimycin、Mytcalin、ALD-2和Crustin-like的基因表达量在以往研究中表现出对真菌胁迫的较高敏感性(Yang et al, 2022)。此外, 基于厚壳贻贝mytilin抗菌肽活性片段设计的人工肽MDP-1和MDP-2表现出明显的体外抗真菌活性(杨金月, 2022); 上述结果表明, 厚壳贻贝体内有部分抗菌肽分子参与了对真菌的免疫作用, 可能会导致贻贝不同组织之间真菌群落结构的差异。
Beta多样性的分解显示贻贝不同组织间和不同海域海水间的差异主要来自物种替换, 这说明贻贝组织间及海水间的空间异质性导致了贻贝体内与海水间的真菌群落结构差异(Bond et al, 2001; Baselga, 2010); 与之相对, 海水和贻贝的Beta多样性差异则主要来自丰富度差异, 反映了一种非随机的物种流失(Wright et al, 1992; Almeida-Neto et al, 2008), 这种流失是由可促使群落有序分解的因素造成的(Bond et al, 2001)。因此接下来仍有必要对贝类组织真菌的群落构建机制展开系统的研究, 以充分掌握贝类组织微生物的形成过程。
4 结论与展望本文围绕厚壳贻贝养殖活动, 结合ITS rDNA高通量测序对厚壳贻贝养殖区海水及组织真菌群落的生物量和群落多样性进行分析。研究结果显示厚壳贻贝养殖活动提高了海水真菌丰度并改变了其真菌群落结构, 厚壳贻贝不同组织真菌群落间具有显著差异性。上述结果进一步地表明贻贝养殖区微生物群落的特殊性, 以及养殖活动对周围海域的深刻影响。另一方面, 大量未注释的真菌OTUs显示真菌注释数据库亟待进一步补充和优化, 也暗示贝类养殖区中蕴含新的真菌生物资源。
于显东, 2019. 粪壳菌纲Chaetosphaeriaceae科分子系统学研究及十六株淡水真菌新种的分类鉴定[D]. 昆明: 昆明理工大学.
|
王旭, 2015. 枸杞岛贻贝养殖营养水平及其碳效应初步研究[D]. 上海: 上海海洋大学.
|
王雪, 肖劲洲, 王永杰, 2021. 枸杞岛海域表层水真菌群落多样性研究[J]. 微生物学杂志, 41(6): 11-17. |
邓明星, 2016. 贻贝筏式养殖对海域水动力及浮游植物生态系统影响[D]. 上海: 上海海洋大学.
|
包炎琳, 段元亮, 杨娜, 等, 2020. 嵊泗列岛贻贝养殖区与无人岛潮间带大型海藻群落结构比较[J]. 海洋渔业, 42(5): 595-607. |
任娜, 朱四东, 陈秀暖, 等, 2022. 海水来源的548株真菌的近海及远海分布模式及其抑菌活性分析[J]. 微生物学报, 62(1): 374-387. |
刘硕博, 2019. 厚壳贻贝含硒GPx克隆及在Cu2+和LPS胁迫下的表达[D]. 舟山: 浙江海洋大学.
|
江建平, 杜诚, 刘冰, 等, 2022. 中国生物物种编目进展与展望[J]. 生物多样性, 30(10): 22531. |
杨金月, 2022. 厚壳贻贝抗菌肽免疫表达模式及组学分析[D]. 舟山: 浙江海洋大学.
|
吴冠举, 缪天音, 胡晓梦, 等, 2023. 外源添加胞外囊泡对海假交替单胞菌生物被膜形成及厚壳贻贝附着的影响[J]. 大连海洋大学学报, 38(6): 994-1002. |
吴紫琪, 何语堂, 羊妍珂, 等, 2023. 厚壳贻贝(Mytilus coruscus)养殖区沉积物微生物多样性及固碳潜力研究[J]. 海洋与湖沼, 54(2): 502-513. |
何治江, 贾梦雪, 王锦乙, 等, 2022. 厚壳贻贝不同组织中的微生物群落结构[J]. 水产学报, 46(12): 2421-2431. |
何建瑜, 赵荣涛, 刘慧慧, 2012. 舟山海域厚壳贻贝软体部分营养成分分析与评价[J]. 南方水产科学, 8(4): 37-42. |
陈明超, 章守宇, 2011. 枸杞岛海域浮游植物群落结构分析[J]. 上海海洋大学学报, 20(4): 607-612. |
段瑛博, 2018. 沿海水域中真菌及真菌类原生生物的时空分布模式[D]. 天津: 天津大学.
|
谢伟娟, 陈晨, 赵志颖, 等, 2021. 象山港硅藻赤潮过程中真菌群落演替及互作特征[J]. 微生物学通报, 48(11): 3958-3970. |
谢林青, 曹为, 蒋凤华, 等, 2022. 印度洋深海热液区贻贝足丝结构和性能特征研究[J]. 海洋与湖沼, 53(2): 364-374. |
ALMEIDA-NETO M, GUIMARÃES P, GUIMARÃES JR P R, et al, 2008. A consistent metric for nestedness analysis in ecological systems: reconciling concept and measurement[J]. Oikos, 117(8): 1227-1239. DOI:10.1111/j.0030-1299.2008.16644.x |
BALTAR F, ZHAO Z H, HERNDL G J, 2021. Potential and expression of carbohydrate utilization by marine fungi in the global ocean[J]. Microbiome, 9(1): 106. DOI:10.1186/s40168-021-01063-4 |
BÄRLOCHER F, BRENDELBERGER H, 2004. Clearance of aquatic hyphomycete spores by a benthic suspension feeder[J]. Limnology and Oceanography, 49(6): 2292-2296. DOI:10.4319/lo.2004.49.6.2292 |
BASELGA A, 2010. Partitioning the turnover and nestedness components of beta diversity[J]. Global Ecology and Biogeography, 19(1): 134-143. DOI:10.1111/j.1466-8238.2009.00490.x |
BOBBIE R J, WHITE D C, 1980. Characterization of benthic microbial community structure by high-resolution gas chromatography of Fatty Acid methyl esters[J]. Applied and Environmental Microbiology, 39(6): 1212-1222. DOI:10.1128/aem.39.6.1212-1222.1980 |
BOND W J, SMYTHE K A, BALFOUR D A, 2001. Acacia species turnover in space and time in an African savanna[J]. Journal of Biogeography, 28(1): 117-128. DOI:10.1046/j.1365-2699.2001.00506.x |
BORZYKH O G, ZVEREVA L V, 2018. Fungal assemblages associated with commercial bivalve species in coastal waters of the Sea of Japan, Russia[J]. Botanica Marina, 61(4): 355-363. DOI:10.1515/bot-2017-0088 |
BRUNS T D, VILGALYS R, BARNS S M, et al, 1992. Evolutionary relationships within the fungi: analyses of nuclear small subunit rRNA sequences[J]. Molecular Phylogenetics and Evolution, 1(3): 231-241. DOI:10.1016/1055-7903(92)90020-H |
BUGNI T S, IRELAND C M, 2004. Marine-derived fungi: a chemically and biologically diverse group of microorganisms[J]. Natural Product Reports, 21(1): 143-163. DOI:10.1039/b301926h |
CANESI L, GALLO G, GAVIOLI M, et al, 2002. Bacteria-hemocyte interactions and phagocytosis in marine bivalves[J]. Microscopy Research and Technique, 57(6): 469-476. DOI:10.1002/jemt.10100 |
CARELLA F, DE VICO G, 2023. Pathology, epidemiology, and phylogeny of mussel egg disease due to the microsporidian Steinhausia mytilovum (Field, 1924) in the Mediterranean mussel (Mytilus galloprovincialis)[J]. Journal of Invertebrate Pathology, 198: 107927. DOI:10.1016/j.jip.2023.107927 |
CLOCCHIATTI A, HANNULA S E, HUNDSCHEID M P J, et al, 2023. Utilizing woody materials for fungal-based management of soil nitrogen pools[J]. Applied Soil Ecology, 181: 104663. DOI:10.1016/j.apsoil.2022.104663 |
COATES D, RAYNER A D M, 1985. Fungal population and community development in cut beech logs: Ⅱ. Establishment via the buried cut surface[J]. New Phytologist, 101(1): 173-181. DOI:10.1111/j.1469-8137.1985.tb02824.x |
DAILIANIS S, 2011. Environmental impact of anthropogenic activities: the use of mussels as a reliable tool for monitoring marine pollution[J]. Mussels: Anatomy, Habitat and Environmental Impact; McGevin, LE, Ed: 43-72. |
DE BEECK M O, LIEVENS B, BUSSCHAERT P, et al, 2014. Comparison and validation of some ITS primer pairs useful for fungal metabarcoding studies[J]. PLoS One, 9(6): e97629. DOI:10.1371/journal.pone.0097629 |
DIXON P, 2003. VEGAN, a package of R functions for community ecology[J]. Journal of Vegetation Science, 14(6): 927-930. DOI:10.1111/j.1654-1103.2003.tb02228.x |
FRANCHINI A, MALAGOLI D, OTTAVIANI E, 2005. Investigation of the loss of byssus in Mytilus galloprovincialis from mussel farms in the Adriatic Sea[J]. Cell Biology International, 29(10): 857-860. DOI:10.1016/j.cellbi.2005.06.001 |
GARDES M, BRUNS T D, 1993. ITS primers with enhanced specificity for basidiomycetes-application to the identification of mycorrhizae and rusts[J]. Molecular Ecology, 2(2): 113-118. DOI:10.1111/j.1365-294X.1993.tb00005.x |
GARDES M, WHITE T J, FORTIN J A, et al, 1991. Identification of indigenous and introduced symbiotic fungi in ectomycorrhizae by amplification of nuclear and mitochondrial ribosomal DNA[J]. Canadian Journal of Botany, 69(1): 180-190. DOI:10.1139/b91-026 |
GATESOUPE F J, 1999. The use of probiotics in aquaculture[J]. Aquaculture, 180(1/2): 147-165. |
GERDOL M, DE MORO G, MANFRIN C, et al, 2012. Big defensins and mytimacins, new AMP families of the Mediterranean mussel Mytilus galloprovincialis[J]. Developmental & Comparative Immunology, 36(2): 390-399. |
GNAVI G, GARZOLI L, POLI A, et al, 2017. The culturable mycobiota of Flabellia petiolata: first survey of marine fungi associated to a Mediterranean green alga[J]. PLoS One, 12(4): e0175941. DOI:10.1371/journal.pone.0175941 |
GROVEL O, POUCHUS Y F, VERBIST J F, 2003. Accumulation of gliotoxin, a cytotoxic mycotoxin from Aspergillus fumigatus, in blue mussel (Mytilus edulis)[J]. Toxicon, 42(3): 297-300. DOI:10.1016/S0041-0101(03)00146-6 |
GUARRO J, GENÉ J, STCHIGEL A M, 1999. Developments in fungal taxonomy[J]. Clinical Microbiology Reviews, 12(3): 454-500. DOI:10.1128/CMR.12.3.454 |
GUTIÉRREZ M H, JARA A M, PANTOJA S, 2016. Fungal parasites infect marine diatoms in the upwelling ecosystem of the Humboldt current system off central Chile[J]. Environmental Microbiology, 18(5): 1646-1653. DOI:10.1111/1462-2920.13257 |
HATAI K, 2012. Diseases of fish and shellfish caused by marine fungi [M] // RAGHUKUMAR C. Biology of Marine Fungi. Berlin: Springer: 15-52.
|
HE J Y, JIA M X, WANG J X, et al, 2023. Mytilus farming drives higher local bacterial diversity and facilitates the accumulation of aerobic anoxygenic photoheterotrophic related genera[J]. Science of the Total Environment, 856: 158861. DOI:10.1016/j.scitotenv.2022.158861 |
HIBBETT D S, VILGALYS R, 1993. Phylogenetic relationships of Lentinus (Basidiomycotina) inferred from molecular and morphological characters[J]. Systematic Botany, 18(3): 409-433. DOI:10.2307/2419417 |
HYDE K D, JONES E B G, LEAÑO E, et al, 1998. Role of fungi in marine ecosystems[J]. Biodiversity & Conservation, 7(9): 1147-1161. |
KAUSERUD H, 2023. ITS alchemy: on the use of ITS as a DNA marker in fungal ecology[J]. Fungal Ecology, 65: 101274. DOI:10.1016/j.funeco.2023.101274 |
KIRAN G S, SEKAR S, RAMASAMY P, et al, 2018. Marine sponge microbial association: towards disclosing unique symbiotic interactions[J]. Marine Environmental Research, 140: 169-179. DOI:10.1016/j.marenvres.2018.04.017 |
KOMATSU K, SHIGEMORI H, MIKAMI Y, et al, 2000. Sculezonones A and B, two metabolites possessing a phenalenone skeleton from a marine-derived fungus Penicillium species[J]. Journal of Natural Products, 63(3): 408-409. DOI:10.1021/np990452t |
KOVAČIĆ I, PUSTIJANAC E, RAMŠAK A, et al, 2018. Variation of parasite and fungi infection between farmed and wild mussels (Mytilus galloprovincialis Lamarck, 1819) from the Adriatic Sea[J]. Journal of the Marine Biological Association of the United Kingdom, 98(8): 1871-1879. DOI:10.1017/S0025315418000644 |
LEONI G, DE POLI A, MARDIROSSIAN M, et al, 2017. Myticalins: a novel multigenic family of linear, cationic antimicrobial peptides from marine mussels (Mytilus spp.)[J]. Marine Drugs, 15(8): 261. DOI:10.3390/md15080261 |
LIAO Z, WANG X C, LIU H H, et al, 2013. Molecular characterization of a novel antimicrobial peptide from Mytilus coruscus[J]. Fish & Shellfish Immunology, 34(2): 610-616. |
LIN G M, LU J G, SUN Z L, et al, 2021. Characterization of tissue-associated bacterial community of two Bathymodiolus species from the adjacent cold seep and hydrothermal vent environments[J]. Science of the Total Environment, 796: 149046. DOI:10.1016/j.scitotenv.2021.149046 |
LIU G F, BAI Z J, SHAH F, et al, 2021b. Compositional and structural changes in soil microbial communities in response to straw mulching and plant revegetation in an abandoned artificial pasture in Northeast China[J]. Global Ecology and Conservation, 31: e01871. DOI:10.1016/j.gecco.2021.e01871 |
LIU C, CUI Y M, LI X Z, et al, 2021a. microeco: an R package for data mining in microbial community ecology[J]. FEMS Microbiology Ecology, 97(2): fiaa255. DOI:10.1093/femsec/fiaa255 |
LONGLEY R, BENUCCI G M N, POCHON X, et al, 2024. Species-specific coral microbiome assemblages support host bleaching resistance during an extreme marine heatwave[J]. Science of the Total Environment, 906: 167803. DOI:10.1016/j.scitotenv.2023.167803 |
MCMURDIE P J, HOLMES S, 2013. phyloseq: an R package for reproducible interactive analysis and graphics of microbiome census data[J]. PLoS One, 8(4): e61217. DOI:10.1371/journal.pone.0061217 |
MITTA G, HUBERT F, NOËL T, et al, 1999a. Myticin, a novel cysteine-rich antimicrobial peptide isolated from haemocytes and plasma of the mussel Mytilus galloprovincialis[J]. European Journal of Biochemistry, 265(1): 71-78. DOI:10.1046/j.1432-1327.1999.00654.x |
MITTA G, VANDENBULCKE F, HUBERT F, et al, 1999b. Mussel defensins are synthesised and processed in granulocytes then released into the plasma after bacterial challenge[J]. Journal of Cell Science, 112(23): 4233-4242. DOI:10.1242/jcs.112.23.4233 |
NEORI A, CHOPIN T, TROELL M, et al, 2004. Integrated aquaculture: rationale, evolution and state of the art emphasizing seaweed biofiltration in modern mariculture[J]. Aquaculture, 231(1/2/3/4): 361-391. |
ORSI W D, VUILLEMIN A, COSKUN Ö K, et al, 2022. Carbon assimilating fungi from surface ocean to subseafloor revealed by coupled phylogenetic and stable isotope analysis[J]. The ISME Journal, 16(5): 1245-1261. DOI:10.1038/s41396-021-01169-5 |
PHILLIPS N W, 1984. Role of different microbes and substrates as potential suppliers of specific, essential nutrients to marine detritivores[J]. Bulletin of Marine Science, 35(3): 283-298. |
PIERCE M L, WARD J E, 2018. Microbial ecology of the bivalvia, with an emphasis on the family ostreidae[J]. Journal of Shellfish Research, 37(4): 793-806. DOI:10.2983/035.037.0410 |
PRUZZO C, GALLO G, CANESI L, 2005. Persistence of vibrios in marine bivalves: the role of interactions with haemolymph components[J]. Environmental Microbiology, 7(6): 761-772. DOI:10.1111/j.1462-2920.2005.00792.x |
QUEK Z B R, ZAHN G, LEE N L Y, et al, 2021. Biogeographic structure of fungal communities in seagrass Halophilia ovalis across the Malay Peninsula[J]. Environmental Microbiology Reports, 13(6): 871-877. DOI:10.1111/1758-2229.13003 |
SANTOS A, HAUSER-DAVIS R A, SANTOS M J S, et al, 2017. Potentially toxic filamentous fungi associated to the economically important Nodipecten nodosus (Linnaeus, 1758) scallop farmed in southeastern Rio de Janeiro, Brazil[J]. Marine Pollution Bulletin, 115(1/2): 75-79. |
SCHOCH C L, SEIFERT K A, HUHNDORF S, et al, 2012. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi[J]. Proceedings of the National Academy of Sciences of the United States of America, 109(16): 6241-6246. |
SONG Z W, VAIL A, SADOWSKY M J, et al, 2014. Quantitative PCR for measuring biomass of decomposer fungi in planta[J]. Fungal Ecology, 7: 39-46. DOI:10.1016/j.funeco.2013.12.004 |
SONTHI M, CANTET F, TOUBIANA M, et al, 2012. Gene expression specificity of the mussel antifungal mytimycin (MytM)[J]. Fish & Shellfish Immunology, 32(1): 45-50. |
STRICKLAND M S, OSBURN E, LAUBER C, et al, 2009. Litter quality is in the eye of the beholder: initial decomposition rates as a function of inoculum characteristics[J]. Functional Ecology, 23(3): 627-636. DOI:10.1111/j.1365-2435.2008.01515.x |
TELESCA L, PECK L S, BACKELJAU T, et al, 2021. A century of coping with environmental and ecological changes via compensatory biomineralization in mussels[J]. Global Change Biology, 27(3): 624-639. DOI:10.1111/gcb.15417 |
VAN DOVER C L, WARD M E, SCOTT J L, et al, 2007. A fungal epizootic in mussels at a deep-sea hydrothermal vent[J]. Marine Ecology, 28(1): 54-62. DOI:10.1111/j.1439-0485.2006.00121.x |
VENIER P, VAROTTO L, ROSANI U, et al, 2011. Insights into the innate immunity of the Mediterranean mussel Mytilus galloprovincialis[J]. BMC Genomics, 12: 69. DOI:10.1186/1471-2164-12-69 |
VITALI F, MASTROMEI G, SENATORE G, et al, 2016. Long lasting effects of the conversion from natural forest to poplar plantation on soil microbial communities[J]. Microbiological Research, 182: 89-98. DOI:10.1016/j.micres.2015.10.002 |
WALKER A K, ROBICHEAU B M, 2021. Fungal diversity and community structure from coastal and barrier island beaches in the United States Gulf of Mexico[J]. Scientific Reports, 11(1): 3889. DOI:10.1038/s41598-021-81688-5 |
WEGLEY L, EDWARDS R, RODRIGUEZ-BRITO B, et al, 2007. Metagenomic analysis of the microbial community associated with the coral Porites astreoides[J]. Environmental Microbiology, 9(11): 2707-2719. DOI:10.1111/j.1462-2920.2007.01383.x |
WHITE T J, BRUNS T, LEE S, et al, 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics [M] // INNIS M A, GELFAND D H, SNINSKY J J, et al. PCR Protocols: A Guide to Methods and Applications. Amsterdam: Elsevier: 315-322.
|
WRIGHT D H, REEVES J H, 1992. On the meaning and measurement of nestedness of species assemblages[J]. Oecologia, 92(3): 416-428. DOI:10.1007/BF00317469 |
YAHYA N, IDRIS I, ROSLI N S, et al, 2020. Mangrove-associated bivalves in Southeast Asia: a review[J]. Regional Studies in Marine Science, 38: 101382. DOI:10.1016/j.rsma.2020.101382 |
YANG J Y, HE J Y, LIU L, et al, 2022. Expression profiles of antimicrobial peptides in Mytilus coruscus[J]. Aquaculture, 548: 737709. DOI:10.1016/j.aquaculture.2021.737709 |
ZENG Q C, LEBRETON A, AUER L, et al, 2023. Stable functional structure despite high taxonomic variability across fungal communities in soils of old-growth montane forests[J]. Microbiome, 11(1): 217. DOI:10.1186/s40168-023-01650-7 |
ZHANG Y, MU J, FENG Y, et al, 2009. Broad-spectrum antimicrobial epiphytic and endophytic fungi from marine organisms: isolation, bioassay and taxonomy[J]. Marine Drugs, 7(2): 97-112. DOI:10.3390/md7020097 |
ZHANG N, WANG Z, 2015. Pezizomycotina: sordariomycetes and leotiomycetes [M] // MCLAUGHLIN D J, SPATAFORA J W. Systematics and Evolution: Part B. 2nd ed. Berlin: Springer: 57-88.
|
ZVEREVA L V, VYSOTSKAYA M A, 2005. Filamentous fungi associated with bivalve mollusks from polluted biotopes of Ussuriiskii Bay, Sea of Japan[J]. Russian Journal of Marine Biology, 31(6): 382-385. DOI:10.1007/s11179-006-0007-3 |
 2024, Vol. 55
2024, Vol. 55


