
中国海洋湖沼学会主办。
文章信息
- 李妍, 姚健涛, 张恩烁, 孙泽轩, 孙国华, 李彬, 杨建敏, 冯艳微, 王卫军. 2024.
- LI Yan, YAO Jian-Tao, ZHANG En-Shuo, SUN Ze-Xuan, SUN Guo-Hua, LI Bin, YANG Jian-Min, FENG Yan-Wei, WANG Wei-Jun. 2024.
- 长牡蛎(Crassostrea gigas)野生与选育群体的微卫星遗传多样性分析
- MICROSATELLITE ANALYSIS ON GENETIC DIVERSITY OF BREEDING POPULATIONS AND WILD POPULATIONS OF CRASSOSTREA GIGAS
- 海洋与湖沼, 55(2): 462-470
- Oceanologia et Limnologia Sinica, 55(2): 462-470.
- http://dx.doi.org/10.11693/hyhz20231000219
文章历史
-
收稿日期:2023-10-24
收修改稿日期:2024-01-07
2. 牡蛎种质创制与高效养殖山东省工程研究中心 山东烟台 264025;
3. 烟台市崆峒岛实业有限公司 山东烟台 264001;
4. 烟台市海育海洋科技有限公司 山东烟台 264004
2. Shandong Engineering Research Center of Oyster Germplasm Creation and Efficient Culture, Yantai 264025, China;
3. Yantai Kongtongdao Industrial Co., Ltd., Yantai 264001, China;
4. Yantai Haiyu Marine Science and Technology Co., Ltd., Yantai 264004, China
长牡蛎(Crassostrea gigas)又称太平洋牡蛎, 其生长速度快、适应性强, 是我国重要的海水养殖经济物种。近年来我国工厂化育苗日益成熟, 长牡蛎养殖产量也逐年增加, 但人工繁育可能出现近亲繁殖等现象会造成群体遗传多样性降低, 伴随出现出肉率低, 死亡率高等问题, 制约着牡蛎产业的发展(张荣良等, 2016; 任晶莹等, 2018)。选育良种, 改善种质性能成为了牡蛎产业可持续发展的重中之重。
遗传多样性与动物的适应能力和分布范围呈正相关, 遗传多样性降低会使物种对环境的适应能力下降(李景芬等, 2020)。物种的遗传多样性评估至关重要, 一方面为种质资源保护提供科学数据参考, 当遗传多样性开始下降时应当采取相应措施加强对种质资源的保护; 另一方面在选育过程中, 对选育群体不同世代进行遗传多样性监测, 使其保持足够高的遗传多样性, 避免出现种质退化问题(孙成飞, 2020)。
微卫星标记(又名简单重复序列, Simple Sequence Repeats, SSR)和单核苷酸多态性标记(Single Nucleotide Polymorphism, SNP)已经被广泛应用于家系分析、遗传多样性分析和种质鉴定等领域(滕爽爽等, 2021; 梁园等, 2022)。田镇等(2021)利用15对微卫星引物分析红壳色文蛤选育群体世代遗传多样性变化和遗传分化情况。高杨等(2023)采用14对微卫星位点检测了8个地区克氏原螯虾的遗传多样性, 了解了不同地区克氏原螯虾的遗传背景, 为资源保护与新品种选育提供参考。在长牡蛎中, 张荣良等(2016)利用微卫星标记对长牡蛎选育基础群体和连续两代选育群体进行了遗传多样性跟踪监测, 为长牡蛎遗传育种工作有效开展提供了基础数据。Qi等(2009)构建了长牡蛎fosmid文库并对文库进行测序, 筛选出了33个微卫星标记用于野生长牡蛎的遗传多样性分析; 余勇(2015)基于长牡蛎表达序列(EST序列)和全基因组序列开发了480个微卫星位点, 从中选取30个位点用于自交群体和反交群体的遗传多样性检测。Zhong等(2016)利用103个SNP标记对3个选育群体和4个野生群体的遗传多样性水平进行评价, 发现选育群体和野生群体相比有遗传多样性丧失的趋势。
本团队自2014年开始开展长牡蛎壳长性状(掌心形)和壳高性状(速生型)快速生长新品种选育工作, 为评估选育过程中选育群体的遗传多样性水平, 本研究利用21个多态性丰富的微卫星位点, 分析了3个长牡蛎选育群体和6个野生群体的遗传多样性和遗传结构, 为进一步开展长牡蛎遗传育种、优良种质挖掘利用及种质资源保护提供参考。
1 材料与方法 1.1 样品采集2个长牡蛎壳长性状(掌心形)快速生长选育群体(LY2-K4、LY2-K7)和1个长牡蛎壳高性状(速生型)快速生长选育群体(LY2-K11)取自于烟台崆峒岛马岛育种基地, 6个长牡蛎野生群体(QHD、LS、HD、ZH、WD和KTD)分别采集于河北秦皇岛、日照岚山、青岛黄岛、大连庄河、威海文登和烟台崆峒岛, 样品如表 1所示。LY2-K4选育群体由KTD和WD群体进行群体间交配并经过连续4代选育获得, LY2-K7选育群体由HD和ZH群体进行群体间交配并经过连续4代选育获得; 四个基础群体雌雄亲本数量分别为300个个体, 选育过程中每代雌雄亲本数量分别保持在60~80个个体。LY2-K11选育群体亲本来源于KTD群体, 经过连续5代选育获得, 其基础群体雌雄亲本数量分别为150个个体, 选育过程中每代雌雄亲本数量分别保持在50~60个个体。测试样品除LY2-K7群体样本采集数量为29外, 其他群体样本采集数量均为30, 共采集269个样本。每个样本取部分闭壳肌组织固定于无水乙醇中, 置于–20 ℃保存, 用于基因组DNA提取。
| 缩写 | 样本采集地 | 样本采集时间 (年.月.日) |
样本数量 |
| LY2-K4 | 烟台崆峒岛 | 2023.06.09 | 30 |
| LY2-K7 | 烟台崆峒岛 | 2023.06.09 | 29 |
| LY2-K11 | 烟台崆峒岛 | 2023.06.09 | 30 |
| QHD | 河北秦皇岛 | 2014.07.10 | 30 |
| LS | 日照岚山 | 2014.08.10 | 30 |
| HD | 青岛黄岛 | 2014.08.12 | 30 |
| ZH | 大连庄河 | 2014.07.10 | 30 |
| WD | 威海文登 | 2014.07.15 | 30 |
| KTD | 烟台崆峒岛 | 2014.07.18 | 30 |
每个样本取30~50 mg组织用传统的苯酚-氯仿抽提法提取基因组DNA (Li et al, 2006), 提取出的DNA用1%琼脂糖凝胶电泳检测DNA纯度和完整度, 用微量紫外分光光度计(Thermo Scientific, 美国)检测DNA浓度。将质检合格的DNA稀释至浓度为100 ng/µL, 置于–20 ℃保存备用。
1.3 多态性微卫星引物筛选、PCR扩增及产物检测本研究从已发表文献(Li et al, 2003; Qi et al, 2009; 余勇, 2015)中随机挑选30对微卫星引物, 每个群体选择8个个体进行微卫星引物多态性筛选, 最终鉴定出21对多态性较好的微卫星引物(表 2)。在21对引物的正向引物5’端分别加上Fam、Tamra和Hex荧光标记, 用于PCR扩增和后续的毛细管电泳分型。本实验所用引物和荧光标记均由上海生工生物工程股份有限公司合成。
| 位点 | 引物序列 | 退火温度/℃ | 产物大小/bp | 荧光标记 |
| ucd-Cg109 | F: GCTATGGTTGTCATCCTCGAA R: TGCCTTTATCGGTTTTGCTT |
53 | 160~240 | Fam |
| ucd-Cg140 | F: TGCTCAATTCACAGCAATCAG R: TCTGACTGCTGAACAGCAAAAT |
60 | 211~295 | Tamra |
| ucd-Cg177 | F: GCTTCCGGGAATTAAACCAT R: TCAAGAAAAAGTCGACGGGTA |
60 | 200~290 | Hex |
| CgGJ196 | F: CGCACACCACGTATGTAAGG R: TGTCATCCATGTTACTTTCGCT |
54 | 180~300 | Fam |
| ucd-Cg149 | F: TGATTAAACGTGGGTGATTCAG R: TTTCTGACTGTCCGTCTGTGA |
60 | 190~300 | Tamra |
| CgGJ202 | F: TTTTGACTGGGGAGCGTATC R: CAGAGTTCAAATCTTCAAACTTTCC |
56 | 170~245 | Hex |
| ucd-Cg194 | F: CCCAGTGAAAACTTGGAGACA R: TTTCGAATCGGGAAAATACG |
52 | 238~297 | Fam |
| ucd-Cg160 | F: GGAGCCATTAACAACACCACA R: TCTCTCCCTTCCCCCTCTTA |
57 | 198~366 | Tamra |
| CgGJ330 | F: CAGGGTTTGTTGGCAGTG R: ATGTCATCTATGATAGACGAAGC |
56 | 209~296 | Hex |
| ucdCg-156 | F: AGCAGACCTTGGCAAATACG R: CCGTCATCAGGTCCTGTTTT |
50 | 115~254 | Fam |
| ucd-Cg162 | F: CCAAATCACCGTTTTAGTTTGTT R: AGCGACACAGAGACCACCTT |
55 | 167~324 | Tamra |
| ucdCg-126 | F: TGGATTTGATCACCCCTTACA R: CCTGGATTCTGTGCGAGATT |
55 | 98~244 | Hex |
| ucd-Cg175 | F: GGGCATGGATCAACTCCTAA R: CCAACCAGCCCTAGTCTGTG |
55 | 213~331 | Fam |
| ucd-Cg157 | F: GGGGGATGTCGGAGAAGTAT R: AACAGAGAAAGGTGGATTTTAGGA |
61 | 216~442 | Tamra |
| ucdCg-131 | F: GCACACGGATCTTGTGAAAA R: GCCAAGCTTGCTTGTAGGTT |
55 | 173~251 | Hex |
| ucdCg-133 | F: GTTGGCGCGAAATATAGGAA R: TGAACTTGTGATGTGCAGGA |
55 | 222~366 | Fam |
| ucdCg-129 | F: CGAATTTTTCGGACATCGTT R: GTGGTATGCCTGCATCATGT |
57 | 204~283 | Tamra |
| ucdCg-119 | F: AGGATGCCAATCGATTTTATTT R: ACCATGCCGTCTTAGTGGAC |
50 | 180~301 | Hex |
| ucdCg-111 | F: TTTTCAACGGAATCTGAACAAA R: GGAAAATTGATGGATGAAACAGA |
60 | 129~217 | Fam |
| otgfa0_3837_C08 | F: AACTCACGACATGCTGTATCAATC R: ATGATAGAACCCTGGCTAGAAAGC |
60 | 148~235 | Tamra |
| ucd-Cg138 | F: CCTCGAACAGCACTCCAAAT R: TTCAGTTCAACGCTCTTGCT |
57 | 223~298 | Hex |
PCR扩增体系: 12.5 µL 2×Quick Taq HS DyeMix (TOYOBO), 10 µmol/L的正反向引物各1 µL, 100 ng/µL的基因组DNA 1 µL, 超纯水补齐至25 µL。PCR扩增程序: 94 ℃预变性2 min; 94 ℃变性30 s、Tm退火30 s、68 ℃延伸30 s, 共35个循环; 68 ℃延伸10 min。
取5 µL PCR产物进行琼脂糖凝胶电泳检测, 检测合格后进行荧光毛细管电泳, 使用GeneMapper 3.0软件对原始数据进行分型, 读取产物大小。
1.4 数据统计与分析根据3个选育群体和6个野生群体在21个微卫星位点的基因分型数据, 使用GenAlex软件计算21个微卫星位点的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、香农-维纳多样性指数(I)、近交系数(Fis)、遗传分化指数(Fst)和基因流(Nm), 以及9个长牡蛎群体的Na、Ne、I、Ho和He。使用PowerMarker 3.25计算各微卫星位点和各群体的多态信息含量(PIC)。使用GenAlex软件检验哈代-温伯格平衡, 进行分子变异方差分析(AMOVA), 计算群体间的Fst和Nm、Nei氏标准遗传距离和遗传相似系数。使用POPULATIONS 1.2.32软件计算遗传距离并绘制UPGMA聚类树。
2 结果 2.1 21个微卫星位点的遗传多样性分析21个微卫星位点的遗传多样性参数见表 3。21个微卫星位点共检测出了460个Na, ucdCg-111位点最少为14个, ucd-Cg177、ucd-Cg157位点最多为27个, 平均每个位点有21.905个; Ne为7.135~19.455, 平均值为13.781; Ho在0.419~0.959之间, 平均值为0.751; He为0.847~0.936, 平均值为0.909; 在各位点检测到的I为2.176~3.025, 平均值为2.748; PIC为0.885~0.979, 平均值为0.953; 各位点的Fst值为0.033~0.076, 平均值为0.048; 21个微卫星位点除ucd-Cg109 (Fis =–0.001)和CgGJ196 (Fis =–0.034)外, 其他各位点的Fis均为正值; 各位点的Nm为3.039~ 7.387, 平均值为5.167。
| 位点 | Na | Ne | Ho | He | I | PIC | Fis | Fst | Nm |
| ucd-Cg109 | 21 | 13.481 | 0.914 | 0.914 | 2.735 | 0.952 | –0.001 | 0.041 | 5.839 |
| ucd-Cg140 | 23 | 14.643 | 0.646 | 0.886 | 2.742 | 0.959 | 0.270 | 0.076 | 3.039 |
| ucd-Cg177 | 27 | 17.383 | 0.863 | 0.934 | 3.025 | 0.975 | 0.076 | 0.042 | 5.703 |
| CgGJ196 | 23 | 14.947 | 0.959 | 0.928 | 2.876 | 0.958 | –0.034 | 0.033 | 7.244 |
| ucd-Cg149 | 26 | 17.378 | 0.762 | 0.936 | 3.001 | 0.976 | 0.185 | 0.041 | 5.860 |
| CgGJ202 | 24 | 15.082 | 0.805 | 0.929 | 2.888 | 0.961 | 0.134 | 0.033 | 7.387 |
| ucd-Cg194 | 18 | 10.541 | 0.553 | 0.896 | 2.545 | 0.941 | 0.383 | 0.050 | 4.739 |
| ucd-Cg160 | 17 | 10.574 | 0.419 | 0.876 | 2.497 | 0.933 | 0.522 | 0.056 | 4.197 |
| CgGJ330 | 15 | 8.321 | 0.672 | 0.865 | 2.308 | 0.903 | 0.223 | 0.049 | 4.899 |
| ucdCg-156 | 22 | 11.126 | 0.891 | 0.901 | 2.695 | 0.945 | 0.010 | 0.048 | 4.952 |
| ucd-Cg162 | 23 | 14.020 | 0.729 | 0.918 | 2.819 | 0.955 | 0.206 | 0.041 | 5.842 |
| ucdCg-126 | 20 | 12.558 | 0.726 | 0.906 | 2.683 | 0.956 | 0.199 | 0.055 | 4.335 |
| ucd-Cg175 | 23 | 13.331 | 0.884 | 0.907 | 2.772 | 0.951 | 0.025 | 0.048 | 5.013 |
| ucd-Cg157 | 27 | 19.455 | 0.632 | 0.931 | 3.025 | 0.977 | 0.321 | 0.047 | 5.025 |
| ucdCg-131 | 21 | 13.610 | 0.665 | 0.908 | 2.721 | 0.958 | 0.268 | 0.053 | 4.509 |
| ucdCg-133 | 26 | 17.607 | 0.816 | 0.929 | 2.970 | 0.979 | 0.121 | 0.051 | 4.632 |
| ucdCg-129 | 20 | 13.463 | 0.685 | 0.908 | 2.698 | 0.965 | 0.245 | 0.060 | 3.915 |
| ucdCg-119 | 23 | 15.319 | 0.761 | 0.922 | 2.847 | 0.970 | 0.174 | 0.050 | 4.722 |
| ucdCg-111 | 14 | 7.135 | 0.755 | 0.847 | 2.176 | 0.885 | 0.108 | 0.048 | 4.988 |
| otgfa0_3837_C08 | 24 | 15.128 | 0.781 | 0.926 | 2.883 | 0.961 | 0.156 | 0.037 | 6.487 |
| ucd-Cg138 | 23 | 14.290 | 0.862 | 0.917 | 2.810 | 0.961 | 0.061 | 0.046 | 5.167 |
| 平均值 | 21.905 | 13.781 | 0.751 | 0.909 | 2.748 | 0.953 | 0.174 | 0.048 | 5.167 |
9个长牡蛎群体的遗传多样性参数见表 4。9个长牡蛎群体共检测出198个Na, 其中, LY2-K11群体最少为13, LS群体和KTD群体最多为29; Ne为7.223~19.306, 其中最低的是LY2-K11群体, 最高的是LS群体; I处于2.128~3.112, 最低的是LY2-K11群体, 最高的是KTD群体; Ho在0.727~0.774之间, 最低的群体是LY2-K11, 最高的是HD; He为0.831~ 0.941, 最低的是LY2-K11群体, 最高的是LS群体和KTD群体; PIC为0.825~0.938, 最低的是LY2-K11群体, 最高的是KTD群体, 9个群体的PIC均大于0.5。
| 群体 | Na | Ne | I | Ho | He | PIC |
| LY2-K7 | 24 | 15.437 | 2.912 | 0.761 | 0.929 | 0.920 |
| LY2-K11 | 13 | 7.223 | 2.128 | 0.727 | 0.831 | 0.825 |
| LY2-K4 | 25 | 16.195 | 2.959 | 0.768 | 0.930 | 0.927 |
| QHD | 18 | 10.612 | 2.585 | 0.739 | 0.901 | 0.894 |
| LS | 29 | 19.306 | 3.107 | 0.750 | 0.941 | 0.937 |
| HD | 14 | 8.100 | 2.284 | 0.774 | 0.867 | 0.856 |
| ZH | 25 | 16.486 | 2.960 | 0.733 | 0.933 | 0.920 |
| WD | 21 | 11.770 | 2.689 | 0.751 | 0.905 | 0.900 |
| KTD | 29 | 18.897 | 3.112 | 0.761 | 0.941 | 0.938 |
对189个群体位点组合(9个群体×21个位点)进行哈代-温伯格平衡检测(表 5), 共发现125个组合不符合哈代-温伯格平衡, 其中选育群体有38个组合, 野生群体有87个组合。ucd-Cg194、ucd-Cg160、ucd-Cg162和ucd-Cg157位点的所有群体均不符合哈代-温伯格平衡。
| 位点 | LY2-K7 | LY2-K11 | LY2-K4 | QHD | LS | HD | ZH | WD | KTD |
| ucd-Cg109 | 0.103 | 0.318 | 0.668 | 0.048* | 0.945 | 0.508 | 0.009** | 0.644 | 0.363 |
| ucd-Cg140 | 0.000** | 0.068 | 0.010* | 0.204 | 0.000** | 0.000** | 0.000** | 0.001** | 0.144 |
| ucd-Cg177 | 0.377 | 0.766 | 0.002** | 0.795 | 0.485 | 0.603 | 0.000** | 0.000** | 0.011* |
| CgGJ196 | 0.309 | 0.055 | 0.085 | 0.176 | 0.004** | 0.367 | 0.178 | 0.319 | 0.808 |
| ucd-Cg149 | 0.001** | 0.000** | 0.000** | 0.038* | 0.055 | 0.047* | 0.007** | 0.115 | 0.003** |
| CgGJ202 | 0.459 | 0.791 | 0.063 | 0.000** | 0.389 | 0.001** | 0.078 | 0.001** | 0.003** |
| ucd-Cg194 | 0.000** | 0.001** | 0.000** | 0.000** | 0.000** | 0.000** | 0.000** | 0.000** | 0.000** |
| ucd-Cg160 | 0.000** | 0.000** | 0.000** | 0.000** | 0.000** | 0.000** | 0.000** | 0.000** | 0.000** |
| CgGJ330 | 0.000** | 0.033* | 0.004** | 0.008** | 0.000** | 0.000** | 0.061 | 0.368 | 0.002** |
| ucdCg-156 | 0.103 | 0.915 | 0.005** | 0.992 | 0.016* | 0.625 | 0.010* | 0.007** | 0.049* |
| ucd-Cg162 | 0.047* | 0.001** | 0.007** | 0.000** | 0.000** | 0.002** | 0.001** | 0.000** | 0.000** |
| ucdCg-126 | 0.081 | 0.026* | 0.015* | 0.007** | 0.001** | 0.001** | 0.002** | 0.000** | 0.000** |
| ucd-Cg175 | 0.080 | 0.061 | 0.000** | 0.572 | 0.807 | 0.869 | 0.010* | 0.966 | 0.351 |
| ucd-Cg157 | 0.000** | 0.000** | 0.004** | 0.001** | 0.001** | 0.003** | 0.000** | 0.000** | 0.000** |
| ucdCg-131 | 0.002** | 0.000** | 0.000** | 0.000** | 0.031* | 0.014* | 0.000** | 0.000** | 0.255 |
| ucdCg-133 | 0.005** | 0.001** | 0.029* | 0.000** | 0.000** | 0.155 | 0.064 | 0.000** | 0.064 |
| ucdCg-129 | 0.003** | 0.211 | 0.017* | 0.000** | 0.000** | 0.018* | 0.001** | 0.000** | 0.066 |
| ucdCg-119 | 0.056 | 0.748 | 0.000** | 0.000** | 0.001** | 0.000** | 0.015* | 0.000** | 0.047* |
| ucdCg-111 | 0.001** | 0.746 | 0.178 | 0.006** | 0.459 | 0.003** | 0.000** | 0.017* | 0.000** |
| otgfa0_3837_C08 | 0.276 | 0.000** | 0.139 | 0.000** | 0.267 | 0.094 | 0.000** | 0.002** | 0.024* |
| ucd-Cg138 | 0.002** | 0.000** | 0.286 | 0.105 | 0.049* | 0.722 | 0.002** | 0.272 | 0.119 |
| 注: *P < 0.05表示差异显著; **P < 0.01表示差异极显著 | |||||||||
9个长牡蛎群体间的遗传分化指数Fst和基因流Nm见表 6。9个野生群体的Fst范围在0.012~0.064, 处于中等偏低的遗传分化水平。其中, LY2-K11群体与QHD群体、HD群体、WD群体间处于中等遗传分化水平(0.05≤Fst < 0.15), 其余群体间均处于较低遗传分化水平(Fst < 0.05)。9个长牡蛎群体除LY2-K11群体和HD群体间的Nm < 4 (3.656)外, 其他群体间的Nm均 > 4 (4.357~20.158), 各个群体间的基因流较强。
| 群体 | LY2-K7 | LY2-K11 | LY2-K4 | QHD | LS | HD | ZH | WD | KTD |
| LY2-K7 | 5.805 | 17.320 | 9.970 | 15.369 | 7.320 | 14.260 | 10.061 | 16.158 | |
| LY2-K11 | 0.041 | 6.814 | 4.357 | 5.380 | 3.656 | 5.364 | 4.756 | 5.525 | |
| LY2-K4 | 0.014 | 0.035 | 10.327 | 14.524 | 7.564 | 13.699 | 10.432 | 15.804 | |
| QHD | 0.024 | 0.054 | 0.024 | 11.248 | 14.254 | 10.886 | 8.779 | 10.958 | |
| LS | 0.016 | 0.044 | 0.017 | 0.022 | 7.988 | 16.552 | 10.065 | 20.158 | |
| HD | 0.033 | 0.064 | 0.032 | 0.017 | 0.030 | 7.812 | 7.225 | 7.848 | |
| ZH | 0.017 | 0.045 | 0.018 | 0.022 | 0.015 | 0.031 | 10.717 | 16.828 | |
| WD | 0.024 | 0.050 | 0.023 | 0.028 | 0.024 | 0.033 | 0.023 | 10.614 | |
| KTD | 0.015 | 0.043 | 0.016 | 0.022 | 0.012 | 0.031 | 0.015 | 0.023 |
群体的AMOVA分析结果见表 7。3%的变异来自于群体间, 19%来自于个体间, 77%来自于个体内, 变异主要来自于个体内。
| 变异来源 | 自由度 | 平方和 | 方差 | 百分率 |
| 群体间 | 8 | 262.404 | 32.800 | 3% |
| 个体间 | 260 | 3 043.741 | 11.707 | 19% |
| 个体内 | 269 | 2 090.500 | 7.771 | 77% |
| 总变异 | 537 | 5 396.645 | — | 100% |
9个长牡蛎群体的Nei氏标准遗传距离和遗传相似系数见表 8。9个群体的Nei氏标准遗传距离在0.300~1.420之间, HD群体和QHD群体的遗传距离最近为0.300, HD群体和LY2-K11群体的遗传距离最远为1.420。9个长牡蛎群体的遗传相似系数在0.242~0.741之间, HD群体和LY2-K11群体的遗传相似系数最小为0.242, HD群体和QHD群体的遗传系数最大为0.741。
| 群体 | LY2-K7 | LY2-K11 | LY2-K4 | QHD | LS | HD | ZH | WD | KTD |
| LY2-K7 | 0.417 | 0.619 | 0.464 | 0.535 | 0.418 | 0.526 | 0.457 | 0.556 | |
| LY2-K11 | 0.875 | 0.517 | 0.278 | 0.334 | 0.242 | 0.353 | 0.334 | 0.356 | |
| LY2-K4 | 0.480 | 0.659 | 0.480 | 0.505 | 0.436 | 0.505 | 0.474 | 0.543 | |
| QHD | 0.768 | 1.280 | 0.733 | 0.496 | 0.741 | 0.499 | 0.470 | 0.481 | |
| LS | 0.626 | 1.096 | 0.684 | 0.702 | 0.445 | 0.552 | 0.420 | 0.604 | |
| HD | 0.872 | 1.420 | 0.830 | 0.300 | 0.809 | 0.449 | 0.468 | 0.434 | |
| ZH | 0.642 | 1.041 | 0.683 | 0.694 | 0.594 | 0.800 | 0.480 | 0.559 | |
| WD | 0.783 | 1.097 | 0.747 | 0.756 | 0.869 | 0.759 | 0.733 | 0.450 | |
| KTD | 0.587 | 1.034 | 0.610 | 0.733 | 0.503 | 0.835 | 0.581 | 0.799 |
基于9个长牡蛎群体的遗传距离构建的UPGMA树(图 1)显示, 9个长牡蛎群体共聚为三支, LY2-K11群体单独聚为一支, QHD和HD群体聚为一支, 其他6个群体聚为一支。PCoA分析结果与UPGMA聚类结果一致, LY2-K11群体单独聚为一类, QHD和HD群体聚为一类, 其他6个群体聚为一类(图 2)。
 |
| 图 1 基于遗传距离构建的9个长牡蛎群体的UPGMA聚类树 Fig. 1 Bifurcation diagram by UPGMA cluster analysis |
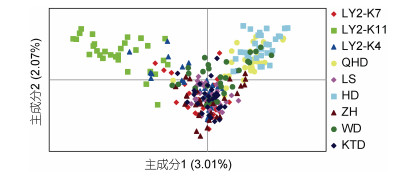 |
| 图 2 长牡蛎9个群体的PCoA分析 Fig. 2 PCoA analysis of 9 populations of C. gigas |
微卫星标记有共显性、易检测、多态性高、稳定性高、对DNA样品质量要求不高等特点, 已经被广泛应用于水产动物遗传变异检测。王九龙等(2023)选用20个中高多态性(PIC为0.361~0.910)微卫星位点比较了绿鳍马面鲀野生群体和养殖群体的遗传多样性, 发现养殖群体的遗传多样性低于野生群体, 但仍维持较高的遗传多样性水平。余成晨等(2022)选择15个高多态性(PIC均大于0.5)微卫星标记对草鱼长江选育群体各世代进行了遗传特征分析, 发现各世代遗传多样性均较高, 世代间的遗传分化水平较低。本研究选取21个微卫星位点对长牡蛎的3个选育群体和6个野生群体进行遗传多样性和遗传结构分析, 发现平均每个位点的Na为21.905、Ne为13.781、Ho为0.751、He为0.909、PIC为0.953, 21个微卫星位点的PIC均大于0.5, 具有高度遗传多态性, 可应用于本研究9个长牡蛎群体的遗传多样性分析, 研究结果将为长牡蛎群体遗传变异监测提供高质量的数据。
3.2 群体遗传多样性Na、Ne、I、Ho、He和PIC是评价群体遗传多样性的重要参数, 与遗传多样性呈正相关。一般认为Na越接近Ne的绝对值说明群体等位基因分布越均匀(周康奇等, 2023), 本研究9个长牡蛎群体Na均大于Ne (约1.5倍), 说明这9个群体都存在等位基因分布不均的现象, 不均匀程度依次为LY2-K11 > WD > HD > QHD > LY2-K7 > LY2-K4 > KTD > ZH > LS, 这种现象在中华圆田螺(周康奇等, 2023)和罗氏沼虾(李景芬等, 2020)等物种中也存在。
I主要反映群体遗传丰富度和均匀度(周姝婧等, 2022)。中国鲎9个群体的平均I值为1.39, 说明中国海域中国鲎种群有较高的遗传多样性(翁朝红等, 2020); 广西中华圆田螺的I值为0.412~1.226, 说明广西境内的中华圆田螺遗传多样性较高(周康奇等, 2023)。本研究中, 3个选育群体和6个野生群体I值在2.128~3.112, 说明选育群体和野生群体均有较高的遗传多样性。与Ho相比, He受样品数量影响较小, 更适合用来评估群体遗传多样性。He数值越大, 群体遗传多样性越高(王世会等, 2022)。14个黄颡鱼群体的He值为0.358~0.749, 遗传多样性较高, 有较大的选育潜力(李燕平等, 2023)。本研究分析发现9个群体的He值为0.831~0.941, 说明这些群体均有较高的遗传多样性。9个群体的He均大于Ho, 推测这些群体可能存在杂合子缺失, 造成这种现象的原因可能有稀有纯合基因型的发生、哑等位基因、人为定向选育和种群内交等(赵斌丽等, 2009)。董在杰等(2018)对6个鲤群体及于爱清等(2019)对长江刀鲚的研究中均发现了这一现象。PIC < 0.25为低度多态性, 0.25≤ PIC < 0.5为中度多态性, PIC≥0.5为高度多态性(田间等, 2023)。本研究中9个群体的PIC值均大于0.5, 都为高度多态性。PIC值的分析结果与I值和He值的分析结果基本一致, 三个选育群体的遗传多样性均较高, 且LY2-K4群体 > LY2-K7群体 > LY2-K11群体。造成这种现象的原因可能为选育过程中雌雄亲贝均在60个以上, 亲本数量较多, 使得多代选育后选育群体的遗传多样性仍处于较高水平。LY2-K11群体的遗传多样性最低, 可能是因为与LY2-K4和LY2-K7群体相比LY2-K11群体的选育代数更多(5代), 另外, LY2-K7群体亲本来源于HD和ZH群体, LY2-K4群体亲本来源于KTD和WD群体, 而LY2-K11群体亲本只来源于KTD群体, 亲本来源单一可能会加快遗传多样性降低的速度。
哈代-温伯格平衡是指在理想情况下, 群体中各基因频率和基因型频率在世代遗传过程中保持不变(宋立民等, 2023)。本研究中各群体均有位点偏离了哈代-温伯格平衡, 说明这9个群体可能有纯合子过量的情况, 与Ho、He结果一致。在团头鲂、黄颡鱼和文蛤等物种中也有类似情况(田镇等, 2021; 宋立民等, 2023; 张芹等, 2023)。
3.3 长牡蛎群体遗传结构和遗传分化Fst < 0.05表明群体间的遗传分化水平低, 0.05 < Fst < 0.15表明群体间处于中等遗传分化水平, 0.15 < Fst < 0.25表明群体间遗传分化水平高(李燕平等, 2023)。本研究中9个群体间的Fst值为0.012~0.064, 除了LY2-K11群体与QHD群体、HD群体和WD群体间处于中等遗传分化水平外, 其他群体间处于较低的遗传分化水平。基因流是引起群体间遗传分化水平低的重要原因, 一般认为Nm < 1表明可能有隔离产生, Nm > 1表示遗传分化不显著, Nm > 4说明遗传分化程度很小(范士琦等, 2023)。本研究中除LY2-K11群体和HD群体间的Nm < 4 (3.656)外, 其他群体间的Nm均 > 4 (4.357~20.158), 表明各个群体间基因交流频繁, 遗传分化水平较低。AMOVA分析表明长牡蛎以个体内的遗传变异为主(77%), 群体间的遗传分化较少(3%), 也说明9个长牡蛎群体间的遗传分化水平较低。
基于Nei氏方法计算的标准遗传距离反映群体间的基因差异程度, 相似系数反映群体间的亲缘关系, 二者呈反比(宋立民等, 2023)。本研究群体间的遗传距离为0.300~1.420, 遗传相似系数为0.242~0.741, HD群体和QHD群体的遗传距离最近(0.300), 遗传相似系数最大(0.741); LY2-K11群体与其他群体的遗传距离均较远(0.659~1.420), 遗传相似系数均较小(0.242~0.517)。UPGMA聚类树的结果与遗传距离结果一致, QHD群体和HD群体聚为一个分支, LY2-K11群体单独聚为一个分支, 其他6个群体聚为一个分支。PCoA结果也显示LY2-K11群体单独聚为一类, QHD和HD群体聚为一类, 其他6个群体聚为一类。相对较大的遗传距离和遗传分化来自于选育群体LY2-K11和其他群体间, 说明人工选育可能会影响野生群体和选育群体间的基因流, 进而影响遗传分化。6个野生长牡蛎群体分别采集于渤海、黄海的不同水域, 在地理位置上有很大差异, 但是群体间的遗传距离和遗传分化较小, 这可能是因为洋流造成的浮游期幼虫扩散或苗种交易造成的较频繁的基因交流。LY2-K7群体和LY2-K4群体的遗传分化较小, 可能是因为这两个群体都是以壳长为指标选育的, 对目标性状的定向选择可能造成了与性状连锁的微卫星标记的定向选择, 使得二者的遗传变异方向趋向一致。
4 结论综上所述, 本研究利用21个高度多态微卫星位点评估了长牡蛎6个野生群体和3个选育群体的遗传多样性和遗传结构。发现9个群体均处于较高的遗传多样性水平, 经过选育LY2-K11群体遗传多样性略有下降, 在后续的育种过程中应尽量选用多种来源、遗传距离较远的亲贝, 避免因近交衰退造成遗传多样性降低, 苗种抗逆性变差。
于爱清, 施永海, 邓平平, 2019. 长江刀鲚选育和野生群体遗传多样性的微卫星分析[J]. 水产科技情报, 46(3): 121-125. |
王九龙, 叶苗, 李洪莉, 等, 2023. 绿鳍马面鲀野生与养殖群体的微卫星遗传多样性分析[J/OL]. 水产科学: 1-9. (2023-06-01).
https://doi.org/10.16378/j.cnki.1003-1111.21165.
|
王世会, 罗亮, 张瑞, 等, 2022. 绥芬河河蟹种质遗传多样性参数优化[J]. 水产学杂志, 35(5): 18-26. |
田间, 马行空, 曾健成, 等, 2023. 基于微卫星标记的中华绒螯蟹遗传多样性分析[J]. 水产养殖, 44(5): 20-25. |
田镇, 陈爱华, 曹奕, 等, 2021. 红壳色文蛤选育群体遗传多样性的微卫星分析[J]. 南方农业学报, 52(9): 2582-2589. |
任晶莹, 王卫军, 徐涛, 等, 2018. 长牡蛎(Crassostrea gigas)多代近交与自交家系遗传差异及生长性状比较[J]. 海洋与湖沼, 49(3): 630-637. |
孙成飞, 2020. 翘嘴鳜遗传图谱构建与"广清1号"选育过程各世代遗传参数与遗传多样性评估[D]. 上海: 上海海洋大学.
|
李景芬, 夏正龙, 栾生, 等, 2020. 五个罗氏沼虾群体遗传多样性的微卫星分析[J]. 水生生物学报, 44(6): 1208-1214. |
李燕平, 覃川杰, 吕云云, 等, 2023. 黄颡鱼养殖群体遗传多样性与遗传结构的微卫星分析[J]. 淡水渔业, 53(3): 3-14. |
余成晨, 张伟, 徐晓雁, 等, 2022. 草鱼选育群体不同世代遗传变异的微卫星分析[J]. 中国水产科学, 29(8): 1109-1118. |
余勇, 2015. 长牡蛎不同选育群体杂交子代的杂种优势及微卫星分析[D]. 青岛: 中国海洋大学.
|
宋立民, 王娜, 郑英珍, 等, 2023. 基于微卫星标记技术的5个黄颡鱼群体遗传多样性分析[J]. 经济动物学报, 27(2): 101-108. |
张芹, 王延晖, 王冰柯, 等, 2023. 基于基因组微卫星的团头鲂养殖群体遗传多样性分析和指纹图谱构建[J]. 安徽农业科学, 51(8): 99-104. |
张荣良, 王卫军, 冯艳微, 等, 2016. 长牡蛎(Crassostrea gigas). 壳宽快速生长选育群体遗传多样性及遗传结构的微卫星标记分析[J]. 渔业科学进展, 37(4): 90-96. |
范士琦, 冯婧昀, 苗晓敏, 等, 2023. 重庆养殖场鳜群体微卫星遗传多样性研究[J]. 水产养殖, 44(7): 18-23. |
周姝婧, 朱翔杰, 徐新建, 等, 2022. 样本量与东方蜜蜂微卫星DNA遗传多样性参数稳定性的关系[J]. 福建农林大学学报(自然科学版), 51(5): 682-689. |
周康奇, 李育森, 林勇, 等, 2023. 广西地区中华圆田螺不同地理种群遗传多样性分析[J/OL]. 广西科学: 1-13. (2023-07-13). https://doi.org/10.13656/j.cnki.gxkx.20230712.001.
|
赵斌丽, 黎中宝, 陈锦, 等, 2009. 日本囊对虾野生群体杂合性的研究[J]. 集美大学学报(自然科学版), 14(2): 21-26. |
翁朝红, 谢仰杰, 肖志群, 等, 2020. 中国鲎(Tachypleus tridentatus)微卫星标记筛选及种群遗传多样性和遗传结构分析[J]. 海洋与湖沼, 51(2): 345-353. |
高杨, 田灿, 姜京京, 等, 2023. 基于微卫星标记的克氏原螯虾种群遗传多样性和遗传结构分析[J]. 江苏农业科学, 51(5): 191-199. |
梁园, 付敬强, 沈铭辉, 等, 2022. 方斑东风螺3个选育世代遗传多样性和遗传结构的微卫星分析[J]. 海洋科学, 46(10): 85-93. |
董在杰, 刘念, 傅建军, 等, 2018. 6个野生与选育鲤群体的微卫星遗传分析[J]. 南方水产科学, 14(4): 46-55. |
滕爽爽, 胡高宇, 范建勋, 等, 2021. 缢蛏5个群体遗传多样性和遗传分化的SNP分析[J]. 水生生物学报, 45(4): 861-870. |
LI G, HUBERT S, BUCKLIN K, et al, 2003. Characterization of 79 microsatellite DNA markers in the Pacific oyster Crassostrea gigas[J]. Molecular Ecology Notes, 3(2): 228-232. DOI:10.1046/j.1471-8286.2003.00406.x |
LI Q, YU H, YU R H, 2006. Genetic variability assessed by microsatellites in cultured populations of the Pacific oyster (Crassostrea gigas) in China[J]. Aquaculture, 259(1/2/3/4): 95-102. |
QI H G, WU Q, LI L, et al, 2009. Development and characterization of microsatellite markers for the Pacific Oyster Crassostrea gigas[J]. Conservation Genetics Resources, 1(1): 451-453. DOI:10.1007/s12686-009-9104-x |
ZHONG X X, FENG D D, YU H, et al, 2016. Genetic variation and breeding signature in mass selection lines of the Pacific oyster (Crassostrea gigas) assessed by SNP markers[J]. PLoS One, 11(3): e0150868. DOI:10.1371/journal.pone.0150868 |
 2024, Vol. 55
2024, Vol. 55


