
中国海洋湖沼学会主办。
文章信息
- 刘丹阳, 孔杰, 王平, 陈荣坚, 傅强, 罗坤, 陈宝龙, 隋娟, 孟宪红, 代平, 谭建, 曹家旺, 李旭鹏, 康子仪, 刘绵宇, 强光峰, 迟长凤, 栾生. 2024.
- LIU Dan-Yang, KONG Jie, WANG Ping, CHEN Rong-Jian, FU Qiang, LUO Kun, CHEN Bao-Long, SUI Juan, MENG Xian-Hong, DAI Ping, TAN Jian, CAO Jia-Wang, LI Xu-Peng, KANG Zi-Yi, LIU Mian-Yu, QIANG Guang-Feng, CHI Chang-Feng, LUAN Sheng. 2024.
- 利用“黄海芯1号”55K SNP芯片评估凡纳滨对虾选育群体的遗传多样性与基因组近交水平
- GENETIC DIVERSITY AND GENOMIC INBREEDING ANALYSIS IN TWO SELECTED POPULATIONS OF LITOPENAEUS VANNAMEI USING THE LIQUID CHIP OF "YELLOW SEA CHIP NO.1" (55K SNP)
- 海洋与湖沼, 55(2): 479-488
- Oceanologia et Limnologia Sinica, 55(2): 479-488.
- http://dx.doi.org/10.11693/hyhz20231000214
文章历史
-
收稿日期:2023-10-17
收修改稿日期:2024-01-11
2. 中国水产科学研究院黄海水产研究所 青岛海洋科技中心海洋渔业科学与食物产出过程功能实验室 农业农村部海洋渔业可持续发展重点实验室 山东青岛 266071;
3. 海南中正水产科技有限公司 海南东方 572633;
4. 广东海兴农集团有限公司 广东广州 511400
2. Key Laboratory for Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao Marine Science and Technology Center, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China;
3. Hainan Zhongzheng Technology Aquatic Products Co., Ltd., Dongfang 572633, China;
4. Guangdong Hisenor Group Co., Ltd., Guangzhou 511400, China
凡纳滨对虾(Litopenaeus vannamei)具有生长速度快、抗性强、盐度适应性广等优点, 是重要的水产养殖济物种之一, 据《中国渔业统计年鉴》统计, 2022年我国凡纳滨对虾海淡水养殖总产量为209万t, 居世界凡纳滨对虾养殖产量首位(农业农村部渔业渔政管理局等, 2023)。然而, 凡纳滨对虾作为引进养殖物种, 每年需从国外引进种虾, 由于国外种虾公司对种质的控制, 引进亲虾存在遗传背景不清晰、遗传多样性较低等问题。直接利用引进种虾留种、制种, 组建基础群体开展选育工作, 自繁后代易因近交产生性状衰退, 严重影响对虾养殖业的可持续发展。因此, 调查凡纳滨对虾现有育种群体的遗传多样性及近交水平, 对于长期育种计划实施具有重要的指导意义和实际价值。Casado等(2022)使用192个SNPs对6个商业群体的遗传结构、杂合度、多态性标记数量等参数进行了评估。Wang等(2022)基于重测序SNP数据对两个选育品系及四个商业品系的遗传分化水平进行了评估。Medrano-Mendoza等(2023)使用32K SNP标记对来自墨西哥的抗WSSV及AHPND的选育群体进行群体结构及遗传多样性分析。这些研究为凡纳滨对虾育种群体的遗传背景分析和选育潜力评估提供了参考。目前, 凡纳滨对虾种虾主要分为两个大类, 以墨西哥蓝色基因、美国API等为代表的具有较强的抗病、抗逆性的“高抗”品系; 以泰国正大、顶峰等为代表的具有快速生长特性的“快大”品系。然而, 目前尚未有针对”高抗”系和“快大”系选育群体遗传多样性的对比分析报道。育种群体的近交水平, 是制定配种方案和评估选育潜力时的重要参考依据。然而, 国内多数育种群体, 由于其奠基者群体的遗传背景不清楚、系谱缺失, 根据已有系谱信息难以准确地评估育种群体的近交水平。研究表明, 通过覆盖全因组的高通量SNP数据对育种群体的近交水平进行评估是一种更为可行的方案, 基因组近交系数的估计方法分为两大类: (1) 基于SNP位点等位基因频率的计算方法; (2) 基于连续性纯合片段(runs of homozygosity, ROH)占比的计算方法(Li et al, 1953; Nejati-Javaremi et al, 1997; Toro et al, 2002; Vanraden, 2008; Yang et al, 2010)。Caballero等(2022)等利用计算机模拟方法调查了多种基因组近交系数计算方法, 当基础群体的等位基因频率已知时, 6种基于等位基因频率的计算方法均获得了较高的准确性; 然而当基础群体等位基因频率未知时, 基于ROH的计算方法则有较高的准确性。使用ROH评估育种群体的近交系数已被证明是评估近交的有效工具(Keller et al, 2011)。个体基因组上ROH的长度及分布可用于评估其近交, 通过计算特定长度的ROH的占比可以反映群体在不同时期的近交水平。基于ROH的基因组近交计算方法不依赖于基础群体的等位基因频率, 其计算结果更接近于真实IBD (identify-by-descent, IBD) (Gengler et al, 2007; Wang, 2014), 即“血缘同源”(两个个体共有的等位基因来源于共同祖先)。目前, 在虹鳟(Oncorhynchus mykiss)、银鲑(Oncorhynchus kisutch)、梭鲈(Sander lucioperca)和太平洋潜泥蛤(Panopea generosa)、大菱鲆(Scophthalmus maximus)等群体中, 已有基于ROH的基因组近交评估研究(Straus et al, 2015; de Los Ríos-Pérez et al, 2017, 2022; Aramburu et al, 2020; Yoshida et al, 2020)。但针对凡纳滨对虾育种群体, 尚未发现上述不同基因组近交评估方法间的对比分析报道。
本研究以“高抗系”和“快大系”两个选育群体为研究对象, 利用团队自主研发的液相芯片“黄海芯1号”(55K SNP)对家系个体开展高通量基因分型, 分析育种群体的遗传结构以及群体内家系间的亲缘关系, 对比分析两个群体间的遗传多样性差异, 评估两个选育群体的ROH分布特征, 利用多种基因组近交系数计算方法评估两个选育群体的近交水平并进行对比分析。研究结果预期可为凡纳滨对虾育种群体的遗传多样性和近交评估提供重要的参考依据。
1 材料与方法 1.1 实验材料研究材料来源于两个选育群体, 第一个群体由具有较强的抗病、抗逆等特性的奠基者群体组成, 命名为GK, 来自广东湛江。第二个群体由具有生长速度快特性的奠基者群体组成, 命名为KD, 来自海南东方。各群体的家系数量和样本数量如表 1所示。其中, GK和KD群体均经历了3次选择, 系谱可向上追溯4代, 分别包含55个家系和30个家系, 基因分型样本数量分别为1 094尾和564尾虾。
| 群体 | 家系数量 | 样本数量 |
| GK | 55 | 1 094 |
| KD | 30 | 564 |
| 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 | ||
两个群体均使用自主研发的55K SNP液相芯片“黄海芯1号”进行基因分型, 原始基因型数据集包含56 214个SNP位点的信息。合并两个群体的基因型文件, 将scaffolds定位至44条连锁群, 删除无法定位的6 553个SNP标记。设置质控参数为: 次等位基因频率≥0.05、个体基因型缺失率 < 20%、标记基因型缺失率 < 10%, 最终用于分析的基因型数据集包含44 135个SNP位点, 1 658尾虾。
1.2.2 群体遗传结构及亲缘关系分析使用三种方法分析育种群体的遗传结构。(1) 使用plink进行主成分分析, 使用R包ggplot绘制PCA图。(2) 使用plink计算个体之间的遗传距离, 构建距离矩阵, 使用PHYLIP 3.69软件中的N-J法计算个体间距, 使用R包ggtree绘制系统进化树(NJ-tree)。(3) 根据Yang等(2011)的方法构建基因组亲缘关系矩阵, 使用R包pheatmap绘制亲缘关系热图。
1.2.3 遗传多样性分析使用plink计算观测杂合度(Ho)、期望杂合度(He)、多态性标记比例(Pn), 根据Botstein等(1980)的公式计算多态信息含量PIC, 使用vcftools计算遗传分化指数Fst。
1.2.4 基因组近交系数评估本研究使用9种方法计算2个群体的近交系数, 使用泊松相关计算不同方法之间的相关性。相关公式如下所示:
(1) 系谱近交系数, 使用asreml包进行计算, 表示为FPED;
(2) 基于加性遗传值的方差计算近交系数, 由VanRaden提出, 计算公式如下:
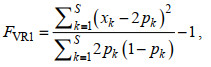 (1)
(1)式中, S为SNP标记总数, xk为标记k的次等位基因数量, pk为标记k在分析群体中的次等位基因频率。
(3) 在上式的基础上, 利用每个SNP位点的次等位基因频率的方差进行加权, 计算公式如下:
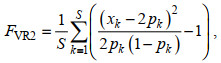 (2)
(2)与FVR1相比, 稀有纯合等位基因型对近交测定的贡献更大。
(4) 基于结合配子之间的相关进行计算, 同样利用每个SNP位点的次等位基因频率的方差进行加权, 但与上述方法相比, 具有更低的抽样方差, 计算公式如下:
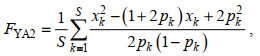 (3)
(3)(5) 在上式基础上提出, 在分子分母中分别对各位点计算结果求和, 计算公式如下:
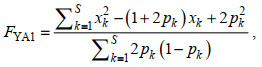 (4)
(4)(6) 根据纯合子观测频率与哈代温伯格平衡期望值的偏差进行度量, 计算公式如下:
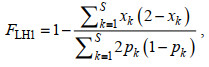 (5)
(5)(7) 在上式的基础上, 对每个SNP位点的等位基因频率的方差分别进行加权, 计算公式如下:
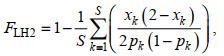 (6)
(6)(8) 根据纯合基因型SNP位点的比例计算, 公式如下:
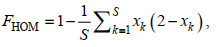 (7)
(7)(9) 基于ROH纯合片段长度占比计算近交系数, 公式如下:
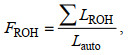 (8)
(8)式中, LROH是每条ROH的长度, Lauto是连锁群总长度, 在本研究中Lauto为1.428 Gb (连锁群LG1~LG44的总长度)。使用“plink --homozyg”指令对两个群体进行检测, 设置参数如下: --homozyg-snp 50 --homozyg-kb 800 --homozyg-gap 800 --homozyg-density 50 --homozyg-window-snp 50 --homozyg-window-het 1 --homozyg-window-missing 1 --homozyg-window-threshold 0.05。组成ROH的最低SNP数目根据以下公式进行计算:
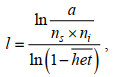 (9)
(9)式中, l为ROH所包含的最低SNP数目, a为假阳性, 本研究中设定为0.05; ns为SNP数目, ns=44 135; ni为样本数, ni=1 658; het为所有SNP平均杂合度, het= 0.34。经过计算l=50.79, 为了便于后续分析, 将l设置为50个SNP。
1.2.5 两个群体的ROH描述性统计及相关性分析本研究仅对可检测出ROH的样本进行ROH描述性统计及近交评估。对两个群体上ROH片段的总数目及总长度进行统计, 对每个个体上的ROH长度均值、ROH数量均值进行统计, 分别对两个群体在连锁群上检测到ROH的样本比例进行统计。根据长度将ROH片段划分为4个类别, 0.8~1.25 Mb、1.25~ 1.75 Mb、1.75~2.25 Mb、> 2.25 Mb, ROH长度与世代数之间的关系(Curik et al, 2014)可近似表示为: ROH长度=100/2g, 其中g代表世代数。上述四个类别可近似反映60~40代、40~28代、28~22代和22个世代之后的近交历史。基于4种长度的ROH以及全部ROH, 分别计算FROH。
使用R包“GGally”计算不同基因组近交系数之间的相关性, 并绘制相关性散点图。
2 结果 2.1 遗传结构及亲缘关系两个群体主成分分析如图 1所示, GK和KD群体明显分离, 同一群体内个体聚集在一起, 主成分图中前两个成分分别解释了总方差的31.29%和6.77%, 表明该主成分分析的结果可靠性较高。
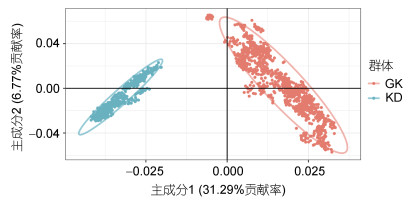 |
| 图 1 凡纳滨对虾两个选育群体的主成分分析 Fig. 1 The principle component analysis for two selected populations of L. vannamei 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 |
两个群体的系统发育树如图 2a所示, 图 2b为GK和KD两个群体各自选取2个家系(每个家系约10尾虾)绘制成的进化树局部图。个体的命名规则为“群体家系编号_个体编号”, 例如: GK01_1。由图 2可知, 两个群体中相同家系的个体均均聚集在一起, 家系之间区分明确, 其次, GK和KD两大群体区分明确。系统进化树呈现的群体结构与主成分图高度一致。两群体基于G矩阵绘制的亲缘关系热图如图 3所示, 亲缘关系热图与PCA及进化树高度匹配, 可分为两个群体, 两个群体间无亲缘关系, 与GK相比, KD个体间的亲缘关系更近。
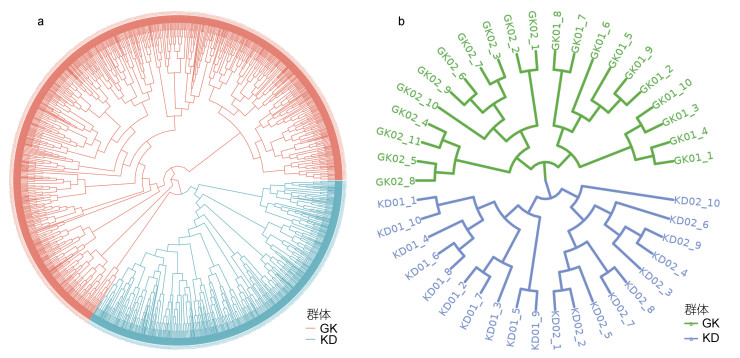 |
| 图 2 凡纳滨对虾两个选育群体的N-J系统进化树及部分家系个体展示 Fig. 2 The phylogenetic tree for two selected populations of L. vannamei 注: “GK”代表高抗选育群体, “KD”代表快大选育群体。a. GK和KD全部个体的系统进化树; b. GK和KD各选取两个家系的局部系统进化树 |
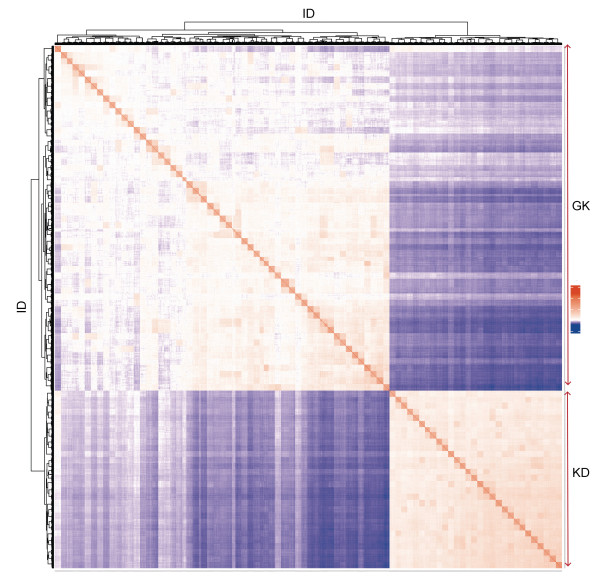 |
| 图 3 凡纳滨对虾两个选育群体的亲缘关系热图 Fig. 3 The genomic relationship heatmap for two selected populations of L. vannamei 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 |
对GK及KD群体的遗传多样性参数进行统计, 结果如表 2所示。GK和KD群体的Ho均低于He, 表明群体选育过程中, 遗传变异有所下降。GK和KD群体的PIC均值分别是0.28和0.26。GK和KD群体的多态性标记比例(Pn)分别是0.97和0.87。GK群体的杂合度总体高于KD群体, 表明GK群体具有更高的遗传多样性。根据Weir和Cockerman的标准(Weir et al, 1984), GK和KD群体的Fst为0.09, 在0.05~0.15之间, 为中等程度的遗传分化。
| 群体 | Ho | He | Pn | PIC |
| GK | 0.35 | 0.36 | 0.97 | 0.28 |
| KD | 0.32 | 0.33 | 0.87 | 0.26 |
| 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 | ||||
如表 3所示, 本研究中, GK群体有393个样本检测出了ROH, KD群体有312个样本检测出了ROH, 后续仅对这705个样本进行ROH统计分析及多种近交系数的对比分析。GK群体一共检测到780个ROH, 样本上单个ROH的平均长度为(1.70±0.34) Mb, 变化范围在0.81~2.92 Mb; 每个样本的ROH数目平均为(1.98±1.30)个, 范围在1~9个; 每个样本的全部ROH的长度均值为(3.41±1.30) Mb, 变化范围在0.81~ 14.87 Mb。KD群体共计检测到647个ROH, 样本上单个ROH的平均长度是(1.65±0.38) Mb, 变化范围是0.82~3.03 Mb; 每个样本的ROH数目平均为(2.07± 1.37)个, 范围在1~8个; 每个样本的全部ROH的长度均值为(3.46±1.37) Mb, 变化范围在0.82~13.97 Mb。
| 群体 | 检测出ROH的样本数 | ROH总数 | ROH平均长度/Mb | ROH平均数量/样本 | ROH总长度均值/样本/(Mb) | |||||
| 均值±标准差 | 范围 | 均值±标准差 | 范围 | 均值±标准差 | 范围 | |||||
| GK | 393 | 780 | 1.70±0.34 | 0.81~2.92 | 1.98±1.30 | 1~9 | 3.41±1.30 | 0.81~14.87 | ||
| KD | 312 | 647 | 1.65±0.38 | 0.82~3.03 | 2.07±1.37 | 1~8 | 3.46±1.37 | 0.82~13.97 | ||
| 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 | ||||||||||
图 4展示了GK和KD群体在每个连锁群上检测到ROH的样本比例, GK和KD群体在44个连锁群上检测到的样本比例均值分别为5.33%和4.77%, 前者高于后者。此外, ROH在连锁群上分布并不均匀, 其中GK和KD群体在LG31上检测到的样本比例最高, 分别是29.49%和26.97%。两个群体在连锁群上分布并不十分一致, 两个群体样本比例的相关性为0.06, 在LG18上, GK的样本比例约为KD的三倍, 在LG34、LG37连锁群上, GK的样本比例约为KD的两倍, 而在LG20上, GK的样本比例仅为KD的1/3。两群体在LG2、LG4、LG5、LG6、LG7、LG8等连锁群上的样本比例非常低, 接近于0。
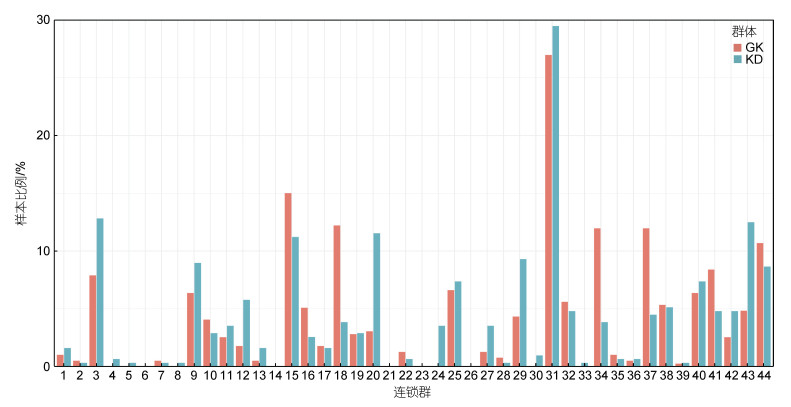 |
| 图 4 凡纳滨对虾两个选育群体ROH在连锁群上的样本比例 Fig. 4 Proportion of samples included ROH in linkage groups for two selected populations of L. vannamei 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 |
凡纳滨对虾两个选育群体不同长度ROH数量占比如表 4所示。在GK和KD两个群体中, 长度为1.25~1.75和1.75~2.25的ROH数量占比最高, 分别为78.34%和71%; 长度为0.8~1.25的ROH数量占比分别为11.41%和19.17%, 后者明显高于前者; 长度 > 2.25 Mb的ROH数量占比最少, 分别为10.26%和9.74%。
| ROH长度/Mb | GK | KD | |||
| 数量 | 比例/% | 数量 | 比例/% | ||
| 0.8~1.25 | 89 | 11.41 | 124 | 19.17 | |
| 1.25~1.75 | 351 | 45.13 | 267 | 41.27 | |
| 1.75~2.25 | 259 | 33.21 | 193 | 29.83 | |
| > 2.25 | 80 | 10.26 | 63 | 9.74 | |
| 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 | |||||
使用不同计算方法得到的GK和KD群体的基因组近交系数如表 5所示。除FVR2外, 对于其他8种基因组近交系数, KD群体的估计值均高于GK群体。两群体的FROH差异较小, KD群体略高于GK群体。KD群体的近交水平整体高于GK群体。
| 基因组近交系数 | 群体 | |
| GK | KD | |
| Fped | 0 | 0.004 |
| FVR1 | 0.071±0.035 | 0.099±0.036 |
| FVR2 | 0.070±0.059 | 0.026±0.031 |
| FYA1 | 0.066±0.034 | 0.119±0.043 |
| FYA2 | 0.053±0.040 | 0.078±0.047 |
| FLH1 | 0.062±0.038 | 0.139±0.055 |
| FLH2 | 0.032±0.061 | 0.126±0.083 |
| FHOM | 0.662±0.014 | 0.690±0.020 |
| FROH | 2.39E-03±1.64E-03 | 2.42E-03±1.70E-03 |
| 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 | ||
凡纳滨对虾两个群体8种基因组近交系数两两间的相关性如图 5所示。图中上三角部分为相关系数及其显著水平, 下三角部分为相关性散点趋势图, 对角线为两个群体不同近交系数的直方图。图中第一行为群体的样本量, GK和KD分别包含393和312个样本, 第一列为频数直方图, 表示两个群体不同近交数的分布比例。其中, GK及KD群体的FLH1与FHOM的相关系数最高(R=1.00, P < 0.01), KD群体FPED与FLH2的相关系数最低(R=–0.229, P < 0.01)。针对GK和KD群体, 进一步分析不依赖群体等位基因频率的3种近交系数(Fped、FROH和FHOM)与其他6种近交系数间的相关性, Fped与FVR1之间的相关性系数为0.277, 表现为中度相关; FROH与FVR1、FYA1、FLH1之间的相关系数变化范围在0.376~0.493; FHOM与FVR1、FYA1、FLH1之间的相关系数变化范围在0.775~1.00, 表现为中高度相关。
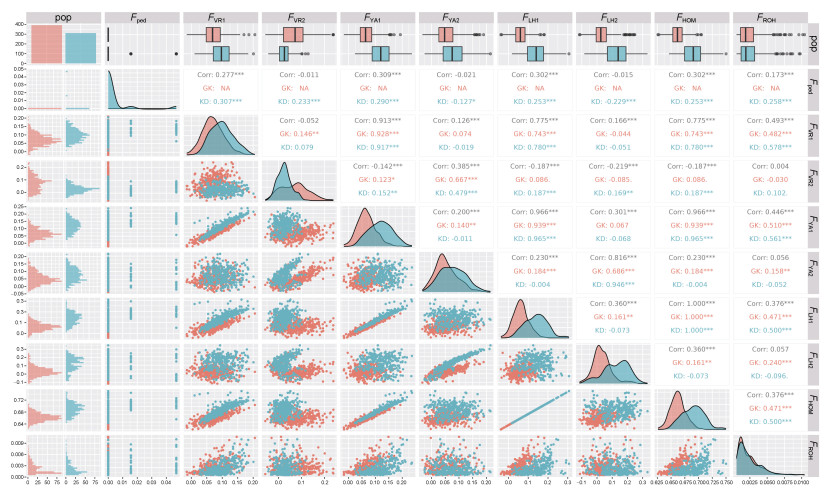 |
| 图 5 凡纳滨对虾两个群体不同近交系数之间的相关性比较 Fig. 5 Comparison of correlation for inbreeding coefficient of two selected populations 注: “GK”代表高抗选育群体, “KD”代表快大选育群体 |
杂合度是衡量群体遗传变异的重要指标。GK群体的Ho、He和PIC均高于KD群体(0.35 vs 0.32; 0.36 vs 0.33; 0.28 vs 0.26), 可能是因为GK群体的家系样本量更大, 其奠基者群体更多所致。在申淑慧等(2020)等的研究中, 使用24个SNPs分析对虾野生群体(2个群体, 每个群体取30尾样本)及选育群体(同为2个群体, 每个群体取30尾样本)的遗传多样性, 结果显示选育群体的Ho和He均值分别为0.169 4、0.168 8, PIC均值为0.134 1。在刘绵宇等(2023)的研究中, 使用38K SNP数据分析三个引进群体的遗传多样性, 结果显示Ho和He均值分别是0.27、0.26, PIC均值为0.22, 也低于本研究的相关结果。Medrano-Mendoza等(2023)针对一个凡纳滨对虾6代选育群体的遗传多样性(176个家系, 1 685尾虾)分析表明, 其Ho和He均为0.38; 厄瓜多尔一个20代选育群体(约为140个家系/世代, 96个基因分型亲本)的遗传多样性分析结果表明, Ho及He均值均为0.37, 上述结果略高于本文研究结果。上述对比结果, 主要受所分析育种群体的奠基者群体数量、家系数量和样本量等因素影响。为了进一步提高育种群体的可持续选育潜力, 仍需进一步引进性能优异的野生或改良种质资源, 扩大家系规模。
水产养殖动物育种群体ROH分布特征相关研究较少, 仅在虹鳟、银鲑、梭鲈、大菱鲆等群体中有少量报道。在本研究中, 凡纳滨对虾GK和KD群体每个ROH的平均长度分别为(1.70±0.34) Mb和(1.65± 0.38) Mb, 每个样本ROH的平均数量分别为1.98± 1.30和2.07±1.37, 远低于上述物种的相关分析结果。譬如, D'Ambrosio等(2019)使用57K SNP固相芯片对虹鳟4个商业选育群体的ROH进行统计分析, 结果显示每尾鱼的平均ROH数量在4个群体间的变化范围(50.2±9)~(68.2±9.4), 每个ROH的平均长度在4个群体间的变化范围4.14~5.38 Mb; Yoshida等(2020)使用200K SNP芯片对银鲑2个选育群体进行ROH检测, 每尾鱼的平均ROH数量在2个群体间的变换范围(35.65±8.64)~(36.93±7.13), 每个ROH的平均长度在2个群体间的变化范围(6.47±7.39)~(7.17±7.67) Mb, 明显高于本文研究结果, 推测其原因可能主要是受育种群体的选育时间长短所致。虹鳟4个选育群体经历了5到10代选择, 银鲑2个选育群体经历了9代选择, 而本文所分析的2个育种群体仅经历了3次选择, 明显少于前者。加之在制定配种方案时, 采取了严格的避免近亲交配策略, 因此导致本文所分析育种群体的ROH数量较少, 片段长度相对较短。此外, 不同的基因组大小以及使用的SNP密度也会对检测出的ROH长度产生影响。银鲑的基因组大小为2.4 Gb, 使用的SNP面板密度为42.6 SNP/Mb (Yoshida et al, 2020), 在本研究中, 凡纳滨对虾基因组大小为1.4 Gb, SNP面板密度为30.9 SNP/Mb, 明显低于银鲑SNP面板密度。
只有准确地估计个体的近交水平, 才能有效地评估其近交衰退程度, 进一步估计育种群体的有效群体大小, 为育种方案优化提供重要参考。在本研究中, GK和KD群体在3次定向配种过程中, 由于严格控制近亲交配, 因此其FPED均很小, 甚至为0 (表 5)。ROH分析也表明, 长片段ROH占比较低, 也表明近期没有发生近交。因此, 8种基因组近交系数主要体现的是GK和KD群体其奠基者群体的近交水平。其中7种基因组近交系数估计值(FVR2除外)显示, KD群体的近交水平高于GK群体, 这表明“快大系”群体的选育历史要明显早于“高抗系”选育群体。“快大系”奠基者群体可追溯到美国1988年建立的SPF群体; 而“高抗系”奠基者多为来自厄瓜多尔的SPT或SPR群体, 是在2000年之后直接从野生群体中筛选出的群体(Wyban, 2019)。与GK群体相比, KD群体中短ROH的比例更高, 也为这一结论提供了数据支持(表 4)。
八种基因组近交系数中, 其中6种方法(FVR1、FVR2、FYA1、FYA2、FLH1、FLH2)均依赖基础群体的等位基因频率pk, 由于在本文中无法获得基础群体样本, 只能用当前群体的等位基因频率代替pk, 因此基于这6种方法的基因组近交系数并不能反映这两个群体的真实基因组近交水平, 更多的是反应两个群体间以及个体间的近交水平差异。其余三种不依赖群体等位基因频率的3种近交系数(FPED、FROH和FHOM), 由于选育群体的奠基者系谱信息缺失, FPED也很难反应出育种群体真实的近交水平; 因此, FHOM和FROH这两种方法更为真实的反应育种群体的近交水平。如果只是要对个体或群体间的近交水平进行比较, 那么与FHOM和FROH存在中高度相关的FVR1、FYA1和FLH1等方法也可以作为替代方法。在大菱鲆和比利时蓝牛育种群体的基因组近交分析中, 也发现了类似的结果(Solé et al, 2017; Aramburu et al, 2020)。FVR2和FYA2等方法, 由于在计算过程中给与了稀有等位基因SNP位点更大的加权, 导致它们跟其余7种近交系数的相关性均不高, Caballero等(2022)的研究也表明它们不适合用于评价基因组近交系数。
4 结论本研究针对三次选择后的凡纳滨对虾“高抗系”(GK)和“快大系”(KD)育种核心群体, 利用液相育种芯片“黄海芯1号”(55 SNP)对家系个体进行高通量基因分型, 主成分及进化树分析表明群体间明确分层; 基于G矩阵的亲缘关系热图分析表明, 与GK群体相比, KD群体内个体间的亲缘关系更近; Fst值显示两个群体间存在中等遗传分化, 可划分为2个群体; ROH和基因组近交分析表明, 两个群体近期内没有发生近亲交配, KD群体的选育历史比GK群体更长; 当无法获得基础群体的等位基因频率时, FROH和FHOM可准确地评价育种群体的真实近交水平, 而FVR1、FYA1和FLH1可以用来比较群体及个体的相对近交水平。上述研究结果为准确地评估育种群体的近交水平和优化育种方案提供了重要参考依据。
申淑慧, 戴习林, 2020. 基于生长和抗逆功能基因SNP分子标记的凡纳滨对虾野生及选育群体遗传多样性分析[J]. 南方农业学报, 51(11): 2836-2845. |
刘绵宇, 李旭鹏, 孔杰, 等, 2023. 液相芯片"黄海芯1号"在凡纳滨对虾急性肝胰腺坏死病抗性基础群体遗传评估中的应用[J]. 水产学报, 47(1): 019417. |
农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会 , 2023. 2023中国渔业统计年鉴[M]. 北京: 中国农业出版社.
|
ARAMBURU O, CEBALLOS F, CASANOVA A, et al, 2020. Genomic signatures after five generations of intensive selective breeding: runs of homozygosity and genetic diversity in representative domestic and wild populations of turbot (Scophthalmus maximus)[J]. Frontiers in Genetics, 11: 296. DOI:10.3389/fgene.2020.00296 |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al, 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 32(3): 314-331. |
CABALLERO A, FERNÁNDEZ A, VILLANUEVA B, et al, 2022. A comparison of marker-based estimators of inbreeding and inbreeding depression[J]. Genetics Selection Evolution, 54(1): 82. DOI:10.1186/s12711-022-00772-0 |
CASADO E, CABRERA H, GONZÁLEZ M, et al, 2022. Genetic diversity and growth-related traits in Penaeus vannamei after ten years without introducing new stocks into Cuba[J]. Aquaculture, 554: 738097. DOI:10.1016/j.aquaculture.2022.738097 |
CURIK I, FERENČAKOVIĆ M, SÖLKNER J, 2014. Inbreeding and runs of homozygosity: a possible solution to an old problem[J]. Livestock Science, 166: 26-34. DOI:10.1016/j.livsci.2014.05.034 |
D'AMBROSIO J, PHOCAS F, HAFFRAY P, et al, 2019. Genome-wide estimates of genetic diversity, inbreeding and effective size of experimental and commercial rainbow trout lines undergoing selective breeding[J]. Genetics Selection Evolution, 51(1): 26. DOI:10.1186/s12711-019-0468-4 |
DE LOS RÍOS-PÉREZ L, CAMPOS-MONTES G R, MARTÍNEZ-ORTEGA A, et al, 2017. Inbreeding effects on reproductive traits in a breeding population of Pacific white shrimp Penaeus (Litopenaeus) vannamei[J]. Aquaculture, 479: 442-446. DOI:10.1016/j.aquaculture.2017.06.013 |
DE LOS RÍOS-PÉREZ L, DRUET T, GOLDAMMER T, et al, 2022. Analysis of autozygosity using whole-genome sequence data of full-sib families in pikeperch (Sander lucioperca)[J]. Frontiers in Genetics, 12: 786934. DOI:10.3389/fgene.2021.786934 |
GENGLER N, MAYERES P, SZYDLOWSKI M, 2007. A simple method to approximate gene content in large pedigree populations: application to the myostatin gene in dual-purpose Belgian Blue cattle[J]. Animal, 1(1): 21-28. DOI:10.1017/S1751731107392628 |
KELLER M C, VISSCHER P M, GODDARD M E, 2011. Quantification of inbreeding due to distant ancestors and its detection using dense single nucleotide polymorphism data[J]. Genetics, 189(1): 237-249. DOI:10.1534/genetics.111.130922 |
LI C C, HORVITZ D G, 1953. Some methods of estimating the inbreeding coefficient[J]. American Journal of Human Genetics, 5(2): 107-117. |
MEDRANO-MENDOZA T, GARCÍA B F, CABALLERO-ZAMORA A, et al, 2023. Genetic diversity, population structure, linkage disequilibrium and GWAS for resistance to WSSV in Pacific white shrimp (Litopenaeus vannamei) using a 50K SNP chip[J]. Aquaculture, 562: 738835. DOI:10.1016/j.aquaculture.2022.738835 |
NEJATI-JAVAREMI A, SMITH C, GIBSON J P, 1997. Effect of total allelic relationship on accuracy of evaluation and response to selection[J]. Journal of Animal Science, 75(7): 1738-1745. DOI:10.2527/1997.7571738x |
SOLÉ M, GORI A S, FAUX P, et al, 2017. Age-based partitioning of individual genomic inbreeding levels in Belgian Blue cattle[J]. Genetics Selection Evolution, 49(1): 92. DOI:10.1186/s12711-017-0370-x |
STRAUS K M, VADOPALAS B, DAVIS J P, et al, 2015. Reduced genetic variation and decreased effective number of breeders in five year-classes of cultured geoducks (Panopea generosa)[J]. Journal of Shellfish Research, 34(1): 163-169. DOI:10.2983/035.034.0120 |
TORO M, BARRAGÁN C, ÓVILO C, et al, 2002. Estimation of coancestry in Iberian pigs using molecular markers[J]. Conservation Genetics, 3(3): 309-320. DOI:10.1023/A:1019921131171 |
VANRADEN P M, 2008. Efficient methods to compute genomic predictions[J]. Journal of Dairy Science, 91(11): 4414-4423. DOI:10.3168/jds.2007-0980 |
WANG J, 2014. Marker-based estimates of relatedness and inbreeding coefficients: an assessment of current methods[J]. Journal of Evolutionary Biology, 27(3): 518-530. DOI:10.1111/jeb.12315 |
WANG H, TENG M X, LIU P P, et al, 2022. Selection signatures of pacific white shrimp Litopenaeus vannamei revealed by whole-genome resequencing analysis[J]. Frontiers in Marine Science, 9: 844597. DOI:10.3389/fmars.2022.844597 |
WEIR B S, COCKERHAM C C, 1984. Estimating F-statistics for the analysis of population structure[J]. Evolution, 38(6): 1358-1370. |
WYBAN J, 2019. Selective breeding of penaeus vannamei: impact on world aquaculture and lessons for future[J]. Journal of Coastal Research, 86(SI): 1-5. |
YANG J, BENYAMIN B, MCEVOY B P, et al, 2010. Common SNPs explain a large proportion of the heritability for human height[J]. Nature Genetics, 42(7): 565-569. DOI:10.1038/ng.608 |
YANG J, LEE S H, GODDARD M E, et al, 2011. GCTA: a tool for genome-wide complex trait analysis[J]. The American Journal of Human Genetics, 88(1): 76-82. DOI:10.1016/j.ajhg.2010.11.011 |
YOSHIDA G M, CÁCERES P, MARÍN-NAHUELPI R, et al, 2020. Estimates of autozygosity through runs of homozygosity in farmed coho salmon[J]. Genes, 11(5): 490. DOI:10.3390/genes11050490 |
 2024, Vol. 55
2024, Vol. 55


