文章信息
- 潘滢, 郑利兵, 王军. 2018.
- PAN Ying, ZHENG Li-bing, WANG Jun. 2018.
- 条纹斑竹鲨5个地理群体的简单重复序列中间区域分析
- Genetic structure analysis of five populations of Chiloscyllium plagiosum using inter-simple sequence repeat molecular markers
- 海洋科学, 42(3): 31-36
- Marine Sciences, 42(3): 31-36.
- http://dx.doi.org/10.11759//hykx20170925003
-
文章历史
- 收稿日期:2017-09-25
- 修回日期:2017-11-29
条纹斑竹鲨(Chiloscyllium plagiosum)属于软骨鱼纲(Chondrichthyes)、须鲨目(Orectolobiformes)、须鲨科(Orectolobidae)、斑竹鲨属(Chiloscyllium), 分布于印度西-太平洋区的沿岸海域。在中国主要栖息于东海、台湾海峡及东海沿岸海域, 游泳能力较弱, 以底栖动物为食, 是一种体内受精、卵生型的暖水性底层小型鲨鱼[1]。由于条纹斑竹鲨经济价值高, 生育力低, 近年来过度捕捞导致其资源量不断衰减, 对自然群体的种质资源和遗传多样性产生巨大的影响。目前对于条纹斑竹鲨群体遗传多样性的研究主要包括形态学水平[2]、分子水平等方面[3-5]。但上述研究中所用群体和样本的数量偏少, 无法全面、客观地反映中国境内条纹斑竹鲨种群的遗传多样性现状。
简单重复序列中间区域(inter-simple sequence repeats, ISSR)是由Zietkiewicz等[6]创建的一种新型的微卫星类分子标记技术。相比RFLP、RAPD技术, 具有重复性与稳定性好、多态性检测率高等优点[7], 广泛应用于种质资源鉴定[8]、多样性分析[9]、指纹图谱的构建等[10]研究, 但在鱼类中ISSR的研究报道较少。硬骨鱼类研究中, 相关报道见于
条纹斑竹鲨样品于2007年10月~2009年1月分别采自福建厦门(XM)、福建平潭(PT)、海南海口(HK)、广东湛江(ZJ)和台湾(TW)沿岸海域(图 1), 其中XM群体28尾, PT群体33尾, HK群体34尾, ZJ群体36尾, TW群体32尾。个体全长20~80 cm, 体质量100~800 g。取新鲜肌肉置于无水乙醇中固定, 于–20℃中保存。DNA提取参考Sambrook[15]等的酚/氯仿抽提方法。
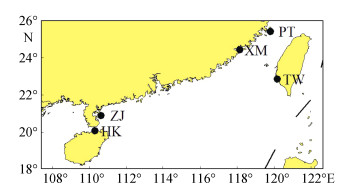 |
| 图 1 采样海域位置图 Fig. 1 Map of sampling area location |
ISSR分析中使用的引物核心序列参考加拿大哥伦比亚大学公布的序列(http://www.biotech.ubc.ca/services/naps/primers/Primers.pdf)以及GenBank中条纹斑竹鲨的12个微卫星序列(EU661893~EU661904), 利用Primer Premier 5.0共设计出56对ISSR引物, 由上海生工生物工程股份有限公司合成。从每个群体随机抽取5个DNA样品, 以此为模板优化PCR扩增条件并筛选出13对条带清晰、多样性高的ISSR引物。25 μL PCR反应体系包括:模板DNA 1 μL(约30 ng), 2.5 μL 10×buffer, 0.5 μL dNTP(2.5 mmol/L), 上、下游引物各1 μL(10 mmol/L), Taq polymerase (TaKaRa) 0.5 μL(2.5 U/μL), 无菌超纯水补齐体积。PCR反应条件为: 95℃ 5 min; 94℃ 30 s, 50~56℃ 1 min, 72℃ 2 min, 35个循环; 72℃ 10 min。PCR产物经1.2%琼脂糖凝胶电泳后, EB染色显带由GAS7001B凝胶成像系统观察拍照。
1.3 数据分析将ISSR电泳图谱拍照记录后进行人工读带, 同一引物扩增的电泳迁移率一致的条带为同一位点的产物, 按扩增阳性记为1和阴性记为0的格式构成0/1数据矩阵[16], 采用Popgene32软件对条纹斑竹鲨5个地理群体进行遗传参数分析分别计算其多态性位点百分率(PPB)、观测等位基因数(Na)、有效等位基因数(Ne)、Shannon’s信息指数(I)、Nei’s基因多样性(H)、总基因多样性(Ht)、群体内基因多样性(Hs)、各群体间的遗传分化指数(Gst)、基因流(Nm)、Nei的遗传距离(D)、遗传相似系数(S)。用Arlequin 3.5软件计算群体间遗传分化系数(Fst)及分子方差分析(AMOVA)。应用Mega5.0软件进行非加权的组平均法(UPGMA)聚类, 构建群体间遗传关系。
2 结果 2.1 条纹斑竹鲨鱼群体多样性分析从56对引物中共筛选出了13对重复性好、条带清晰、多态性高的引物对条纹斑竹鲨5个群体的遗传多样性进行分析, 图 2为代表性引物issr24和issr25的扩增电泳图谱。13对ISSR引物扩增出的条带数目4~9条不等, 条带大小在100~2 000 bp; 共扩增产生81个可统计的条带, 其中多态性位点57个, 总PPB为70.37%(表 1)。
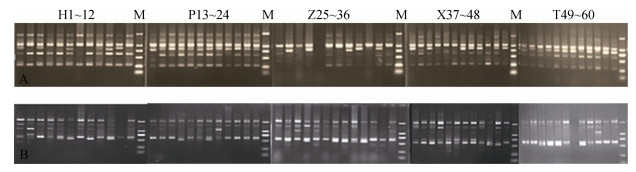 |
| 图 2 引物issr24和issr25的扩增结果 Fig. 2 Amplification results of primers issr24 and issr25 A、B.分别为引物issr24和issr25扩增结果, 其中M为DL2000 DNA Marker, H1~12、P13~24、Z25~36、X37~48和T49~60分别为HK、PT、ZJ、XM和TW群体12个个体的电泳条带 A and B. the amplification results of primers issr24 and issr25, respectively; M. DL2000 DNA marker; H1-12, P13-24, Z25-36, X37-48, and T49-60. the electrophoretic bands of 12 samples from HK, PT, ZJ, XM, and TW populations, respectively |
| 引物名称 | 引物序列 | 退火温度/℃ | 统计条带数 | 多态性条带 | PPB/% |
| issr9 | (CT)7TAG | 52 | 7 | 5 | 71.4 |
| issr12 | (CT)8GA | 54 | 4 | 3 | 75.0 |
| issr13 | (CT)8A | 54 | 6 | 2 | 33.3 |
| issr21 | AGGTC(CAG)4 | 56 | 6 | 6 | 100 |
| issr24 | (GA)8YT | 52 | 9 | 9 | 100 |
| issr25 | (AG)8YA | 52 | 8 | 7 | 87.5 |
| issr27 | (AG)8C | 54 | 6 | 5 | 83.3 |
| issr29 | (AC)7CG | 50 | 5 | 2 | 40.0 |
| issr31 | (GT)7CA | 52 | 6 | 5 | 83.3 |
| issr35 | (AC)7GGG | 54 | 5 | 2 | 40.0 |
| issr39 | (GT)7TG | 52 | 6 | 3 | 50.0 |
| issr46 | (GT)7CG | 50 | 7 | 5 | 71.4 |
| issr51 | (AC)7AT | 52 | 6 | 3 | 50.0 |
| 合计 | 81 | 57 | 70.37 |
不同群体的遗传多样性参数结果表明: HK和ZJ群体的PPB、Na最高, 分别达到58.02%和1.5802; PT和TW群体最低, 分别为38.27%和1.3827; HK群体的Ne、H和I参数值最高, 分别达到1.3717、0.2155和0.3193; TW群体最低, 各数值分别为1.2269、0.1353和0.203 2, 且HK和ZJ群体遗传多样性各参数都明显高于PT、XM和TW群体(表 2)。
| 群体 | 个体数 | PPB/% | Na | Ne | H | I |
| HK | 34 | 58.02 | 1.5802 | 1.3717 | 0.2155 | 0.3193 |
| PT | 33 | 38.27 | 1.3827 | 1.2413 | 0.1411 | 0.2096 |
| ZJ | 36 | 58.02 | 1.5802 | 1.3402 | 0.2030 | 0.3049 |
| XM | 28 | 44.44 | 1.4444 | 1.2969 | 0.1716 | 0.2529 |
| TW | 32 | 38.27 | 1.3827 | 1.2269 | 0.1353 | 0.2032 |
依据Nei’s基因多样性指数得出Ht、Hs和Gst分别为0.2026、0.1733和0.1447, 根据Gst计算得出群体间的Nm为2.9550, 表明不同的条纹斑竹鲨群体存在一定的基因交流。分子方差分析(AMOVA)结果显示:群体间遗传分化系数Fst值为0.1338, 表明总的遗传变异中有13.38%的变异存在于群体间, 86.62%的遗传变异存在于群体内(表 3)。
| 变异来源 | 自由度 | 平方和 | 方差组分 | 方差比例 |
| 群体间 | 4 | 112.629 | 0.72534 | 13.38 |
| 群体内 | 157 | 737.192 | 4.69549 | 86.62 |
| 合计 | 161 | 849.821 | 5.42083 |
两两群体间遗传距离在0.0097~0.0767, 其中ZJ群体和TW群体的遗传距离最大, 达0.0767, PT群体和XM群体的遗传距离最小, 为0.0097(表 4)。应用UPGMA进行聚类分析表明: PT群体和XM群体聚为一支, 再与TW群体聚类, 最后与ZJ群体和HK群体形成的分支聚类(图 3)。
| 群体 | HK | PT | ZJ | XM | TW |
| HK | — | 0.9448 | 0.9886 | 0.9517 | 0.9334 |
| PT | 0.0567 | — | 0.9390 | 0.9903 | 0.9791 |
| ZJ | 0.0114 | 0.0629 | — | 0.9532 | 0.9262 |
| XM | 0.0495 | 0.0097 | 0.0479 | — | 0.9852 |
| TW | 0.0690 | 0.0211 | 0.0767 | 0.0149 | — |
 |
| 图 3 条纹斑竹鲨5个群体的UPGMA聚类图 Fig. 3 UPGMA dendrogram among five populations of Chiloscyllium plagiosum |
PPB和H是评估鱼类种群遗传变异及其种质资源状况的2个主要参数。通过比较条纹斑竹鲨(P= 38.27%~58.02%, h=0.1353~0.2155)与鳡(Elopichthys bambusa)(P=18.18%~27.27%, h=0.0245~0.0763)[17]、松江鲈鱼(Trachidermus fasciatus)(P=49.17%~74.03%, h=0.1530~0.2777)[18]、瓯江彩鲤(Cyprinus carpio var. color)(P=51.5%~67.8%, h=0.2176~0.2745)[19]、刀鲚(Coilia nasus)(P =70.04%~76.76%, h=0.1885~0.2285)[13]的多态位点比例和基因多样性, 结果显示条纹斑竹鲨的遗传多样性较为丰富, 表明被检测的条纹斑竹鲨的种质资源良好。目前对于中国境内条纹斑竹鲨的遗传多样性的研究已有相关报道, 陈骁等[3]利用RAPD技术分析表明中国南部沿海闽东、闽南、粤西3个条纹斑竹鲨地理群体遗传多样性水平较低, 相比RAPD技术, 本研究使用的ISSR技术具有更高的多态性检测率[7], 同时采用更多的群体数和个体数, 因此所获得的研究结果更加客观和全面。
条纹斑竹鲨栖息于近海沿岸海域, 容易受到人类捕捞行为影响导致其种质的下降, 对几种沿岸型鲨鱼的种质资源的研究也支持这个观点。Weydder等[20]利用线粒体控制区和细胞色素b基因研究大西洋沿岸的黑边鳍真鲨(Carcharhinus limbatus)、小尾真鲨(Carcharhinus porosus)、加勒比斜锯牙鲨(Rhizo prionodon porosus)和小眼双髻鲨(Sphyrna tudes)的遗传结构, 发现由于资源的过度捕捞导致4种鲨鱼的遗传多样性持续降低; Silvana等[21]对沿岸型种类舒氏星鲨(Mustelus schmitti)的遗传多样性的研究也得到相似的结论。因此条纹斑竹鲨种群需要科学合理的利用和保护。
条纹斑竹鲨主要栖息于近岸浅海底层, 为适应底栖生活而牺牲自身游泳活动的灵活性[22], 因此很难进行长距离的基因交流, 形成了与地理位置远近相关的基因交流距离隔离模式。陈骁等对我国南部沿海5个海域的条纹斑竹鲨5个群体的线粒体控制区的研究[5]以及傅蒙娜等[4]对条纹斑竹鲨的3个地理群体的线粒体细胞色素b的研究都支持了这个结论。另外, 对于游泳活动能力不强的其他种类如口虾蛄(Oratosqilla oratoria)也得到相似的结论, 即群体的系统发育关系一定程度上是由地理位置分布情况决定[23], 但对于幼体及成体具有较强迁移能力的种类如大斑石鲈(Pomadasys maculatus), 群体无明显的地理结构[24]。本研究中条纹斑竹鲨5个群体的聚类结果分析表明, PT、XM和TW群体形成以台湾海峡海域为主的地理群体, HK和ZJ群体形成以南海北部海域为主的地理群体, 另外, XM与HK群体, XM与ZJ群体的S都高于PT与HK群体, PT与ZJ群体, 而XM群体在地理位置上比PT群体更靠近HK和ZJ群体。总之, 条纹斑竹鲨群体间的基因交流与地理远近息息相关, 进一步论证了条纹斑竹鲨基因交流的距离隔离模式。
在海峡对鲨鱼种群间基因交流的影响问题上, Chrysoula等[25]对深海鲨鱼黑腹乌鲨(Etmopterus spinax)研究表明, 直布罗陀海峡阻碍大西洋和地中海海域群体的基因交流, 但是Duncan等[26]却认为海峡不仅不会阻碍鲨鱼的基因交流, 反而会成为基因交流的通道; 海峡海域中鲨鱼种群间的基因交流由海峡的水文特征, 鲨鱼种群的摄食和生殖行为等因素共同作用。本研究中PT、XM和TW群体间的S十分接近, 然而目前条纹斑竹鲨在台湾海峡海域的摄食和生殖活动未见报道, 因此暂时未能阐明台湾海峡对条纹斑竹鲨基因交流的影响机理; 但是从PT、XM和TW群体的UPGMA聚类结果显示TW群体单独聚为一支, 表明台湾海峡可能对条纹斑竹鲨的基因交流有一定的阻碍作用。
有些学者认为, 在更新世冰期, 亚洲的太平洋海域种群可能被3个陆缘海所隔离:中国东部海域、中国南部海域以及日本海; 台湾海峡暴露在外的大陆架阻碍了中国东部和南部海域种群的基因交流, 而后随着海平面的上升, 中国东部和南部海域种群逐渐开始进行频繁的基因交流[27-29]。由于本研究中缺少中国东部海域条纹斑竹鲨样品, 无法对不同海域条纹斑竹鲨种群系统地理生物学进行深层次的探讨。因此对于条纹斑竹鲨在我国沿海种质资源的调查需更系统和深入的研究。
| [1] |
朱元鼎, 孟庆闻. 福建鱼类志(上卷)[M]. 福州: 福建科学技术出版社, 1984: 13-103. Zhu Yuanding, Meng Qingwen. Fujian fish Chronicles (Volume 1)[M]. Fuzhou: Fujian Science and Technology Press, 1984: 13-103. |
| [2] |
李萌, 曾凡荣, 王军. 中国南部海域不同地理群体条纹斑竹鲨的形态差异分析[J]. 厦门大学学报(自然科学版), 2013, 52(1): 139-144. Li Meng, Zeng Fanrong, Wang Jun. Morphological variations of Chiloscyllium plagiosum in different geographical populations along southern China coast[J]. Journal of Xiamen University(Natural Science), 2013, 52(1): 139-144. |
| [3] |
陈骁, 杨圣云, 潘聪. 中国南部沿海条纹斑竹鲨遗传多样性研究[J]. 厦门大学学报(自然科学版), 2008, 47(4): 580-584. Chen Xiao, Yang Shengyun, Pan Cong. Genetic diversity of Chiloscyllium plagiosum in China's southern coast[J]. Journal of Xiamen University(Natural Science), 2008, 47(4): 580-584. |
| [4] |
傅蒙娜, 王军, 丁少雄, 等. 基于线粒体DNA细胞色素b序列分析3个条纹斑竹鲨群体的遗传结构和遗传分化[J]. 热带海洋学报, 2010, 29(6): 86-91. Fu Mengna, Wang Jun, Ding Shaoxiong, et al. Studies on the genetic structure and genetic subdivision of white spotted bamboo shark, Chiloscyllium plagiosum, by analysing mitochondrial Cyt b genes[J]. Journal of Tropical Oceanography, 2010, 29(6): 86-91. DOI:10.11978/j.issn.1009-5470.2010.06.086 |
| [5] |
陈骁, 杨圣云, 潘聪. 我国南部海域条纹斑竹鲨线粒体DNA控制区遗传多态性研究[J]. 海洋学报, 2008, 30(6): 115-121. Chen Xiao, Yang Shengyun, Pan Cong. Genetic polymorphism of mitochondrial control region in Chiloscyllium plagiosum from coastal waters of southern China[J]. Acta Oceanologica Sinica, 2008, 30(6): 115-121. |
| [6] |
Zietkiewicz E, Rafalske A, Labuda D. Genome fingerprinting simple sequence repeat (SSR) anchored polymerase chain reaction amplification[J]. Genome, 1994, 20: 178-183. |
| [7] |
Nagaoka T, Ogihara Y. Applicability of inter-simple sequence repeat polymorphisms in wheat for use as DNA markers in comparison to RFLP and RAPD markers[J]. Theoretical and Applied Genetics, 1997, 94: 597-602. DOI:10.1007/s001220050456 |
| [8] |
Marco C, Tiziana L, Marco C G, et al. Identification of mediterranean Diplodus spp. and Dentex dentex (Sparidae) by meansof DNA inter-simple sequence repeat (ISSR) markers[J]. Journal of Experimental Marine Biology and Ecology, 2009, 368: 147-152. DOI:10.1016/j.jembe.2008.09.021 |
| [9] |
Zhao H, Wang Y X, Yang D Z, et al. An analysis of genetic diversity in Marphysa sanguinea from different geographic populations using ISSR polymorphisms[J]. Biochemical Systematics and Ecology, 2016, 64: 65-69. DOI:10.1016/j.bse.2015.11.002 |
| [10] |
Zhao W G, Miao X X, Zang Bo, et al. Construction of Fingerprinting and Genetic Diversity of Mulberry cultivars in China by ISSR Markers[J]. Acta Genetica Sinica, 2006, 33(9): 851-860. DOI:10.1016/S0379-4172(06)60119-4 |
| [11] |
刘红艳, 熊飞, 董元火, 等.  浪白鱼野生与养殖群体遗传多样性的ISSR分析[J]. 华中师范大学学报, 2012, 46(5): 591-595. 浪白鱼野生与养殖群体遗传多样性的ISSR分析[J]. 华中师范大学学报, 2012, 46(5): 591-595.Liu Hongyan, Xiong Fei, Dong Yuanhuo, et al. ISSR analysis on genetic diversity between the wild and cultured populations of Anabarulius grahami[J]. Journal of Central China Normal University, 2012, 46(5): 591-595. |
| [12] |
韩晓磊, 郁建峰, 陆秀乡, 等. 雅罗鱼亚科鱼类(鳡鱼、草鱼及赤眼鳟的ISSR分子鉴定[J]. 水产科学, 2010, 29(9): 546-549. Han Xiaolei, Yu Jianfeng, Lu Xiuxiang, et al. Molecular identification of Leuciscinae (yellowcheek carp Elopichthys bambusa, grass carp Ctenopharyngodon idellus and Squaliobarbus curriculus) by ISSR[J]. Fisheries Science, 2010, 29(9): 546-549. |
| [13] |
Yang Q L, Gao T X, Miao Z Q. Differentiation between populations of Japanese grenadier anchovy (Coilia nasus) in Northwestern Pacific based on ISSR markers:Implications for biogeography[J]. Biochemical Systematics and Ecology, 2011, 39: 286-296. DOI:10.1016/j.bse.2011.07.019 |
| [14] |
Liu Y G, Yu Z G, Bao B L, et al. Population genetics studies of half-smooth tongue sole Cynoglossus semilaevis using ISSR markers[J]. Biochemical Systematics and Ecology, 2009, 36: 821-827. |
| [15] |
Sambrook J, Fritsch E F, Maniatis T. Molecular cloning:alaboratory manual[M]. New York: Cold Spring Harbor Laboratory Press, 1989: 672-683, 955.
|
| [16] |
王世锋, 杜佳莹, 苏永全, 等. 斜带髭鲷野生与养殖群体遗传结构的ISSR分析[J]. 海洋学报, 2007, 29(4): 105-110. Wang Shifeng, Du Jiaying, Su Yongquan, et al. The genetic structure of Nature and reared groups of Hapalogenys nitens by ISSR analysis[J]. Acta Oceanologica Sinica, 2007, 29(4): 105-110. |
| [17] |
韩晓磊, 徐建荣, 郁建峰, 等. 鳡遗传多样性的ISSR分析[J]. 水产科学, 2009, 28(9): 509-512. Han Xiaolei, Xu Jianrong, Yu Jianfeng, et al. Analysis of genetic diversity of yellowcheek carp Elopichthys bambusa by ISSR Markers[J]. Fisheries Science, 2009, 28(9): 509-512. |
| [18] |
马召腾, 刘至治, 潘连德, 等. 中国境内松江鲈鱼群体遗传变异的ISSR分析[J]. 水产学报, 2012, 36(7): 1042-1048. Ma Zhaoteng, Liu Zhizhi, Pan Liande, et al. Genetic variation analysis of roughskin sculpin (Trachidermus fasciatus) in native populations of China by ISSR[J]. Journal of Fisheries of China, 2012, 36(7): 1042-1048. |
| [19] |
Lv Y P, Hu Z H, Yang X Q, et al. Analysis of genetic variation in selected generations of "Whole Red" pattern Cyprinus carpio var. color using ISSR markers[J]. Biochemical Systematics and Ecology, 2012, 44: 243-249. DOI:10.1016/j.bse.2012.05.002 |
| [20] |
Weydder T, Luis F R, Davidson S, et al. Multiple substitutions and reduced genetic variability in sharks[J]. Biochemical Systematics and Ecology, 2013, 49: 21-29. DOI:10.1016/j.bse.2013.02.004 |
| [21] |
Silvana P, Graciela G, Philip M, et al. Low genetic diversity and population structure of the narrownose shark (Mustelus schmitti)[J]. Fisheries Research, 2010, 106: 468-473. DOI:10.1016/j.fishres.2010.09.022 |
| [22] |
Wilga C D, Lauder G V. Functional morphology of the pectoral fins in bamboo sharks, Chiloscyllium plagiosum: benthic vs. pelagic station-holding[J]. Journal of Morphology, 2001, 249: 195-209. DOI:10.1002/(ISSN)1097-4687 |
| [23] |
董鑫, 邢坤, 隋宥珍, 等. 基于线粒体COI基因序列的4个海域口虾蛄群体的遗传多样性研究[J]. 海洋科学, 2015, 39(7): 29-36. Dong Xin, Xing Kun, Sui Youzhen, et al. Genetic diversity of Oratosqilla oratoria from four sea waters based on the mitochondrial COI gene sequences analysis[J]. Marine Sciences, 2015, 39(7): 29-36. DOI:10.11759/hykx20140412002 |
| [24] |
郜星晨, 章群, 薛丹, 等. 基于线粒体控制区部分序列的南海大斑石鲈遗传多样性分析[J]. 海洋科学, 2016, 40(7): 41-45. Gao Xingchen, Zhang Qun, Xue Dan, et al. Genetic diversity of Pomadasys maculatus in coastal waters of South China Sea via mt DNA control region partial sequences[J]. Marine Sciences, 2016, 40(7): 41-45. DOI:10.11759//hykx20141118001 |
| [25] |
Chrysoula G, Kirsty M, William P, et al. Connectivity in the deep:Phylogeography of the velvet belly lanternshark[J]. Deep-Sea Research I, 2016, 115: 233-239. DOI:10.1016/j.dsr.2016.07.002 |
| [26] |
Duncan K M, Martin A P, Bowen B W, et al. Globalphylogeography of the scalloped hammerhead shark (Sphyrna lewini)[J]. Molecular Ecology, 2006, 15: 2239-2251. DOI:10.1111/j.1365-294X.2006.02933.x |
| [27] |
Liu J X, Gao T X, Wu S F, et al. Pleistocene isolation in the Northwestern Pacific marginal seas and limited dispersal in a marine fish, Chelon haematocheilus (Temminck & Schlegel, 1845)[J]. Molecular Ecology, 2007, 16: 275-288. |
| [28] |
Ni G, Li Q, Kong L F, et al. Phylogeography of bivalve Cyclina sinensis:testing the historical glaciations and Changjian River outflow hypotheses in northwestern Pacific[J]. PLoS One, 2012, 7(11): 1-12. |
| [29] |
Qiu F, Li H, Lin H D, et al. Phylogeography of the inshore fish, Bostrychus sinensis, along the Pacific coastline of China[J]. Molecular Phylogenetics and Evolution, 2016, 96: 112-117. DOI:10.1016/j.ympev.2015.11.020 |
 2018, Vol. 42
2018, Vol. 42


