文章信息
- 刘嘉佩, 陈蔷薇, 徐倩, 刘雪珠, 樊英萍, 戴秋萍, 王健鑫. 2018.
- LIU Jia-pei, CHEN Qiang-wei, XU Qian, LIU Xue-zhu, FAN Ying-ping, DAI Qiu-ping, WANG Jian-xin. 2018.
- 北极黄河站邻近海域浮游细菌多样性初步研究
- Preliminary study on the diversity of planktonic bacteria in the adjacent sea area of the Arctic Yellow River Station
- 海洋科学, 42(4): 53-63
- Marine Sciences, 42(4): 53-63.
- http://dx.doi.org/10.11759/hykx20171105001
-
文章历史
- 收稿日期:2017-12-26
- 修回日期:2018-02-12
2. 浙江海洋大学 东海科学技术学院, 浙江 舟山 316000
2. College of Donghai Science and Technology, Zhejiang Ocean University, Zhoushan 316000, China
北极地区是指66°34~90°N的区域(即为北极圈), 总面积为2.1×107km2, 其中海洋面积为1.3×107km2, 因常年低温、结冰期长、降水少以及强辐射等自然环境, 而构成了特殊的极端环境[1-2]。北极地区生活着许多独特的生物类群, 也不乏具有特殊代谢能力的微型生物, 它们不仅对人类理解生命起源和进化有积极的帮助, 同时能产生大量其他环境微生物所不具备的天然活性物质[3-6]。另外, 微型浮游生物的群落结构和生物量对环境变化的反应极为敏感[7]。目前由于全球变暖引起了一系列北极环境变化[8-9], 这些变化将对北极生态系统, 特别是作为分解者的浮游细菌产生深远影响, 因此, 研究北极生态系统中的浮游细菌群落结构, 对了解该地区的环境变化有重要的意义[10]。
寡营养细菌在通过传统微生物培养方法进行分离时, 由于受到少数优势生长微生物的竞争而不能正常生长, 导致分离效率不高, 很可能会丢失许多宝贵的生物资源[11-12]。自从Button等[13]提出稀释培养技术后, Schut等[14]采用稀释培养法在海洋中分离到37株兼性寡营养细菌和15株专性寡营养细菌; 戴欣等[15]在对太湖水体的研究中发现, 稀释培养基分离的细菌数量远大于牛肉汁蛋白胨培养基。上述研究表明, 稀释培养法比传统培养方法对寡营养细菌分离效果更好。20世纪80年代以来, 人们开始应用分子生物学技术研究海洋微生物[16], 尤其是高通量测序技术, 由于具有数据量大、成本低、耗时短的特点, 近年来被广泛应用于海洋微生物功能及物种多样性研究, 丰富了微生物海洋学领域的研究成果[17-18]。
本研究采用荧光显微计数对北极黄河站附近海水的浮游细菌生物量作初步统计, 结合稀释培养方法和现代分子生物学方法, 对分离纯化的浮游细菌进行系统发育分析, 并通过高通量测序技术对北极黄河站海水不可培养浮游细菌的群落结构进行了初步探讨, 以期了解北极黄河站邻近海域海水浮游细菌的多样性, 为更好地认识和开发北极地区微生物资源提供参考。
1 材料和方法 1.1 样品采集与保存实验所用的海水样品是实验室成员于2014年7月24日~30日参加北极科学考察项目时所得。采样站点分布于11°05′~15°36′E; 78°12′~79°33′N, 命名为BJ1-BJ9(图 1)。样品在科考船上暂存于–20℃冰箱, 带回实验室后马上进行可培养实验, 剩余样品保存于–80℃冰箱中, 不可培养实验于2016年4月进行。
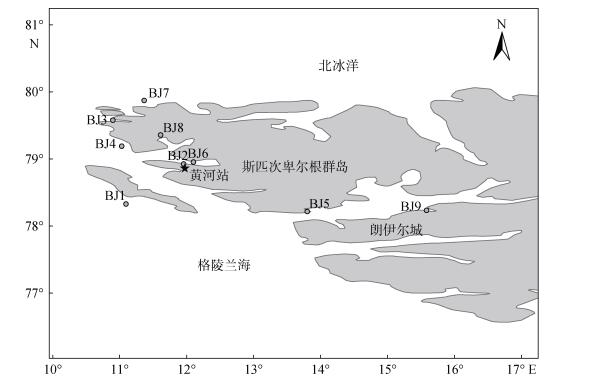 |
| 图 1 样品采集站点分布图 Fig. 1 Distribution of the sampling sites |
LB培养基[19](1%胰蛋白胨, 0.5%酵母提取物, 0.5% NaCl, pH 7.2~7.5, 固体加1.5%琼脂)。
休和利夫森二氏培养基[19](蛋白胨0.2%, NaCl 0.5%, K2HPO4 0.02%, 葡萄糖1%, 琼脂0.6%, 1%溴百里酚蓝水溶液3 mL, 蒸馏水1 000 mL, pH 7.0~7.2)。
1.3 吖啶橙染色计数(AODC)取1 mL的水样与0.05 mL的40%的甲醛溶液混合, 固定10 min, 再加入0.2 mL的0.1%吖啶橙染液进行染色, 染色后置于荧光显微镜(100×油镜)下观察并计数。对有细菌形态、呈现亮绿色或红色荧光的细菌, 随机取20个视野进行拍照并计数, 然后换算成单位样品中细菌数。
根据公式BN=Na×S/(Sf ×(1–0.05)V)计算细菌生物量[20]。其中BN为细菌的数量, Na为视野中亮点的平均数, S为滤膜的面积, Sf为显微镜的视野面积, V为过滤样品的体积(式中0.05为加入37%~40%甲醛固定样品总体积的比例), 单位为L。
1.4 可培养浮游细菌的分离与鉴定 1.4.1 浮游细菌稀释培养样品分批处理, 采用稀释培养法[13], 其中一批样品直接涂布在不同浓度的LB固体培养基(正常浓度、培养基稀释10倍、50倍及100倍)上进行培养, 另一批样品稀释至1×10–3和1×10–4浓度梯度后分别涂布于上述培养基, 4℃培养。每组样品3个平行重复, 并以无菌培养基作为空白对照。
1.4.2 菌株纯化根据观测菌落在培养基上的颜色及菌落形态, 初步排重后进行划线纯化。从纯的筛选平板上挑选出颜色、形态各异的单菌落, 用接种环取1环菌落在稀释50倍的LB固体培养基上再一次划线培养。
1.4.3 形态学观察和革兰氏染色将待鉴定菌株接种于平板上, 4℃培养后进行革兰氏染色, 用油镜观察菌株的形态和革兰氏染色结果, 并对杆菌进行芽孢染色并观察结果。
1.4.4 生理生化鉴定按照东秀珠等[19]描述的方法, 对分离纯化的菌株进行生理生化鉴定。
1.4.5 16S rRNA测序和系统发育分析菌株在150 r/min的转速下摇床培养12~24 h, 用紫外分光光度计测菌液浓度, 用TIANamp Bacteria DNA Kit试剂盒提取DNA。提取产物做PCR扩增反应, 引物为细菌通用引物27f、1492r(27f:5′-AGAG TTTGATCCTGGCTCAG-3′; 1 492r:5′GGTTACCTT GTTACGACTT-3′)[21]。结合形态学观察结果和生理生化鉴定结果, 选取21株菌株送上海凌恩生物科技有限公司测序, 菌株16S rDNA序列结果同NCBI数据库比对并选取同源核酸序列, 用MEGA5.1软件以Neighbor-Joining算法构建系统发育树, 1 000次重复计算Bootstrap值, 进行系统发育分析。
1.5 不可培养浮游细菌群落结构分析 1.5.1 高通量测序为丰富黄河站附近的细菌群落研究数据, 选取黄河站附近的站点BJ2进行不可培养浮游细菌群落结构分析。将BJ2站点的水样通过0.22μm孔径滤膜过滤, 使用PowerWater®DNA Isolation Kit试剂盒提取浮游细菌总DNA。DNA样品送至北京诺禾致源科技股份有限公司, 使用通用引物515F(5′-GTGCCA GCMGCCGCGGTAA-3′)和907R(5′-CCGTCAATTCC TTTGAGTTT-3′)[22]对浮游细菌16S rRNA序列V4-V5区域进行扩增, 将PCR产物条带上机测序。
1.5.2 浮游细菌基因序列分析对测序公司返回的原始测序结果进行优化, 丢弃长度短于150bp、含有模糊碱基、引物含2个以上错配碱基的低质量序列。利用QIIME软件以97%划定阈值, 对16S序列划分操作分类单元(Operational Taxonomic Unit, OTU), 并对OTU进行生物信息统计分析, 分别用Chao1指数和Shannon指数表示浮游细菌群落的丰富度及多样性, 来分析BJ2站点浮游细菌的α-多样性特征[23-24]; 使用EXCEL 2013、Origin9.0等软件将OTU信息进行整理, 分析该站点浮游细菌的群落结构。
2 结果与分析 2.1 浮游细菌计数结果浮游细菌计数结果(表 1)表明BJ5和BJ9站点的细菌数量最多, 分别达5.29×108和4.30×108(个/L)。浮游细菌数量最少的为BJ6站点, 其数量1.22× 108(个/L)约为BJ5的1/5。图 1中可看出, BJ5和BJ9站点临近朗伊尔城, 人类活动相对频繁, 陆源输入影响较大。
| 站点 | BJ1 | BJ2 | BJ3 | BJ4 | BJ5 | BJ6 | BJ7 | BJ8 | BJ9 | 平均 |
| 菌株数/(108个/L) | 1.88 | 1.87 | 1.79 | 2.99 | 5.29 | 1.22 | 3.26 | 3.37 | 4.30 | 2.88 |
菌株的分离实验中, 1×10–3浓度梯度的浮游细菌在稀释50倍的LB培养基上生长情况较为理想, 挑取合适菌株进行纯化。本次实验总共分离到21株代表细菌, 命名为AB(Arctic Bacteria)01~21, 从分离平板上挑取的菌落大多是白色, 部分菌落为黄色和粉色, 菌落较小, 透明度较好, 菌落边缘多为光滑。
对21株可培养菌株进行革兰氏染色与显微观察(部分显微结果见图 2)。结果表明, AB08、AB09和AB17为革兰氏阳性菌, AB08和AB09有明显的杆、球周期变化(图 2-A中细菌形状为杆状, 2-B中细菌形状变成椭球形), AB17可观察到菌丝; 其余皆为革兰氏阴性细菌, 呈直杆状或短杆状; 细菌皆无芽孢。菌株的形态特征(颜色和形状)以及革兰氏染色结果所对应的菌株数量见图 3。
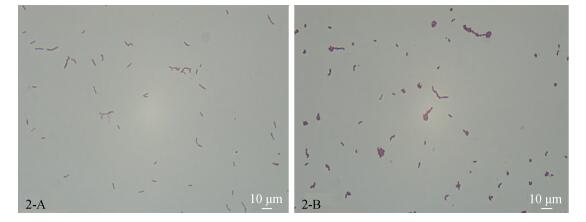 |
| 图 2 菌株AB08革兰氏染色结果 Fig. 2 Gram staining results of strain AB08 2-A为培养一周后染色结果, 2-B为培养两周后染色结果 2-A shows the staining result after 1 week of culture, and 2-B shows the staining result after 2 weeks of culture |
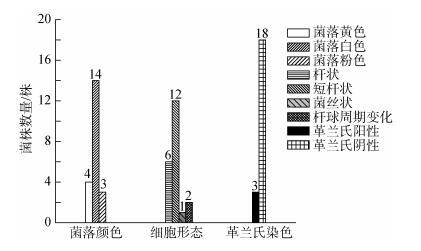 |
| 图 3 北极海水浮游细菌形态特征统计 Fig. 3 Morphological summary of the planktonic bacteria isolated from the seawater of the Arctic Ocean |
对北极分离到的可培养浮游细菌进行的生理生化鉴定, 结果表明, 3项常规生化鉴定试验显示有15株细菌能利用柠檬酸盐, 菌株AB01、AB09、AB12、AB13和AB19的氧化酶反应呈阳性; 菌株AB05、AB10、A12和AB21的硝酸盐还原试验呈阳性; 4项常见碳源利用试验显示, 分别有17株、15株细菌能利用葡萄糖和木糖, 其中AB01、AB02等9株细菌能利用甘露醇, 所有的细菌都不能利用麦芽糖; 18株细菌能产生乙酰胺酶, AB06、AB07等10株菌能产生DNA酶, 所有细菌都不能产生精氨酸双水解酶(表 2)。葡萄糖氧化发酵的结果显示, AB05、AB09、AB18、AB19为产碱性细菌, 其余细菌为氧化型菌株。三株革兰氏阳性细菌中, AB08和AB17菌株的生理生化结果相同, AB09菌株除氧化酶和乙酰胺酶两种试验反应为阳性, 其余试验皆为阴性。
| 菌株编号 | Ⅰ组 | Ⅱ组 | Ⅲ组 | Ⅳ组 | 乙酰胺酶 | |||||||||
| 葡萄糖(厌氧) | 葡萄糖(需氧) | 氧化酶 | 柠檬酸盐 | 麦芽糖 | 精氨酸双水解酶 | 甘露醇 | 木糖 | 硝酸盐还原 | DNA酶 | |||||
| AB01 | - | + | + | + | - | - | + | + | - | - | + | |||
| AB02 | - | + | - | + | - | - | + | + | - | - | + | |||
| AB03 | - | + | - | + | - | - | + | + | - | - | + | |||
| AB04 | - | + | - | + | - | - | + | + | - | - | + | |||
| AB05 | - | - | - | - | - | - | - | - | + | - | + | |||
| AB06 | - | + | - | + | - | - | + | + | - | + | + | |||
| AB07 | - | + | - | + | - | - | + | + | - | + | + | |||
| AB08 | - | + | - | + | - | - | - | + | - | + | + | |||
| AB09 | - | - | + | - | - | - | - | - | - | - | + | |||
| AB10 | - | + | - | - | - | - | - | - | + | - | + | |||
| AB11 | - | + | - | + | - | - | + | + | - | + | + | |||
| AB12 | - | + | + | + | - | - | - | + | + | + | + | |||
| AB13 | - | + | + | - | - | - | - | + | - | + | - | |||
| AB14 | - | + | - | + | - | - | - | + | - | + | + | |||
| AB15 | - | + | - | + | - | - | + | + | - | + | + | |||
| AB16 | - | + | - | + | - | - | - | + | - | + | + | |||
| AB17 | - | + | - | + | - | - | - | + | - | + | - | |||
| AB18 | - | - | - | - | - | - | - | - | - | - | + | |||
| AB19 | - | - | + | - | - | - | - | - | - | - | - | |||
| AB20 | - | + | - | + | - | - | - | - | - | - | + | |||
| AB21 | - | + | - | + | - | - | + | + | + | - | + | |||
| 阳性总计 | 0 | 17 | 5 | 15 | 0 | 0 | 9 | 15 | 4 | 10 | 18 | |||
对21株纯化菌株的16S rRNA序列进行测序, 测序片段长度均超过1300bp, 将所测序列与NCBI数据库比对, 选取相似性99%以上的菌株作为参考菌株, 比对的结果发现, 数据库中相似性最高的菌株中有9株来自极区环境, 还有8株来自冰山、雪水等寒冷环境(表 3)。
| 菌株编号 | 站点 | 相似菌株(登录号) | 采集环境 | 参考文献 |
| γ-变形菌纲(γ-Proteobacteria) | ||||
| AB02 | BJ5 | Pseudomonas Fluorescens strain KB6(JF327445.1) | 俄罗斯白海 Kandalaksha 湾海水 | [25] |
| AB03 | BJ8 | Pseudomonas Fluorescens strain KB6(JF327445.1) | 俄罗斯白海 Kandalaksha 湾海水 | [25] |
| AB04 | BJ2 | Pseudomonas sp.J3.2C9(KF317744.1) | Cuatro Cienegas 池塘水 | [26] |
| AB05/AB07 | BJ5 | Pseudomonas Antarctica O-5(HG518622.1) | 南极海 Galindezme 岛苔藓 | [27] |
| AB06 | BJ7 | Pseudomonas fluorescens strain KOPRI 25717 (HQ824935.1) | 阿尔卑斯山冰川冰尘穴 | [28] |
| AB10/AB11/AB19 | BJ9 | Pseudomonas fluorescens strain KOPRI 25610 (HQ824878.1) | 阿尔卑斯山冰川冰尘穴 | [28] |
| AB12 | BJ3 | Psychrobacter glacincola strain ANT9253(AY167308.1) | 南极威德尔海(Weddell Sea)冰盖 | [29] |
| AB14 | BJ1 | Pseudomonas meridiana strain PAMC 25154 (KF528725.1) | 南极 King Sejong 研究站淡水 | [30] |
| AB15 | BJ3 | Pseudomonas fluorescens strain KOPRI 25934 (HQ825057.1) | 阿尔卑斯山冰川冰尘穴 | [28] |
| AB16 | BJ8 | Pseudomonas antarctica O-5(HG518622.1) | 南极 Galindez 岛苔藓 | [27] |
| AB20 | BJ4 | Pseudomonas fluorescens strain KOPRI 25610 (HQ824878.1) | 阿尔卑斯山冰川冰尘穴 | [28] |
| AB21 | BJ9 | Psychrobacter sp.KJF12-16(JQ800211.1) | 北极王湾(Kongsfjorden)冰川 | [31] |
| 黄杆菌纲(Flavobacteriia) | ||||
| AB01 | BJ7 | Flavobacterium sp.WB 3.3.42(AM177619.1) | 德国哈尔茨山(Harz Mountains)硬质溪水 | [32] |
| AB13 | BJ7 | Flavobacterium sp.EE-QS1(HE802578.1) | 苏黎世湖湖雪 | [33] |
| AB18 | BJ6 | Flavobacterium frigidarium strain A2i(NR_025020.1) | 南极阿德莱德岛(Adelaide Island)表层海水沉积物 | [34] |
| 放线菌纲(Actinobacteria) | ||||
| AB08 | BJ9 | Arthrobacter sulfureus strain BGSLP24(KP192002.1) | 西藏高原疏勒河流域永久冻土 | [35] |
| AB09 | BJ9 | Arthrobacter sp.Lc10-5(GU244356.1) | 南极湖泊沉积物 | [36] |
| AB17 | BJ1 | Salinibacterium sp.KJF5-12(JQ800089.1) | 北极王湾(Kongsfjorden)冰川 | [31] |
表 3结果显示, 分离出的21株浮游细菌中分别属于γ-变形菌纲(15株, 占71.43%), 黄杆菌纲(3株, 占14.29%)和放线菌纲(3株, 占14.29%)三大类群。用Cluster软件对21株菌进行聚类分析, 相似性97%以上的菌株归为一个OTU, 总共得到5个OUT (OTU01-05)。将这5个OTU与NCBI中序列比对后作系统发育树, 结果如图 4所示。结果表明, γ-变形菌纲包括假单胞菌属(Pseudomonas)13株和嗜冷杆菌属(Psychrobacter)2株(分别显示为OTU01和OTU02);放线菌纲包括盐杆菌属(Salinibacterium)1株(OTU03)和节杆菌属(Arthrobacter)2株(OTU04);黄杆菌纲中有黄杆菌属(Flavobacterium)3株(OTU05)。
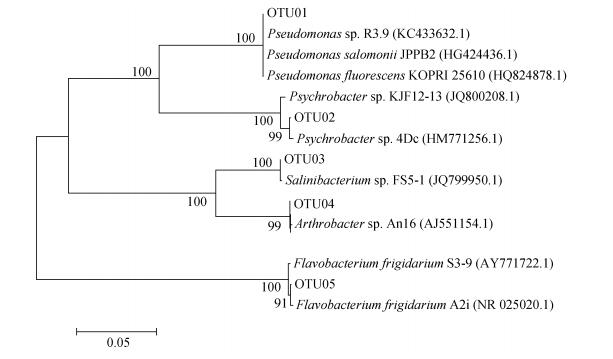 |
| 图 4 基于16S rRNA基因序列构建的细菌系统发育树 Fig. 4 Phylogenetic tree of bacteria based on 16S rRNA gene sequences |
黄河站附近站点(BJ2)所提取的总DNA, 通过高通量测序, 共得到58 303条序列, 拼接质控后用以进行后续分析的共有56 409条序列。将所有有效序列聚类后共得到1 467个OTU, 分析其α-多样性, 发现BJ2站点浮游细菌的丰富度指数(Chao1指数)为4 254.70, 多样性指数(Shannon指数)为5.75。
2.4.2 不可培养浮游细菌群落结构基于OTU分型数据, 将OTU数量小于0.5%的种类归为其他(Others), 具体浮游细菌种类及其占比例见图 5。从图 5中可看出, 以门为分类阶元(图 5-A), BJ2站点浮游细菌群落中, 变形菌门(Proteobacteria, 占49.71%)和拟杆菌门(Bacteroidetes, 占41.61%)数量最多, 除此以外, 还有少量的浮霉菌门(Planctomycetes)和疣微菌门(Verrucomicrobia)等; 以纲为分类阶元(图 5-B), 除了常见的α-、γ-变形菌纲(α-、γ-Proteobacteria)、黄杆菌纲(Flavobacteriia)、放线菌纲(Actinobacteria)、噬纤维菌纲(Cytophagia)、疣微菌纲(Verrucomicrobiae)、浮霉菌纲(Planctomycetia)等7种浮游细菌(其中又以α-变形菌纲、黄杆菌纲和噬纤维菌纲数量最多)外, 还存在少量的Sphingobacteriia纲(0.26%)和Acidimicrobiia纲(0.12%)。
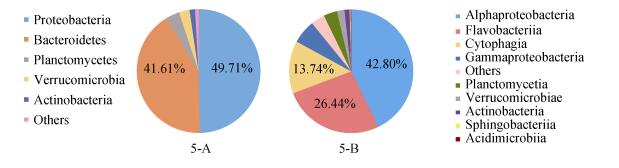 |
| 图 5 BJ2站点的细菌群落结构 Fig. 5 Bacterial community structure in BJ2 site 5-A为门水平上的分类; 5-B为纲水平上的分类 5-A, 5-B represented the classification of the phylum level and class level, respectively. |
对所占比例最大的变形菌门进行进一步分析, 结果表明:变形菌门中又以α-变形菌纲为主要优势类群, 占变形菌数量的86.10%, 其次为γ-变形菌纲, 占12.48%;包括β-变形菌在内其他3种变形菌所占比例很少, 占总体的1.42%(图 6)。
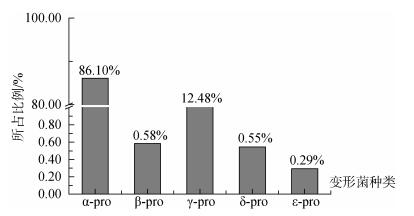 |
| 图 6 BJ2站点变形细菌门各纲所占比例图 Fig. 6 Ratio of five classes of Proteobacteria in BJ2 site Pro是Proteobacteria(变形菌)的缩写 Pro is the abbreviation of Proteobacteria |
本文对北极海水可培养浮游细菌的研究, 共得到21株细菌, 分别属于γ-变形菌纲的Pseudomonas和Psychrobacter属, 黄杆菌纲的Flavobacterium属, 放线菌纲的Arthrobacter和Salinibacterium属。李海[37]从北极王湾(Kongsfjorden)表层海水样品中分离得到的浮游细菌菌株分别属于γ-变形菌纲的Pseudoalteromonas、Vibrio、Alteromonas和Psychrobacter等属, 放线菌纲的Kocuria和Rhodococcus等属及黄杆菌纲的Maribacter、Cellulophaga、Flavobacterium、Bizionia和Olleya等属。与李海[37]的研究结果相比, 我们未能分离到γ-变形菌纲的Pseudoalteromonas、Vibrio、Alteromonas等种类, 也未能分离黄杆菌纲的Maribacter、Cellulophaga等种类, 而放线菌中分离出的两株菌是李海的研究中未提到的。
分离出来的菌株中, γ-变形菌纲所占比例最多, 达到71.4%, 为本次研究的主要优势类群。李晓亮[38]从北极沉积物中分离到的6个纲中, γ-变形菌纲的菌株数量最多, 占总分离菌株数的63.6%。两项研究皆表明γ-变形菌纲为北极地区可培养浮游细菌中的优势类群, 本实验分离出的Pseudomonas属又占γ-变形菌纲的92%。孙景[39]从北极冰川流域(北纬78°53′~79°32′, 东经11°43′~12°43′)分离得到63株一般性可培养细菌(LB培养基分离), 其中优势菌主要集中在Gammaproteobacteria纲的Pseudomonas属。作为自然界分布最广的微生物之一, 假单胞菌属不仅具有强大的环境适应能力, 而且也为人类提供了多样的遗传资源, 在环境修复、生物防治、生物转化、耐药机制等领域逐步体现出越来越重要的作用[40]。研究发现, 假单胞菌对于多环芳烃[41]、氰化物[42]、有机溶剂[43]等有机污染物的降解具有较大作用。孙景[39]发现假单胞菌在固氮、反硝化、亚硝化等氮循环作用中具有重要功能。陶玲[44]等研究天山冰川融水中细菌的生理生化特性发现, 属于Pseudomonas的菌株TRB-6和TRB-8对实验中的10种抗生素具有耐药性。此外, 从北极分离出来的假单胞菌还具有产生低温蛋白酶的特性[45], 因而具有重要的研究与开发价值, 本实验室也在进行相关研究。
本次分离得到的黄杆菌和放线菌分别占总数的14.3%, 是本次实验的第二大类群。黄杆菌和放线菌也被证明具有较好的耐药性[45]和产生低温酶的特性[46]。与滕海波[47]分离得到的8种放线菌(Streptomyces、Arthrobacter、Rhodococcus、Saccharothrix、Rathayibacter、Micrococcus、Nocardia、Kribbella)比较, 作者也分离得到了Arthrobacter; 此外, 作者还分离得到了Salinibacterium属。Prasad等[31]研究北极Kongsfjorden峡湾可培养细菌的多样性和生态功能时发现, Salinibacterium属是一种耐冷、嗜盐的菌, 能够在低温下产生脂肪酶、酪蛋白酶等一系列产物, 具有较广泛的研究前景。
3.2 不可培养浮游细菌的多样性本次实验中, 利用高通量技术研究不可培养浮游细菌多样性, 发现变形菌门为该区域的优势类群, 结论与常绪路[48]和杨晓[49]的结论相似。拟杆菌门、浮霉菌门分别为本研究的第二和第三大类群, 常绪路[48]发现除变形菌门外, 放线菌门、酸杆菌门和疣微菌门为优势类群; 杨晓[49]发现酸杆菌门的丰度高于拟杆菌门, 而浮霉菌门只占很少的一部分。
以纲为研究对象, 本次实验中, α-变形菌纲、黄杆菌纲、噬纤维菌纲和γ-变形菌纲数量较多。张灿[50]研究北极加拿大海盆浮冰区微生物组成及多样性, 发现黄杆菌纲的丰度最高, 其次为α-变形菌纲, β-变形菌纲和放线菌纲。对比其他用分子生物学技术研究的结果, 汪涛等[51]通过构建北极海水细菌16S rDNA文库, 发现细菌属于α-变形菌纲, γ-变形菌纲和拟杆菌三个大类。和振花[2]等结合16S rDNA文库法和PCR-DGGE法研究北极海水中寡营养细菌多样性, 研究发现两个站位中α-变形菌纲所占比例皆为最高, 其他还有γ-变形菌纲、鞘脂杆菌纲、放线菌纲等种类。刘莹[52]采用变性梯度聚丙烯酰胺凝胶电泳(DGGE)和构建克隆文库等技术, 研究夏季北冰洋海域浮游生物的多样性及其群落结构, 发现α-变形杆菌门、γ-变形杆菌和拟杆菌为该海域的优势类群。
无论是本次实验结果, 还是比较前人的研究结论, 不难发现, 变形菌门、α-变形菌纲是北极海域不可培养浮游细菌中的主要种类, 而其他浮游细菌的种类和数量并不完全一致, 其间的差异可能与采样地点和季节演替有关[7]。
除了比较常见的种类, 高通量测序结果还显示, 该地区存在少量的Sphingobacteriia纲和Acidimicrobiia纲。这两种浮游细菌在海洋中的含量较少, 但在世界各地海域[53], 特别是较寒冷的冰川环境中普遍存在[54]。研究表明, 这两种浮游细菌在代谢海洋不稳定溶解有机物[55]和降解包括多环芳烃在内的有机物[56]等方面意义重大, 特别是Sphingobacteriia纲, 其在低温污水中大量存在[57], 且在多药耐药(multidrug resistant)的调查中起着重要的作用[58], 有较大的研究价值。
综合可培养和不可培养的实验结果, 作者能够发现, 不论是在可培养浮游细菌中, 还是在不可培养浮游细菌中, 变形菌门所占比例皆为最高。虽然高通量测序结果表明, 该海域浮游细菌中α-变形菌纲所占比例最多(86.1%), 但在可培养实验中, 本文未能分离得到这一种类, 所分离出的变形菌皆属于γ-变形菌, 王桢[59]等人的研究也有类似的结果。冀世奇[60]的研究结论也表明, 海洋中已获取的可培养细菌中以γ-变形菌为主要类群, 不可培养细菌中以α-变形菌为主导。作者认为:这种差异反映出高通量法与可培养法的差异, 高通量测序得到的结果并不能完全体现细菌的可培养特征和在生态环境中的作用, 而可培养也不一定能分离得到与高通量测序结果一致的种类。
| [1] |
李莎.北极深海沉积物中细菌多样性的研究[D].武汉: 华中师范大学, 2010. Li Sha.The investigation on microbial diversity of Arctic deep sea sediments[D].Wuhan: Central China Normal University, 2010. |
| [2] |
和振花.北极海水中可培养寡营养细菌多样性研究[D].武汉: 华中师范大学, 2011. He Zhenhua.Diversity of culturable oligotrophic bacteria in water from the Arctic[D].Wuhan: Central China Normal University, 2011. |
| [3] |
丁慧, 王能飞, 臧家业, 等. 北极黄河站地区不同基底中真菌的分离培养及初步鉴定[J]. 海洋学报, 2014, 36(10): 124-130. Ding Hui, Wang Nengfei, Zang Jiaye, et al. Isolation and preliminary identification of fungi in different substrate from the Arctic Yellow River Station[J]. Acta Oceanologica Sinica, 2014, 36(10): 124-130. DOI:10.3969/j.issn.0253-4193.2014.10.013 |
| [4] |
辛玉华, 周宇光, 东秀珠. 低温细菌与古菌的生物多样性及其冷适应机制[J]. 生物多样性, 2013, 21(4): 468-480. Xin Yuhua, Zhou Yuguang, Dong Xiuzhu. Biodiversity and cold adaptive mechanisms of psychrophiles[J]. Biodiversity Science, 2013, 21(4): 468-480. |
| [5] |
李友训, 关翔宇, 高焱, 等. 北极地区深海微生物研究进展及对策[J]. 海洋科学, 2016, 40(12): 138-145. Li Youxun, Guang Xiangyu, Gao Yan, et al. Progress on microbial research in the deep Arctic[J]. Marine Sciences, 2016, 40(12): 138-145. DOI:10.11759/hykx20160318001 |
| [6] |
何剑锋, 崔世开, 张芳, 等. 北冰洋海域微食物环研究进展[J]. 生态学报, 2011, 31(23): 7279-7286. He Jianfeng, Cui Shikai, Zhang Fang, et al. Progress in research on the marine microbial loop in the Arctic Ocean[J]. Acta Ecologica Sinica, 2011, 31(23): 7279-7286. |
| [7] |
石涵琨, 罗玮, 高颖, 等. 夏季北极斯瓦尔巴群岛周边海域微型浮游生物群落分布特征及环境影响初探[J]. 海洋开发与管理, 2013, 30(1): 39-45. Shi Hankun, Luo Wei, Gao Ying, et al. Characteristics of micro-plankton community distribution and environmental impact in the surrounding waters of the Arctic Svalbard Islands in summer[J]. Ocean Development and Management, 2013, 30(1): 39-45. |
| [8] |
Overpeck J, Hughen K, Hardy D, et al. Arctic environmental change of the last four centuries[J]. Science, 1997, 278(5341): 1251-1256. DOI:10.1126/science.278.5341.1251 |
| [9] |
Morison J, Aagaard K, Steele M. Recent environmental changes in the Arctic:A review[J]. Arctic, 2000, 53(4): 359-371. |
| [10] |
曾胤新.北极不同海洋环境中的浮游细菌多样性比较[C]//第十五次全国环境微生物学学术研讨会, 2012. Zeng Yinxin.Comparison of bacterioplankton diversity in different marine environments in the Arctic[C]//The 15th National Symposium on Environmental Microbiology, 2012. |
| [11] |
王保军, 刘双江. 环境微生物培养新技术的研究进展[J]. 微生物学通报, 2013, 40(1): 6-17. Wang Baojun, Liu Shuangjiang. Perspectives on the cultivability of environmental microorganisms[J]. Microbiology China, 2013, 40(1): 6-17. |
| [12] |
和振花, 杨季芳, 陈吉刚, 等. 北极海水中可培养寡营养细菌多样性[J]. 海洋湖沼通报, 2011(4): 58-67. He Zhenhua, Yang Jifang, Chen Jigang, et al. Analysis of bacterial community composition by 16SrDNA clone library sampling from Arctic seawater[J]. Transactions of Oceanology and Limnology, 2011(4): 58-67. DOI:10.3969/j.issn.1003-6482.2011.04.009 |
| [13] |
Button D K, Schut F, Quang P, et al. Viability and isolation of marine bacteria by dilution culture:theory, procedures, and initial results[J]. Appl Environ Microbiol, 1993, 59(3): 881-891. |
| [14] |
Schut F, Vries E J D, Gottschal J C, et al. Isolation of Typical Marine Bacteria by Dilution Culture:Growth, Maintenance, and Characteristics of Isolates under Laboratory Conditions[J]. Appl Environ Microbiol, 1993, 59(7): 2150-2160. |
| [15] |
戴欣, 王保军, 黄燕, 等. 普通和稀释培养基研究太湖沉积物可培养细菌的多样性[J]. 微生物学报, 2005, 45(2): 161-165. Dai Xin, Wang Baojun, Huang Yan, et al. Bacterial diversity in the sediments of Taihu Lake by using traditional nutrient medium and dilution nutrient medium[J]. Acta Microbiologica Sinica, 2005, 45(2): 161-165. DOI:10.3321/j.issn:0001-6209.2005.02.001 |
| [16] |
Moran M A. The global ocean microbiome[J]. Science, 2015, 350(6266): aac8455. DOI:10.1126/science.aac8455 |
| [17] |
付新华, 刘国宁, 何健龙, 等. 山东省渤海海洋保护区典型海域表层海水微生物群落多样性分析[J]. 海洋科学, 2017, 41(1): 39-47. Fu Xinhua, Liu Guoning, He Jianlong, et al. Analysis of microbial community diversity in the Bohai Sea marine protected areas of the Shandong Province[J]. Marine Sciences, 2017, 41(1): 39-47. |
| [18] |
段曌, 肖炜, 王永霞, 等. 454测序技术在微生物生态学研究中的应用[J]. 微生物学杂志, 2011, 31(5): 76-81. Duan zhao, Xiao Wei, Wang Yongxia, et al. Application of 454 sequencing technique in microbial ecology[J]. Journal of Microbiology, 2011, 31(5): 76-81. DOI:10.3969/j.issn.1005-7021.2011.05.016 |
| [19] |
东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2002. Dong Xiuzhu, Cai Miaoying. Common bacterial manual of systematic bacteriology[M]. Beijing: Science Press, 2002. |
| [20] |
赵海萍, 李清雪, 陶建华. 海洋细菌荧光显微计数法及其应用[J]. 河北工程大学学报(自然科学版), 2007, 24(1): 57-60. Zhao Haiping, Li Qingxue, Tao Jianhua. Methods of fluorescence enumeration of marine bacteria and application[J]. Journal of Hebei University of Engineering (Natural Science Edition), 2007, 24(1): 57-60. DOI:10.3969/j.issn.1673-9469.2007.01.015 |
| [21] |
李宝明, 阮志勇, 姜瑞波. 石油降解菌的筛选、鉴定及菌群构建[J]. 中国土壤与肥料, 2007, 2007(3): 68-72. Li Baoming, Ruan Zhiyong, Jiang Ruibo. Screen and identification of oil degrading bacteria and community construction[J]. Soils and Fertilizers Sciences in China, 2007, 2007(3): 68-72. DOI:10.3969/j.issn.1673-6257.2007.03.018 |
| [22] |
Kendall M M, Boone D R. Cultivation of methanogens from shallow marine sediments at Hydrate Ridge, Oregon[J]. Archaea-an International Microbiological Journal, 2006, 2(1): 31-38. |
| [23] |
王恩辉, 张晓黎, 张莹, 等. 山东半岛北岸不同生境潮间带浮游细菌多样性研究[J]. 海洋科学, 2016, 40(6): 8-16. Wang Enhui, Zhang Xiaoli, Zhang Ying, et al. Bacterioplankton diversity in different littoral habitats along the coast of the northern Shandong peninsula[J]. Marine Sciences, 2016, 40(6): 8-16. |
| [24] |
Weisburg W G, Barns S M, Pelletier D A, et al. 16S ribosomal DNA amplification for phylogenetic study[J]. Journal of Bacteriology, 1991, 173(2): 697-703. DOI:10.1128/jb.173.2.697-703.1991 |
| [25] |
Pesciaroli C, Cupini F, Selbmann L, et al. Temperature preferences of bacteria isolated from seawater collected in Kandalaksha Bay, White Sea, Russia[J]. Polar Biology, 2012, 35(3): 435-445. DOI:10.1007/s00300-011-1091-1 |
| [26] |
Aguirrevon Wobeser E, Soberón Chávez G, Eguiarte L E, et al. Two-role model of an interaction network of free-living γ-proteobacteria from an oligotrophic environment[J]. Environmental Microbiology, 2014, 16(5): 1366-1377. DOI:10.1111/emi.2014.16.issue-5 |
| [27] |
Vasileva-Tonkova E, Romanovskaya V, Gladka G, et al. Ecophysiological properties of cultivable heterotrophic bacteria and yeasts dominating in phytocenoses of Galindez Island, maritime Antarctica[J]. World J Microbiol Biotechnol, 2014, 30(4): 1387-1398. DOI:10.1007/s11274-013-1555-2 |
| [28] |
Lee Y M, Kim S Y, Jia J, et al. Cultured bacterial diversity and human impact on alpine glacier cryoconite[J]. Journal of Microbiology, 2011, 49(3): 355-362. DOI:10.1007/s12275-011-0232-0 |
| [29] |
Brinkmeyer R, Knittel K, Jürgens J, et al. Diversity and structure of bacterial communities in Arctic versus Antarctic pack ice[J]. Applied & Environmental Microbiology, 2003, 69(11): 6610-6619. |
| [30] |
Jeong H J, Lee H, Hong S G, et al. Transposon mutagenesis of Psychrobacter cryohalolentis PAMC 21807 by tri-parental conjugation[J]. Advances in Polar Science, 2013, 24(4): 223-230. DOI:10.3724/SP.J.1085.2013.00223 |
| [31] |
Prasad S, Manasa P, Buddhi S, et al. Diversity and bioprospective potential (Cold-Active Enzymes) of cultivable marine bacteria from the Subarctic Glacial Fjord, Kongsfjorden[J]. Current Microbiology, 2014, 68(2): 233. DOI:10.1007/s00284-013-0467-6 |
| [32] |
Xiao X, Yin X, Lin J, et al. Chitinase genes in lake sediments of Ardley Island, Antarctica[J]. Appl Environ Microbiol, 2005, 71(12): 7904-7909. DOI:10.1128/AEM.71.12.7904-7909.2005 |
| [33] |
Eckert E M, Baumgartner M, Huber I M, et al. Grazing resistant freshwater bacteria profit from chitin and cell-wall-derived organic carbon[J]. Environmental microbiology, 2013, 15(7): 2019-2030. DOI:10.1111/1462-2920.12083 |
| [34] |
Humphry D R, George A, Black G W, et al. Flavobacterium Frigidarium sp.nov., an aerobic, psychrophilic, xylanolytic and laminarinolytic bacterium from Antarctica[J]. Int J Syst Evol Microbiol, 2001, 51(4): 1235-1243. DOI:10.1099/00207713-51-4-1235 |
| [35] |
Zhang B, Wu X, Zhang G, et al. Response of soil bacterial community structure to permafrost degradation in the upstream regions of the Shule River Basin, Qinghai Tibet Plateau[J]. Geomicrobiology Journal, 2017, 34(4): 300-308. DOI:10.1080/01490451.2016.1159768 |
| [36] |
Shivaji S, Kumari K, Kishore K H, et al. Vertical distribution of bacteria in a lake sediment from Antarctica by culture-independent and culture-dependent approaches[J]. Research in Microbiology, 2011, 162(2): 191-203. DOI:10.1016/j.resmic.2010.09.020 |
| [37] |
李海.北极王湾可培养细菌的多样性及两株黄杆菌的多相分类学研究[D].济南: 山东大学, 2014. Li Hai.Studies on the diversity of the culturable bacteria from Kongsfjorden and the polyphasic taxonomy of two flavobacterial strains[D].Jinan: Shandong Normal University, 2014. |
| [38] |
李晓亮, 蔡明明, 李阳, 等. 第六次北极科学考察海洋沉积物可培养细菌的多样性分析[J]. 微生物学通报, 2016, 43(5): 974-983. Li Xiaoliang, Cai Mingming, Li Yang, et al. Biodiversity analysis of cultivable bacteria isolated from marine sediments collected during 6th Arctic Expedition[J]. Microbiology China, 2016, 43(5): 974-983. |
| [39] |
孙景.北极斯瓦尔巴群岛冰川流域氮循环细菌的多样性及分类学研究[D].青岛: 青岛科技大学, 2015. Sun Jing.The diversity and taxonomic studies of nitrogen cycle bacteria isolated from Svalbard glacier of Arctic[D].Qingdao: Qingdao University of Science & Technology, 2015. |
| [40] |
杨光富, 魏云林. 假单胞菌研究现状及应用前景[J]. 生物技术通报, 2011(1): 37-39. Yang Guangfu, Wei Yunlin. The research status and application prospect of Pseudomonas[J]. Biotechnology Bulletin, 2011(1): 37-39. |
| [41] |
Weissenfels W D, Beyer M, Klein J. Degradation of phenanthrene, fluorene and fluoranthene by pure bacterial cultures[J]. Applied Microbiology & Biotechnology, 1990, 32(4): 479-484. |
| [42] |
Huertas M J, Luquealmagro V M, Martínezluque M, et al. Cyanide metabolism of pseudomonas pseudoalcaligenes CECT5344:role of siderophores[J]. Biochemical Society Transactions, 2006, 34(1): 152-155. DOI:10.1042/BST0340152 |
| [43] |
Shim H, Yang S T. Biodegradation of benzene, toluene, ethylbenzene, and o-xylene by a coculture of Pseudomonas putida and Pseudomonas fluorescens immobilized in a fibrous-bed bioreactor[J]. Journal of Biotechnology, 1999, 67(2–3): 99-112. |
| [44] |
陶玲, 顾燕玲, 郑晓吉, 等. 天山乌鲁木齐河源1号冰川融水可培养细菌生理生化特性及其系统发育[J]. 冰川冻土, 2015, 37(2): 511-521. Tao Ling, Gu Yanling, Zheng Xiaoji, et al. Cultivable bacteria isolated from the meltwater of the Glacier No.1 at headwater of the rümqi River in Tianshan Mountains:Physiological-biochemical characteristics and phylogeny[J]. Journal of Glaciology and Geocryology, 2015, 37(02): 511-521. |
| [45] |
Singh S K, Singh S K, Tripathi V R, et al. A novel psychrotrophic, solvent tolerant Pseudomonas putida SKG-1 and solvent stability of its psychro-thermoalkalistable protease[J]. Process Biochemistry, 2011, 46(7): 1430-1435. DOI:10.1016/j.procbio.2011.03.012 |
| [46] |
Kim E H, Cho K H, Lee Y M, et al. Diversity of cold- active protease-producing bacteria from arctic terrestrial and marine environments revealed by enrichment culture[J]. Journal of Microbiology, 2010, 48(4): 426-432. DOI:10.1007/s12275-010-0015-z |
| [47] |
滕海波, 唐学玺, 肖慧, 等. 北极黄河站植物根际土壤放线菌分离培养及其鉴定[J]. 极地研究, 2009, 21(1): 33-42. Teng Haibo, Tang Xuexi, Xiao Hui, et al. Isolation and identification of Actinobacteria from rhizospheric soil in the Arctic Yellow River Station[J]. Chinese Journal of Polar Research, 2009, 21(1): 33-42. |
| [48] |
常绪路.北极新奥尔松地区冰川前缘微生物多样性与功能研究[D].武汉大学, 2016. Chang Xulu.The study of microbial community diversity and function along glacier foreland in Ny-Alesund, Arctic[D].Wuhan University, 2016. |
| [49] |
杨晓.北极新奥尔松地区土壤微生物多样性初步分析[D].青岛: 青岛大学, 2016. Yang Xiao.Preliminary analysis of soil microbial diversity in Ny-Alesund, Arctic[D].Qingdao: Qingdao University, 2016. |
| [50] |
张灿.夏季北极加拿大海盆浮冰区微生物区系组成及多样性[D].合肥: 中国科学技术大学, 2016. Zhang Can.Microflora community and diversity of pack ice in the Arctic Canada Basin during summer[D].Hefei: University of Science and Technology of China, 2016. |
| [51] |
汪涛.北极海水中可培养细菌多样性研究及可培养细菌的分离鉴定[D].武汉: 华中师范大学, 2012. Wang Tao.Diversity of culturable bacteria and screening and identification of cultural bacteria in Arctic sea[D].Wuhan: Central China Normal University, 2012. |
| [52] |
刘莹.夏季北冰洋海域微微型和微型浮游生物的多样性及其群落结构研究[D].上海: 上海海洋大学, 2013. Liu Ying.Research on diversity and community structure of picoplankton and nanoplankton in the Arctic Ocean during summer seasons[D].Shanghai: Shanghai Ocean University, 2013. |
| [53] |
Vetterli A, Hyyti inen K, Ahjos M, et al. Seasonal patterns of bacterial communities in the coastal brackish sediments of the Gulf of Finland, Baltic Sea[J]. Estuarine Coastal & Shelf Science, 2015, 165: 86-96. |
| [54] |
师瑞芳.冰川前沿退缩地土壤微生物多样性及其演替的比较研究[D].兰州: 兰州交通大学, 2016. Shi Ruifang.Comparative study on the microbial diversity and succession in the forefield of different glaciers in China[D].Lanzhou: Lanzhou Jiaotong University, 2016. |
| [55] |
Liu S, Wawrik B, Liu Z. Different Bacterial Communities Involved in Peptide Decomposition between Normoxic and Hypoxic Coastal Waters[J]. Front Microbiol, 2017, 8: 353-356. |
| [56] |
Chen P, Zhang L, Guo X, et al. Diversity, Biogeography, and Biodegradation Potential of Actinobacteria in the Deep-Sea Sediments along the Southwest Indian Ridge[J]. Front Microbiol, 2016, 7: 121-124. |
| [57] |
Kruglova A, Gonzalez-Martinez A, Krkstrm M, et al. Bacterial diversity and population shifts driven by spotlight wastewater micropollutants in low-temperature highly nitrifying activated sludge[J]. Science of The Total Environment, 2017, s 605: 291-299. |
| [58] |
Narcisodarocha C, Manaia C M. Multidrug resistance phenotypes are widespread over different bacterial taxonomic groups thriving in surface water[J]. Science of The Total Environment, 2016, 9: 563-564. |
| [59] |
王桢, 李阳, 车帅, 等. 北极海洋沉积物中可培养细菌及其多样性分析[J]. 海洋学报, 2014, 36(10): 116-123. Wang Zhen, Li Yang, Che Shuai, et al. Diversity analysis of culturable bacteria isolated from marine sediments of Arctic[J]. Acta Oceanologica Sinica, 2014, 36(10): 116-123. DOI:10.3969/j.issn.0253-4193.2014.10.012 |
| [60] |
冀世奇.海洋微生物高通量培养和分选技术的建立及应用[D].青岛: 中国海洋大学, 2011. Ji Shiqi.The establishment and applications of high- throughput cultivating and sorting method for marine microorganisms[D].qingdao: Ocean University of China, 2011. |
 2018, Vol. 42
2018, Vol. 42


