中国海洋湖沼学会主办。
文章信息
- 许净, 刘慧敏, 林筱钧, 郑江, 江兴龙, 鄢庆枇, 范云庭, 汤学敏. 2021.
- XU Jing, LIU Hui-Min, LIN Xiao-Jun, ZHENG Jiang, JIANG Xing-Long, YAN Qing-Pi, FAN Yun-Ting, TANG Xue-Min. 2021.
- 鳗弧菌(Vibrio anguillarum)核酸适配体筛选中高频序列的进化多样性研究
- STUDY ON Evolutionary Diversity of SEQUENCES WITH high frequency DURING SELECTION OF APTAMERS against vibrio anguillarum
- 海洋与湖沼, 52(5): 1315-1322
- Oceanologia et Limnologia Sinica, 52(5): 1315-1322.
- http://dx.doi.org/10.11693/hyhz20210100027
文章历史
-
收稿日期:2021-01-31
收修改稿日期:2021-04-20
2. 鳗鲡现代产业技术教育部工程研究中心 厦门 361021;
3. 福建省水产生物育种与健康养殖工程研究中心 厦门 361021;
4. 福建省特种水产配合饲料重点实验室 福清 350308
2. Engineering Research Center of the Modern Technology for Eel Industry, Ministry of Education, Xiamen 361021, China;
3. Engineering Research Center of Aquaculture Breeding and Healthy of Fujian, Xiamen 361021, China;
4. Fujian Province Key Laboratory of Special Aquatic Formula Feed, Fuqing 350308, China
核酸适配体是通过指数富集配体的系统进化技术(Systematic Evolution of Ligands by Exponential Enrichment), 即SELEX筛选技术, 获得的对靶目标有较好亲和特异性的寡核苷酸分子(Ellington et al, 1990)。核酸适配体具有靶目标范围广、体积小、易合成、亲和力高、特异性强等优点, 在癌症治疗、微生物检测、食品安全等领域中呈现出广阔的应用前景(Hamula et al, 2011; 于寒松等, 2015; Yazdian-Robati et al, 2017; Liu et al, 2020)。SELEX筛选中, 需要对获得的筛选产物进行测序, 然后从测序结果中选择部分序列进行亲和特异性的验证, 亲和特异性较好的序列则被确认为核酸适配体序列。然而, 通过传统克隆测序获得的序列有成百上千条, 通过高通量测序获得的序列更是高达数万条, 因此, 如何从如此众多的序列中精准、高效的选择出相应的候选序列进行核酸适配体的验证, 是一个亟待解决的问题。
高频序列是测序结果中出现的频率大于等于2次的序列, 研究表明, 相当多的高频序列都是在筛选进化中具有竞争优势的序列, 且大概率是对靶目标有较好亲和力的核酸适配体序列(郑江等, 2014), 而高频序列的数量在总序列中的占比常常很低, 因此, 对高频序列进行分析研究, 研究其在筛选进化过程中的优势序列及其多样性变化, 将能有针对性地寻找到相应的核酸适配体序列, 从而能够降低筛选的盲目性, 提高筛选的效率。
鳗弧菌(Vibrio anguillarum)是多种淡海水养殖品种的致病菌(饶颖竹等, 2016; 王凤青等, 2018; Mohamad et al, 2019), 由其导致的疾病给水产养殖业造成巨大的经济损失, 而对鳗弧菌进行快速的分析检测则是其病害防治的前提和基础。核酸适配体因其具有亲和力高、特异性强等优点, 已广泛应用于微生物检测(Tang et al, 2013; Zheng et al, 2015; Yu et al, 2019; Sadsri et al, 2020), 因此筛选鳗弧菌的核酸适配体, 对于鳗弧菌的快速检测及其病害防治都具有积极意义。而研究其筛选过程中高频序列的进化特点及其多样性变化, 不仅能快速准确地获得鳗弧菌核酸适配体的候选序列, 对于理解掌握筛选文库的进化特点, 提高相关水产病原菌核酸适配体的筛选效率都具有积极意义。
本文选择了13轮鳗弧菌核酸适配体的筛选产物进行高通量测序, 采用生态学中常用的丰富度指数、均匀度指数、多样性指数和优势度指数对其中的高频序列进行了多样性分析, 并采用相对重要性指数(IRI)对高频序列进行了归纳分类, 最后利用在线网站对IRI较高的7种核酸适配体的二级结构进行了预测模拟, 相关研究对于核酸适配体的筛选以及鳗弧菌的检测防治都具有重要意义。
1 材料与方法 1.1 序列的来源及其种类、个体数的定义本文中的高频序列来自于以鳗弧菌为靶目标的SELEX筛选, 具体流程如下: (1) 构建并合成初始随机寡核苷酸文库: 5′-TCAGTCGCTTCGCCGTCTCC TTC(N35)GCACAAGAG GGAGACCCCAGAGGG-3′, N35为含35个随机碱基的寡核苷酸序列。(2) 孵育: 将上述寡核苷酸文库与鳗弧菌共同孵育30 min—2 h, 使寡核苷酸分子与鳗弧菌结合; (3) 分离: 利用离心、洗涤、热变性等分离技术, 将未结合的或结合较弱的寡核苷酸分子与鳗弧菌分离, 获得能与鳗弧菌较好结合的寡核苷酸分子; (4) 扩增: 将能与鳗弧菌结合的寡核苷酸分子进行不对称PCR扩增, 此为完成一轮筛选, 该PCR产物即为该轮的筛选产物, 也是下一轮筛选的次级文库; (5) 重复: 重复步骤(1)到(4), 即次级文库再次与鳗弧菌进行孵育、分离、扩增, 获得相应的筛选产物; (6) 测序: 对筛选产物进行高通量测序, 从中选择核酸适配体的候选序列进行后续的验证和鉴定。我们共进行了15轮的筛选, 选择了其中的13轮筛选产物, 即第1、2、3、4、5、6、7、9、11、12、13、14、15轮的PCR产物, 进行高通量测序, 选择测序结果中的高频序列进行分析研究。高通量测序由生工生物工程有限公司(上海)完成。
为了方便理解, 我们对本文中核酸序列的种类和个体数作如下定义或表述: 不同种类的序列是指碱基的组成、数量或排列顺序不同的序列, 通常被简称为不同序列; 同一种序列在测序结果中如果出现多次, 它的出现频率可以看作是这种序列的个体数, 而每轮测序结果中的总序列数则可看作该轮测序获得的总个体数。
1.2 高频序列在总序列中的占比统计每轮测序结果中高频序列的个体数和每种高频序列出现的频率(即该高频序列的个体数), 则该轮高频序列个体数的占比(%)=(该轮高频序列个体数/该轮测序序列总个体数)×100=(该轮所有高频序列的频率之和/该轮测序序列总个体数)×100, 分别计算出13个测序轮中高频序列的占比, 比较分析其变化规律。
1.3 高频序列的多样性分析参考王旭娜等(2018)和黄雅琴等(2020)的方法, 分别选用丰富度指数、均匀度指数、多样性指数和优势度指数, 来研究高频序列的多样性, 这些指数的原计算公式如下:
 (1)
(1) (2)
(2) (3)
(3)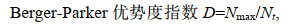 (4)
(4)原公式中, S为所有物种的种类数, N为每轮物种的个体数量的总和, Ni为第i个物种的个体数, Pi = Ni/N, Nmax为优势种的总个体数, Nt为各物种的总个体数。将上述公式应用于本文的高频序列, 则公式中的S就是某测序轮中高频序列的种类数, N为该轮中高频序列的总个体数, Ni是该轮第i个高频序列的个体数, 即第i个高频序列的出现次数或频率, Pi = Ni/N, Nmax为该轮中优势高频序列的个体数, Nt是该轮中高频序列的总出现次数。因此, 根据上述公式, 采用Primer 6.0软件可计算出每一个测序轮中的上述四种指数。
1.4 高频序列的相对重要性分析优势种的研究通常是先选择一系列的站点, 然后计算每个站点中的各个物种的相对重要性指数(Index of relative importance, IRI), 再根据该指数对相应的物种进行判定分类(王雪辉等, 2010; 刘晓霞等, 2016; 张鹏弛等, 2017; 粟丽等, 2018)。IRI的原始计算公式为: IRI = (N+W)×F×10 000 (沃佳等, 2017; 丁朋朋等, 2019), 式中, N为某一种类的个体数占总个体数的百分率; W为某一种类的重量占总重量的百分率; F为某一种类出现的站数占调查总站数的百分率。在本文高频序列的研究中, 我们将各个筛选轮或测序轮看作是原公式中的各个站点, 则总共13个测序轮就对应着13个站点, 某种高频序列出现的次数或频率就相当于该高频序列的个体数, 则公式中N就是某种高频序列在13个测序轮中出现的总次数占13轮中全部高频序列的出现总次数的百分比, F则为该高频序列出现的轮数占全部轮数(即13轮)的百分比。另外, 由于核酸序列重量与其序列长度成正比, 本文中的高频序列的长度大多在82 nt左右, 差别不大, 因此公式中的重量百分比W就近似等于其数量百分比N, 因此IRI公式就无需加和, 可只取数量百分比N, 由此相对重要性指数IRI的公式可修正为IRI = N×F×10 000。然后参考相关文献的判定方法(陈国宝等, 2007; 李显森等, 2013; 粟丽等, 2018), 依据IRI的值对不同序列进行判断分类, 具体如下: 当该序列的IRI≥500时, 该序列为优势序列; 100≤IRI < 500时, 该序列为重要序列; 10≤IRI < 100时, 该序列为常见序列, 1≤IRI < 10时, 该序列为一般序列; IRI < 1时, 该序列为少见序列。
1.5 序列的二级结构模拟利用RNA structure网站(http://rna.urmc.rochester.edu/RNAstructureWeb/)进行在线预测, 核酸类型选择为DNA, 其他参数为默认值, 最后选择最大可能性的二级结构, 即MaxExpect二级结构作为各序列的二级结构。
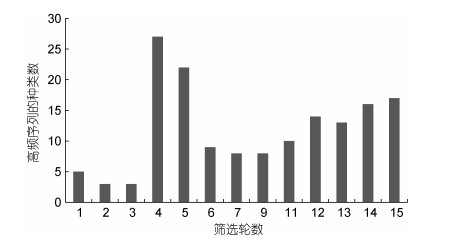
2 结果 2.1 筛选过程中高频序列的种类数及其在总序列中的占比变化高频序列种类数的变化(图 1)与其占比的变化(图 2)相类似, 都是在第4、5轮出现突然性的大幅上升, 在第4轮达到最大值, 之后快速回落后出现稳步增加, 在最后一轮达到次高点。从总趋势上看, 高频序列的种类数在前6轮出现较剧烈的波动, 之后随着筛选的进行呈稳步增加的趋势, 高频序列的占比也呈现类似变化, 不过高频序列占比的波动要更剧烈些, 后续的增加趋势也要更明显。
 |
| 图 1 筛选过程中高频序列的种类数变化 Fig. 1 Changes of high frequency sequences in the species during the selection |
 |
| 图 2 筛选过程中高频序列在总序列中的占比变化 Fig. 2 The proportion changes of high frequency sequence in the total sequence during the selection |
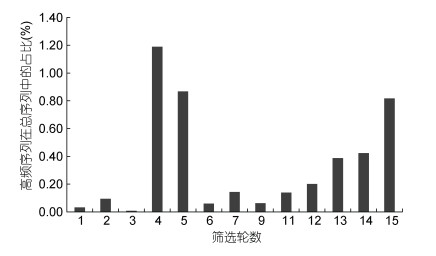
反映多样性的Shannon-Wiener指数和丰富度指数与高频序列的种类数和占比变化(如图 1和图 2)也类似, 也是在筛选的第4轮达到最高, 之后快速回落, 然后再逐步上升, 说明随着筛选进行, 高频序列也在进化, 其丰富度和多样性在经历了前6轮的剧烈波动后, 都随着筛选进化过程而逐步提高。
Pielou均匀度指数反映的是各物种个体数目分配的均匀程度, 指数越高, 说明个体间数目越均匀。图 3显示, 该指数在前6轮在一定区间内呈现较大波动, 之后则趋于稳定, 并呈现逐渐降低趋势, 说明随着筛选进行, 均匀度在经历了前6轮的剧烈波动后, 呈现越来越低的趋势, 这也进一步说明筛选进化是向着某一方向进行的, 适应这一进化方向的高频序列, 其个体数就会越来越多, 不适应这一方向的高频序列, 其个体数就会越来越少, 从而呈现出越来越大的差异和不均匀性。
 |
| 图 3 筛选过程中Margalef丰富度指数、Shannon-Wiener多样性指数、Pielou均匀度指数、Berger-Parker优势度指数的变化 Fig. 3 Changes of the Margalef richness index, Shannon-Wiener diversity index, Pielou evenness index, and Berger-Parker dominance index during the selection |
优势度指数反映的是优势种的优势程度, 优势度指数越高, 说明该轮优势种的优势越突出。图 3显示, 该指数在前6轮波动较为剧烈, 说明优势种的优势度并不稳定, 其他种的高频序列可能对优势种呈现较大的竞争压力, 之后随着筛选进行, 优势度指数也逐渐走高, 说明优势种在筛选进化中的竞争力越来越强, 其在进化竞争中的优势越来越明显, 这也反映了筛选效果越来越好。
2.3 高频序列的相对重要性分析及其分类对13轮中的79个高频序列进行了相对重要性指数(IRI)的计算, 结果如表 1。依据IRI的大小可将这些高频序列分为以下五种类型, 即优势序列(IRI≥500)、重要序列(100≤IRI < 500)、常见序列(10≤IRI < 100)、一般序列(1≤IRI < 10)和少见序列(IRI < 1)。从表中可以看出, 优势序列和重要序列分别只有1种, 约占全部高频序列的1.27%, 常见序列、一般序列和少见序列, 则分别约占全部高频序列的8.86%、18.99%和69.62%。另外, 优势序列的IRI值高达8 707.25, 是重要序列的19倍多, 这表明优势序列在筛选进化中的竞争优势是较为突出的。
| 类别 | 数量 | 高频序列 | 相对重要性指数(IRI) |
| 优势序列 | 1 | H5 | 8 707.25 |
| 重要序列 | 1 | H1 | 455.01 |
| 常见序列 | 7 | H6、H12、H17、H21、H25、H28、H32 | 12.13—31.90 |
| 一般序列 | 15 | H2、H3、H4、H7、H8、H9、H10、H11、H13、H14、H15、H26、H33、H38、H42 | 1.12—5.97 |
| 少见序列 | 55 | H16、H18、H19、H22—H24、H27、H29—H31、H35—H37、H39—H41、H43—H81 | 0.37—0.93 |
通过表 1的分类可看出, 优势序列、重要序列和常见序列应该是筛选进化中的相对优势种群, 它们对靶目标鳗弧菌的竞争力大概率要高于一般序列和少见序列, 因此, 按照IRI指数从高到低, 优先从优势序列、重要序列中挑选相应的序列进行核酸适配体的验证, 将能大大降低验证的盲目性和随机性, 提高筛选效率。后续选择了IRI最高的7个高频序列(H5、H1、H6、H12、H17、H28、H21)进行了亲和特异性验证, 发现这7个高频序列均是亲和特异性较好的核酸适配体(相关数据另文发表)。
2.4 核酸适配体序列的二级结构选择已验证确认的7个核酸适配体(H5、H1、H6、H12、H17、H28、H21)进行了二级结构的模拟(图 4), 从图中可看出, 这些序列均形成了多个大小不一的闭合环状结构, 表 2对这些核酸适配体序列中的闭合环进行总结, 可以发现, 每个序列都含有2—6个闭合环, 每个环由5—25核苷酸组成, 根据每个环中的核苷酸数量可以把这些环大致分为三种: 小型环(由5—8个核苷酸构成)、中型环(由9—13个核苷酸构成)、大型环(由17个以上的核苷酸构成)。这些环的大小和数量很可能与核酸适配体的结合位点相匹配。
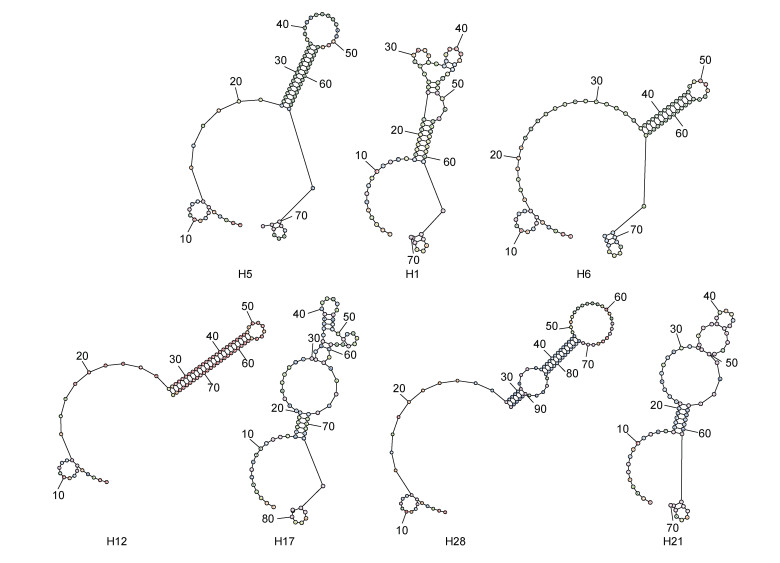 |
| 图 4 核酸适配体二级结构 Fig. 4 Secondary structures of the aptamers |
| 序列名 | IRI值 | 环的数量 | 各个环的核苷酸数量 |
| H5 | 8 707.25 | 3 | 6、10、17 |
| H1 | 455.01 | 5 | 5、7、7、7、10 |
| H6 | 31.90 | 3 | 6、9、10 |
| H12 | 27.99 | 2 | 9、10 |
| H17 | 25.19 | 6 | 6、6、7、8、10、19 |
| H28 | 15.67 | 3 | 10、13、25 |
| H21 | 15.11 | 4 | 6、8、12、19 |
研究鳗弧菌核酸适配体筛选中高频序列的进化多样性对于揭示核酸适配体的筛选进化规律、提高核酸适配体的筛选效率都具有重要意义。本文分别从高频序列的种类数、多样性、相对重要性指数(IRI)和二级结构等四个方面对高频序列的筛选进化特点进行了研究, 下面也从这四个方面对其进行分析讨论。
3.1 筛选过程中高频序列的种类数和占比变化理论上, 高频序列是在筛选竞争中占优势的序列, 其种类数量和在总序列中的占比应该随着筛选轮数的增加而逐步增加。但我们的研究却发现, 在筛选的中间阶段出现了高频序列大量爆增、剧烈波动的现象, 其种类和占比都超过了最后一轮。出现这种剧烈波动现象的原因很可能和筛选条件有关。前6轮的筛选是在较为宽松的条件下进行的(结合1—2 h, 洗涤1—2次), 所以, 虽然第4、5轮出现了高频序列的大爆发, 但大部分高频序列的亲和力可能并不高, 第7轮后为了去除亲和力较弱的序列, 筛选条件逐渐严苛, 结合时间缩短为0.5 h, 洗涤次数增加为3次, 从而导致相当多亲和力不高的高频序列被淘汰, 最后虽然高频序列的数量和占比没有达到最高, 但其中高亲和力序列的数量大大增加, 文库中高频序列的质量得到了较大的改善。
3.2 筛选过程中高频序列的进化多样性Margalef丰富度指数、Shannon-Wiener多样性指数、Pielou均匀度指数和Berger-Parker优势度指数, 经常被用于动植物的多样性研究。作为一种有效的分析和描述方法, 这些指数还被用于手足口病的流行性特征分析(Yang et al, 2020)、口腔微生物菌群的组成分析(Wolff et al, 2019)、以及土壤微生物多样性的研究(夏围围等, 2014)。本文则采用这四个指数研究了核酸适配体筛选过程中高频寡核苷酸序列的进化多样性, 结果表明, 随着筛选进行, 高频序列的丰富度、多样性和优势度都呈现逐步增加趋势, 而均匀度出现降低。这一结果很可能是因为在筛选竞争中, 在总序列中占比90%以上的、大量亲和力较弱的非高频序列逐渐被淘汰, 而亲和力较强的非高频序列则逐渐进化为高频序列, 从而导致含有大量核酸适配体的、有较高亲和力的高频序列在筛选竞争中逐渐占据优势, 其种类数量不断增加, 竞争优势也越来越强, 并呈现出强者越强的定向进化趋势。这些变化趋势与核酸适配体的筛选进化特点也是一致的。
3.3 相对重要性指数的意义核酸适配体的筛选中, 需要对筛选产物进行测序, 然后再从众多的测序序列中挑选出可能的候选序列, 进行亲和特异性的验证, 只有亲和特异性较好的序列才被认定为核酸适配体。如何挑选候选序列, 目前尚没有统一的方法。早期是采用随机的方法从测序结果中随机挑选数个序列去进行亲和特异性验证(李卫滨等, 2007), 或者利用同源性将测序的序列分为数个家族, 然后从数个家族中随机挑选序列进行验证(Liu et al, 2014), 这种随机方法要进行大量的验证实验, 工作量较大。另外, 也有学者是按照序列的频率和占比来挑选候选序列的(Zhu et al, 2021)。而我们则综合考虑了频率和出现轮数, 采用相对重要性指数(IRI)这个指标, 按照IRI从大到小来挑选相应的候选序列, 挑选出的7个高频序列后续均被确认是亲和特异性较好的核酸适配体, 说明采用IRI指数来挑选候选核酸适配体序列也是可行的。
3.4 高频序列的二级结构核酸适配体通常借助其结构中的堆积作用、形状互补、静电作用和氢键等相互作用, 通过颈环等结构, 与靶目标或嵌合或包被, 从而形成稳定的复合物(Ellington et al, 1992; Hermann et al, 2000)。有研究发现, 适配体二级结构中的不同环区可以联合参与靶标的识别与结合(孙羽菡, 2019), 还有研究表明, 两个颈环的适配体要比一个颈环的适配体有更好的稳定性和亲和特异性(Bhardwaj et al, 2019), 我们的研究也表明二级结构中环的数量和大小很可能是与其结合位点相匹配的, 进而影响其与靶目标的结合和稳定性。
4 结论利用Margalef丰富度指数、Shannon-Wiener多样性指数、Pielou均匀度指数和Berger-Parker优势度指数, 研究了鳗弧菌核酸适配体的筛选中高频序列的进化多样性, 结果发现, 在筛选进化过程中, 高频序列的丰富度、多样性、优势度和均匀度都在前6轮出现剧烈的波动, 之后随着筛选的进行, 丰富度、多样性和优势度都呈逐步增加趋势, 而均匀度则呈现逐步降低的趋势, 说明含有大量核酸适配体的高频序列在筛选竞争中逐渐占据优势, 其种类数量不断增加, 竞争优势越来越强; 利用相对重要性指数(IRI)可将高频序列分成优势序列、重要序列、常见序列、一般序列和少见序列, IRI越高的序列在筛选进化中的竞争优势也越大, 也越有可能成为高亲和力适配体; 最后, 对IRI较高的7个核酸适配体的二级结构进行了模拟分析, 发现它们的二级结构中都存在有大、中、小三种不同尺寸的环状结构, 推测这些环的大小和数量是与靶目标上的结合位点相对应的。
丁朋朋, 高春霞, 田思泉, 等. 2019. 浙江南部近海蟹类群落结构及其与环境因子的关系. 海洋渔业, 41(6): 652-662 DOI:10.3969/j.issn.1004-2490.2019.06.002 |
于寒松, 隋佳辰, 代佳宇, 等. 2015. 核酸适配体技术在食品重金属检测中的应用研究进展. 食品科学, 36(15): 228-233 DOI:10.7506/spkx1002-6630-201515042 |
王凤青, 孙玉增, 任利华, 等. 2018. 海水养殖中水产动物主要致病弧菌研究进展. 中国渔业质量与标准, 8(2): 49-56 DOI:10.3969/j.issn.2095-1833.2018.02.007 |
王旭娜, 钱宏革, 白晓拴. 2018. 包头市九峰山蝴蝶群落多样性. 生态学杂志, 37(7): 2040-2044 |
王雪辉, 杜飞雁, 邱永松, 等. 2010. 1980-2007年大亚湾鱼类物种多样性、区系特征和数量变化. 应用生态学报, 21(9): 2403-2410 |
刘晓霞, 周天舒, 唐文乔. 2016. 长江近口段沿岸4种珍稀、重要鱼类的资源动态. 长江流域资源与环境, 25(4): 552-559 DOI:10.11870/cjlyzyyhj201604003 |
孙羽菡, 2019. 单增李斯特氏菌及副溶血性弧菌适配体的结合机制与应用研究. 无锡: 江南大学硕士学位论文, 21
|
李卫滨, 兰小鹏, 杨湘越, 等. 2007. 筛选环孢霉素A适体的SELEX技术的建立(英文). 中国生物化学与分子生物学报, 23(10): 829-834 DOI:10.3969/j.issn.1007-7626.2007.10.007 |
李显森, 于振海, 孙珊, 等. 2013. 长江口及其毗邻海域鱼类群落优势种的生态位宽度与重叠. 应用生态学报, 24(8): 2353-2359 |
沃佳, 徐宾铎, 薛莹, 等. 2017. 胶州湾鱼类群落优势种组成的时空变化. 中国水产科学, 24(5): 1091-1098 |
张鹏弛, 徐勇, 李新正, 等. 2017. 南黄海冷水中心内外夏季大型底栖动物群落分析. 海洋与湖沼, 48(2): 312-326 |
陈国宝, 李永振, 陈新军. 2007. 南海主要珊瑚礁水域的鱼类物种多样性研究. 生物多样性, 15(4): 373-381 DOI:10.3321/j.issn:1005-0094.2007.04.006 |
郑江, 郝聚敏, 宋林生, 等. 2014. 哈维氏弧菌适配子的SELEX筛选及其亲和特异性研究. 生物化学与生物物理进展, 41(7): 704-711 |
饶颖竹, 梁静真, 陈蓉, 等. 2016. 海水养殖斜带石斑鱼致病性鳗弧菌的分离鉴定及药敏试验. 南方农业学报, 47(8): 1416-1422 DOI:10.3969/j:issn.2095-1191.2016.08.1416 |
夏围围, 贾仲君. 2014. 高通量测序和DGGE分析土壤微生物群落的技术评价. 微生物学报, 54(12): 1489-1499 |
黄雅琴, 王建军, 何雪宝, 等. 2020. 三沙湾互花米草(Spartina alterniflora)入侵对大型底栖动物群落结构的影响. 海洋与湖沼, 51(3): 506-519 |
粟丽, 陈作志, 张魁, 等. 2018. 南海北部深海区灯光罩网渔获物组成及渔获率的时空分布. 海洋渔业, 40(5): 537-547 DOI:10.3969/j.issn.1004-2490.2018.05.004 |
Bhardwaj J, Chaudhary N, Kim H et al, 2019. Subtyping of influenza a H1N1 virus using a label-free electrochemical biosensor based on the DNA aptamer targeting the stem region of HA protein. Analytica Chimica Acta, 1064: 94-103 DOI:10.1016/j.aca.2019.03.005 |
Ellington A D, Szostak J W, 1990. In vitro selection of RNA molecules that bind specific ligands. Nature, 346(6287): 818-822 DOI:10.1038/346818a0 |
Ellington A D, Szostak J W, 1992. Selection in vitro of single-stranded DNA molecules that fold into specific ligand-binding structures. Nature, 355(6363): 850-852 DOI:10.1038/355850a0 |
Hamula C L A, Zhang H Q, Li F et al, 2011. Selection and analytical applications of aptamers binding microbial pathogens. TrAC Trends in Analytical Chemistry, 30(10): 1587-1597 DOI:10.1016/j.trac.2011.08.006 |
Hermann T, Patel D J, 2000. Adaptive recognition by nucleic acid aptamers. Science, 287(5454): 820-825 DOI:10.1126/science.287.5454.820 |
Liu H M, Hao J M, Xu J et al, 2020. Selection and identification of common aptamers against both Vibrio harveyi and Vibrio alginolyticus. Chinese Journal of Analytical Chemistry, 48(5): 623-631 DOI:10.1016/S1872-2040(20)60018-4 |
Liu G Q, Lian Y Q, Gao C et al, 2014. In vitro selection of DNA aptamers and fluorescence-based recognition for rapid detection Listeria monocytogenes. Journal of Integrative Agriculture, 13(5): 1121-1129 DOI:10.1016/S2095-3119(14)60766-8 |
Mohamad N, Amal M N A, Yasin I S et al, 2019. Vibriosis in cultured marine fishes: a review. Aquaculture, 512: 734289 DOI:10.1016/j.aquaculture.2019.734289 |
Sadsri V, Trakulsujaritchok T, Tangwattanachuleeporn M et al, 2020. Simple colorimetric assay for Vibrio parahaemolyticus detection using aptamer-functionalized nanoparticles. ACS Omega, 5(34): 21437-21442 DOI:10.1021/acsomega.0c01795 |
Tang X M, Zheng J, Yan Q P et al, 2013. Selection of aptamers against inactive Vibrio alginolyticus and application in a qualitative detection assay. Biotechnology Letters, 35(6): 909-914 DOI:10.1007/s10529-013-1154-1 |
Wolff D, Frese C, Schoilew K et al, 2019. Amplicon-based microbiome study highlights the loss of diversity and the establishment of a set of species in patients with dentin caries. PLoS One, 14(7): e0219714 DOI:10.1371/journal.pone.0219714 |
Yang D, Liu R C, Ye L et al, 2020. Hand, foot, and mouth disease in Changsha City, China, 2009-2017: a new method to analyse the epidemiological characteristics of the disease. Infectious Diseases, 52(1): 39-44 DOI:10.1080/23744235.2019.1675902 |
Yazdian-Robati R, Arab A, Ramezani M et al, 2017. Application of aptamers in treatment and diagnosis of leukemia. International Journal of Pharmaceutics, 529(1/2): 44-54 |
Yu Q, Liu M Z, Su H F et al, 2019. Selection and characterization of ssDNA aptamers specifically recognizing pathogenic Vibrio alginolyticus. Journal of Fish Diseases, 42(6): 851-858 DOI:10.1111/jfd.12985 |
Zheng J, Tang X M, Wu R X et al, 2015. Identification and characteristics of aptamers against inactivated Vibrio alginolyticus. LWT-Food Science and Technology, 64(2): 1138-1142 DOI:10.1016/j.lwt.2015.07.021 |
Zhu C, Li L S, Fang S B et al, 2021. Selection and characterization of an ssDNA aptamer against thyroglobulin. Talanta, 223: 121690 DOI:10.1016/j.talanta.2020.121690 |
 2021, Vol. 52
2021, Vol. 52


